Учебный Сайт Николая Николаева
Назад на страницу семестра
Домены и профили.
1. Построение HMM-профиля семейства белков и проверка его работы.
Для построения профиля был выбран короткий консервативный сигма-домен NACHT-нуклеозидтрифосфатаз грибов NACHT_sigma (PF17106), имеющий неизвестную функцию. Его средняя длина - 40.2 аминокислотных остатка. Полное выравнивание содержит 180 последовательностей. Средняя идентичность доменов 42%. Среднее покрытие белка доменом - 9.31%. HMM-профиль домена имеет длину 42 и доступен по ссылке.
Двухдоменная архитектура, для которой строился профиль - SesA, NACHT_sigma - содержится в 66 белках.
Из PFAM были скачаны полные последовательности белков с данным доменом и список AC белков с искомой архитектурой. Затем с помощью Python-скрипта были отобраны последовательности этих белков, которые были выровнены с помощью веб-версии Muscle. Нередактированное выравнивание доступно по ссылке.
В ходе ревизии в JalView:
- В последовательности A0A059IYE6_TRIIM были определены координаты двух доменов (10-205). Колонки, соответствующие в этом белке амионокислотным остаткам до 10-го и с 205-го были удалены из выравнивания ('Edit' > 'Delete');
- Были удалены последовательности F7WB29_SORMK, A0A2V1BBS1_9HELO, A0A2V1BBB9_9HELO, A0A2V1BBK6_9HELO, A0A4Q4W442_9PEZI, A0A4Q4WAG9_9PEZI, A0A4Q4WAG9_9PEZI, A0A3E2H848_SCYLI, содержащие бросающиеся в глаза инсерции;
- Наконец, были удалены последовательности, более чем на 80% идентичные уже имеющимся в выравнивании ('Edit' > 'Remove redundancy')
Отредактированное выравнивание, в котором осталось 42 последовательности из 66, доступно по ссылке.
Построение профиля и поиск доменной архитектуры среди белков проводился программой HMMER 3.0 (выравнивание для этого было переведено в формат sto). Использовались команды:
hmmbuild --cpu 1 profile.hmm pr9_align.sto
hmmsearch --cpu 1 -o hmmsearch.txt -A hmmalign.fasta profile.hmm full.fasta
Построенный профиль доступен по ссылке; выдача hmmsearch также доступна по ссылке. Наличие архитектуры было предсказано во всех 180 белках, кроме A0A4Q4YIA8_9PEZI, поэтому информация о том, найдена ли в белках архитектура, в финальной таблице не приводится (см. ниже).
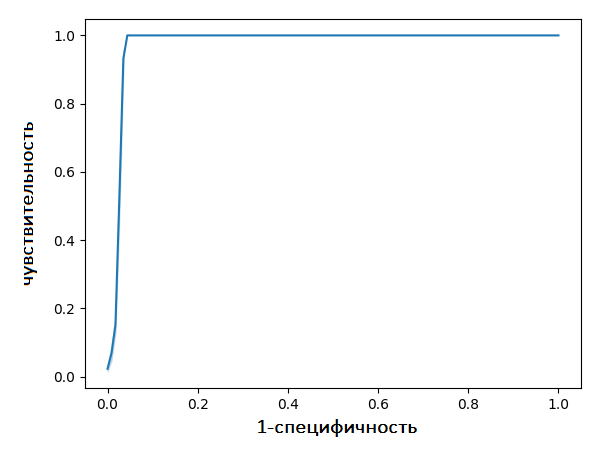
Информация о присутствии в каждом из белков архитектуры "SesA, NACHT_sigma", а также E-value и score по результатам поиска по построенному профилю доступны в финальной таблице, отсортированной по E-value и score. С помощью Python-скрипта по этим данным была построена ROC-кривая полученной модели (Рис. 1). Как по самой таблице, так и по ROC-кривой видно, что профиль довольно точно отражает реальную доменную архитектуру и hmmsearch редко ошибается в своих предсказаниях.