Выравнивание геномов, NPG
В этом практикуме нужно было построить нуклеотидный пангеном для нескольких штаммов бактерий одного вида с помощью программы NPG-explorer. Затем этот НПГ нужно было проанализировать и исследовать несколько крпных эволюционнных событий на примере выбранных штаммов.
Построение НПГ
Для выполнения практикума было выбранно 5 штаммов бактерии Bacillus atrophaeus: PENSV20, SRCM101359, BA59, GQJK17 и CNY01. И создан входной файл с данными всех этих штаммов.
Далее, по инструкции, было запущено несколько команд для построения НПГ, stdout и stderr каждой команды были выведены в отдельные лог файлы: log_prepare, Log_examine, log_make, log_post.
Для запуска команды npge MakePangenome были использованны измененные по рекомендациям из файла identity_recommended.txt параметры: MIN_IDENTITY: 0.878, WORKERS: 1. Изменения внесены в файл npge.conf.
| Файл | Содержание |
|---|---|
| pangenome.info | Сводная информация про все типы блоков |
| pangenome.bi | Информация о фрагментах генов, входящих в разные блоки. Можно использовать для поиска крупных делеций/вставок и для анализа блоков с повторами |
| global-blocks/blocks.gbi | Список глобальных блоков (синтений) |
| fragments.tsv | Координаты всех блоков |
| mut.tsv | Информация о мутациях по блокам |
| features.bs | Описание всех генов |
Стабильное ядро нуклеотидного пангенома
Информация о стабильном ядре содержится в файле pangenome/pangenome.info.
| Число S-блоков | 277 |
| Размер нуклеотидного ядра (% нулкеотидов в ядре от числа всех нуклеотидов в геноме) | 85.98% |
| % консервативных колонок в объединенном выравнивании s-блоков | 97.7% |
Описание самых крупных делеций в каждом геноме
Проследить делеции можно с помощью файла pangenome/pangenome.bi. Для удобства файл был импортирован в EXCEL. Нас интересуют "полустабильные" h-блоки, так как в них содержатся последовательности не из всех геномов. С помощью фильтра по букве h в названии блока и сортировки столбца cols по убыванию была получена информация о крупных делециях (табл. 2).
| Идентификатор блока | Длина делеции | Штаммы с делецией | Белок |
|---|---|---|---|
| h2x45547 | 45547 | BA, CNY, PENSV | Putative lipoprotein YerH |
| h2x11262 | 11137 | BA, CNY, SRCM | Amino acid adenylation domain-containig protein |
| h2x9518 | 9518 | BA, CNY, PENSV | Cell wall anchor protein |
| h3x7891 | 7891 | GQJK, PENSV | Oxidoreductase |
Описание перестановок синтений
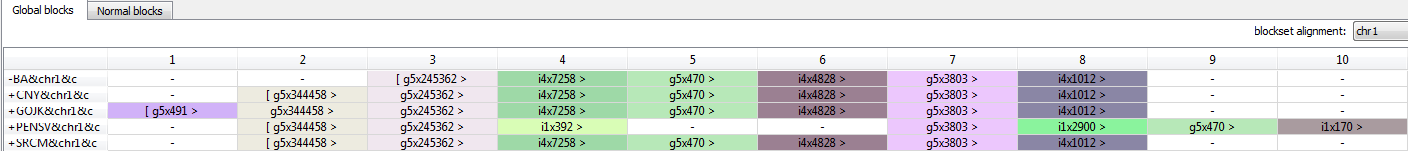


На рис. 1 показаны все g-блоки выбранных бактерий. Видно, что все блоки у всех бактерий идут в одинаковом порядке, перестановок нет. Это подтверждает родство выбранных бактерий.
Ошибки в аннотации генов
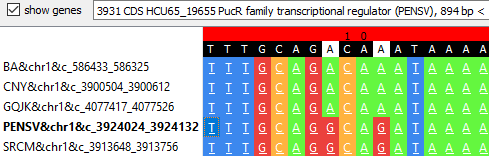
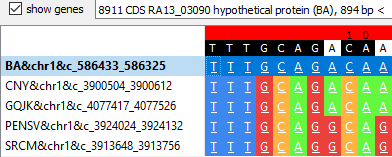
Большинство ошибок в аннотации генов связано с недорасшифровкой генома у некоторых организмов - на рис. 2-4 показан блок s5x110n2, в котором у штаммов CNY и PENSV фннотирован белок PucR family transcriptional regulator, а у остальных штаммов лишь найден белок, но его функция еще не определена (hypothetical protein/uncharacterized protein). В таких ситуациях, при высоком проценте идентичности участков, у разных штаммов должен наблюдаться один белок (скорее всего именно тот, который аннотирован).
Но иногда в одном блоке встречаются гены, аннотированные по-разному (рис. 5-8). Например, на рис. 5,6 показан блок s5x10158, в котором у всех штаммов, кроме CNY аннотирован general stress protein (на рис. 6 показана аннотация белка для GQJK), а у CNY аннотирован белок polysaccharide pyruvyl transferase family (рис.5).
На рис. 7,8 показан участок блока s5x31903. У всех штаммов, кроме SRCM в этом месте аннотирован белок GntR family transcriptional regulator (рис. 7). У SRCM aннотирован HTH-type transcriptional repressor YvoA(рис. 8).
Такие ошибки могут возникать, если гены аннотированы в разных рамках считывания или же некоторые участки генов могут сильно отличаться. Также в одном блоке могут находиться гены разных организмов, близкие по функциям (например, регуляторы транскрипции и репрессоры транскрипции (рис. 7,8)).
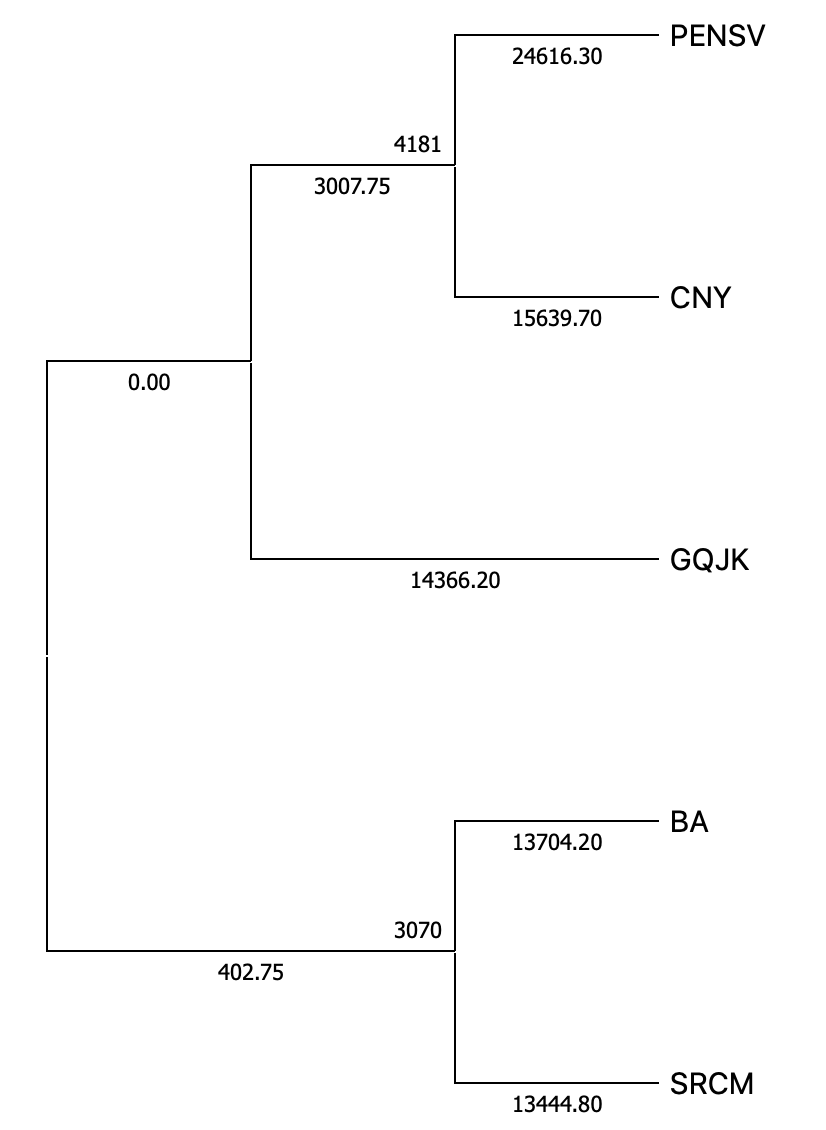
Дерево
На основании получившегося НПГ в программе MEGA было построено дерево (рис. 9), которое демонстрирует родство выбранных бактерий.