Сборка de novo
Для начала выполнения сборки de novo, был скачан один из проектов по секвенированию бактерии Buchnera aphidicola - SRR4240379.
1. Подготовка чтений программой trimmomatic
С помощью команды cp /mnt/scratch/NGS/adapters/* . в рабочую директорию были скопированы последовательности адаптеров, используемых для ILLUMINA, а командой cat *.fa >> adapters.fa эти последовательости были объединены в один файл.
Остатки адаптеров были удалены с чтений командой java -jar /usr/share/java/trimmomatic.jar SE -phred33 -threads 10 SRR4240379.fastq.gz reads_noad.fatq.gz ILLUMINACLIP:adapters.fa:2:7:7 2> removing.log. Удаленные остатки адаптеров составили 1.76%.
Затем командой java -jar /usr/share/java/trimmomatic.jar SE -phred33 -threads 10 reads_noad.fastq.gz good_reads.fastq.gz TRAILING:20 MINLEN:32 2> trim.log с концов были удалены нуклеотиды с качесвтом ниже 20, а также отобраны чтения длиной не менее 32 нуклеотидов. Из 7269852 чтений удалилось 295585. Оставшиеся очищенные чтения можно использовать в сборке de novo.
Вес исходного файла: 167Mb. Вес после удаления адаптеров: 165Mb. Вес после очистки: 156Mb.
2. Подготовка k-merов и сборка генома
Для получения k-merov длины 31 была запущена команда velveth kmers 31 -fmtAuto -short good_reads.fastq.gz. Hа основе полученных k-merов была составлена сборка генома: velvetg kmers &> velv.log.
Было получено 440 контигов разной длины. В конце log файла содержится информация об N50 получившейся сборки - 25646 нуклеотидов. Информация о получившихся контигах содержится в файле stats.txt, а последовательности контигов находятся в файле contigs.fa. В таблице 1 представлены 3 самых длинных контига. Также интересно появление контигов с аномально большим покрытием, а также контига длины 0. Эти контиги описаны в табл. 2. Помимо этого, многие из получившихся контигов имеют покрытие 1.
| ID | Длина | Покрытие |
|---|---|---|
| 6 | 49912 | 35.907237 |
| 9 | 49262 | 34.772177 |
| 5 | 33085 | 36.259030 |
| ID | Длина (в нуклеотидах) | Покрытие |
|---|---|---|
| 105 | 1 | 2694 |
| 133 | 1 | 474299 |
| 292 | 0 | Inf |
3. Анализ
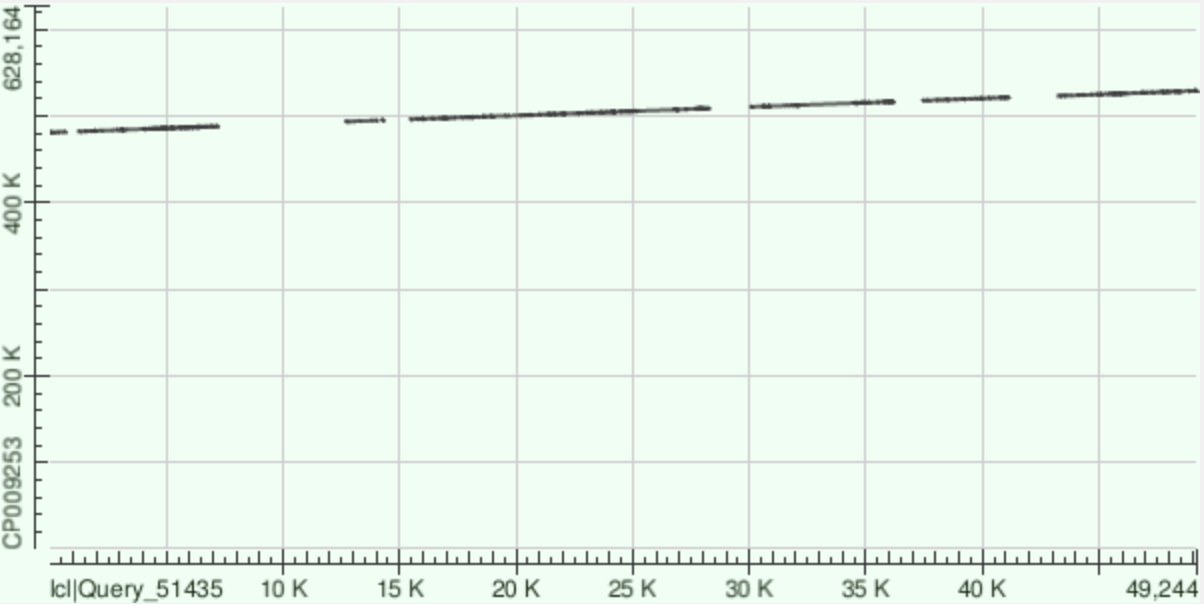
Программой MEGABLAST три самых длинных контига (записаны в отдельные файлы командой seqret) были сравнены с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253). На картинках представлены графики распределения участков контигов на хромосоме и DotPlot проведенных выравниваний. Исследование показывает, что эти контиги действительно соответствуют участкам хромосомы бактерии и получившаяся сборка соответствует действиетльности. Делеции на графиках показывают, что контиги частично покрывают хромосому (участки покрытия указаны в табл. 3). Это может возникать из-за того, что некоторые участки не отсеквенировались.
| ID | Длина | Занимаемые участки хромосомы | Ссылка на выравнивание |
|---|---|---|---|
| Контиг 6 | 49912 | 127825 - 140555; 153752 - 161738; 144368 - 151796; 161898 - 166752; 166750 - 173180 | 6alignment.txt |
| Контиг 9 | 49262 | 500370 - 508806; 510438 - 516539; 523105 - 528679; 481997 - 488106; 517766 - 521500; 496111 - 500325; 493487 - 494864; 480874 - 481545; 528794 - 529211; 495033 - 495148 | 9alignment.txt |
| Контиг 5 | 33085 | 467412 - 474667; 462496 - 467421; 474844 - 480660; 451729 - 454069 | 5alignment.txt |