A-, B- и Z- формы ДНК. Структура РНК.
Задание 1. Построение разлных структур ДНК.
Модели структур A-, B- и Z- форм ДНК были построены с помощью программы fiber пакета для анализа и моделирования труктур нуклеиновых кислот 3DNA на LINUX.
Дуплекс А-формы ДНК: gatc-a.pdb
Дуплекс B-формы ДНК: gatc-b.pdb
Дуплекс Z-формы ДНК: gatc-z.pdb
Задание 2. Сравнение сгенерированных форм ДНК с эксперементальными.
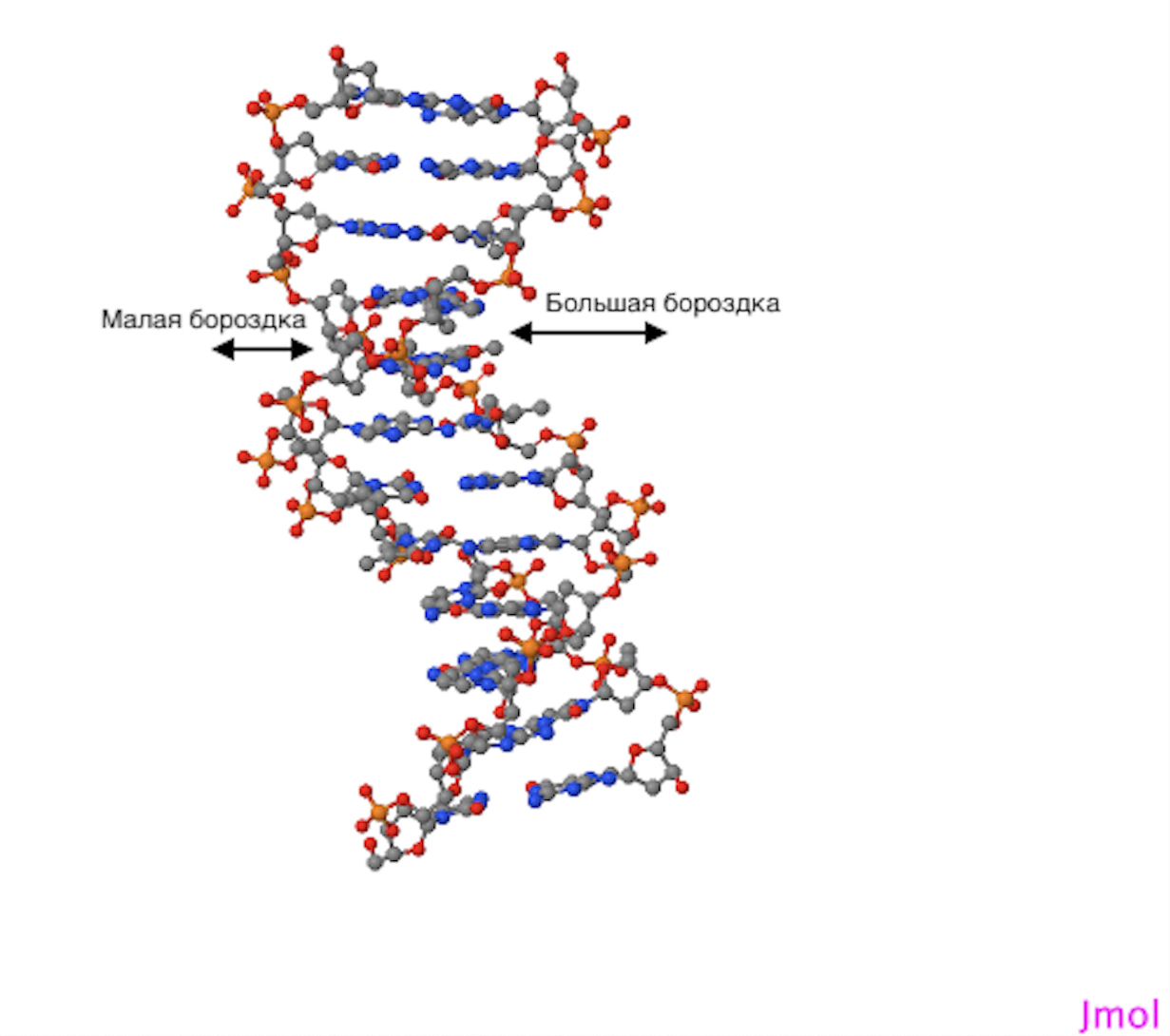
Для сравнения мной была выбрана B-форма ДНК. Визуальное различие между полученной с помощью пакета 3DNA B-ДНК и эксперементальной 1bna показано на рис. 1 и 2.
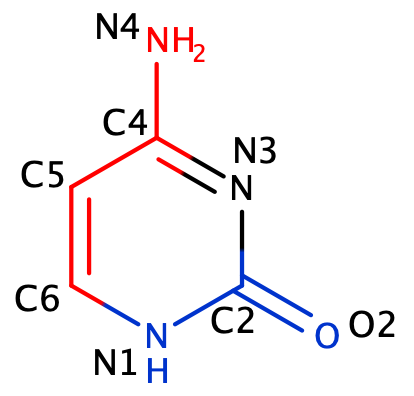
Также было мной было изучено, какие атомы цитозина в структуре 1bna в сторону какой бороздки обращены. На рис. 3 изображен цитозин, нарисованный в программе MarvinSketch.
| Атомы, обращенные в сторону большой бороздки (обозначены красным цветом): C3.C6, C3.С5, C3.C4, C3.N4 |
| Атомы, обращенные в сторону малой бороздки (обозначены синим цветом): C3.N1, C3.C2, C3.O2 |
Исследование эксперемантальных структур разных форм ДНК представлено в табл. 1. Ширина бороздок - наименьшее расстояние между двумя фосфатами соответствующих нуклеотидов.
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 пар | 10 пар | 12 пар |
| Ширина большой бороздки | 18.5 (A14:A-T31:B) | 17.21 (G13:A-G25:B) | 18.3 (C4:A-C14:B) |
| Ширина малой бороздки | 7.98 (T11:A-C24:B) | 11.69 (T15:A-A30:B) | 7.2 (G7:A-G17:B) |
Задание 3. Определение параметров нуклеиновых кислот с помощью пакета 3DNA
PDB заданной структуры РНК: 1qrt (glutaminyl-TRNA synthetase mutant). Эта РНК состоит из двух цепей: цепь А из остатков аминокилот (белковая часть) и цепь В из нуклеотидов.
Упражнение 1.
Значения торсионных углов в разных частях тРНК можно скачать по ссылке. Средние значения углов были посчитаны с помощью импорта данных в таблицы EXCEL, итоги подсчета переписаны в строку "Среднее" файла с торсионными углами. Таким образом, углы альфа, бета и эпсилон очень сильно отличаются от таковых в A- и В- формах ДНК, значения остальных углов ближе к А-форме ДНК.Упражнение 2.
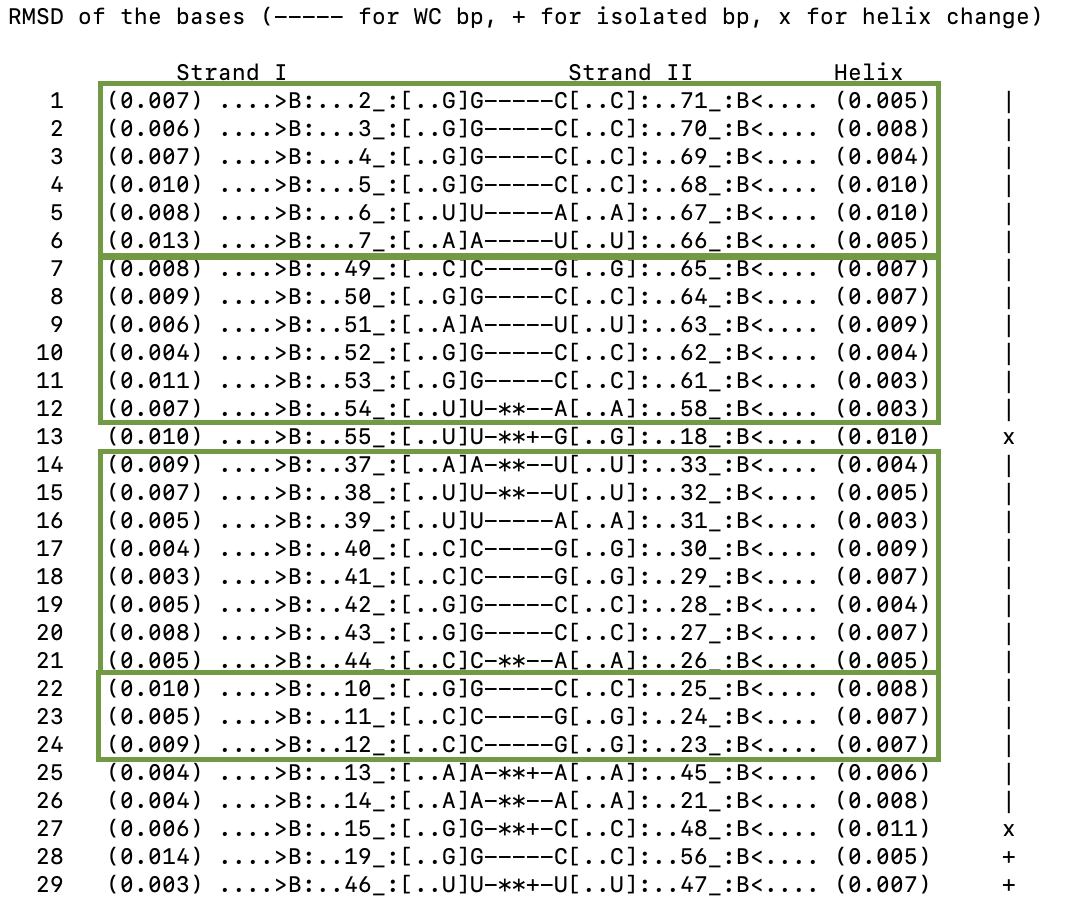
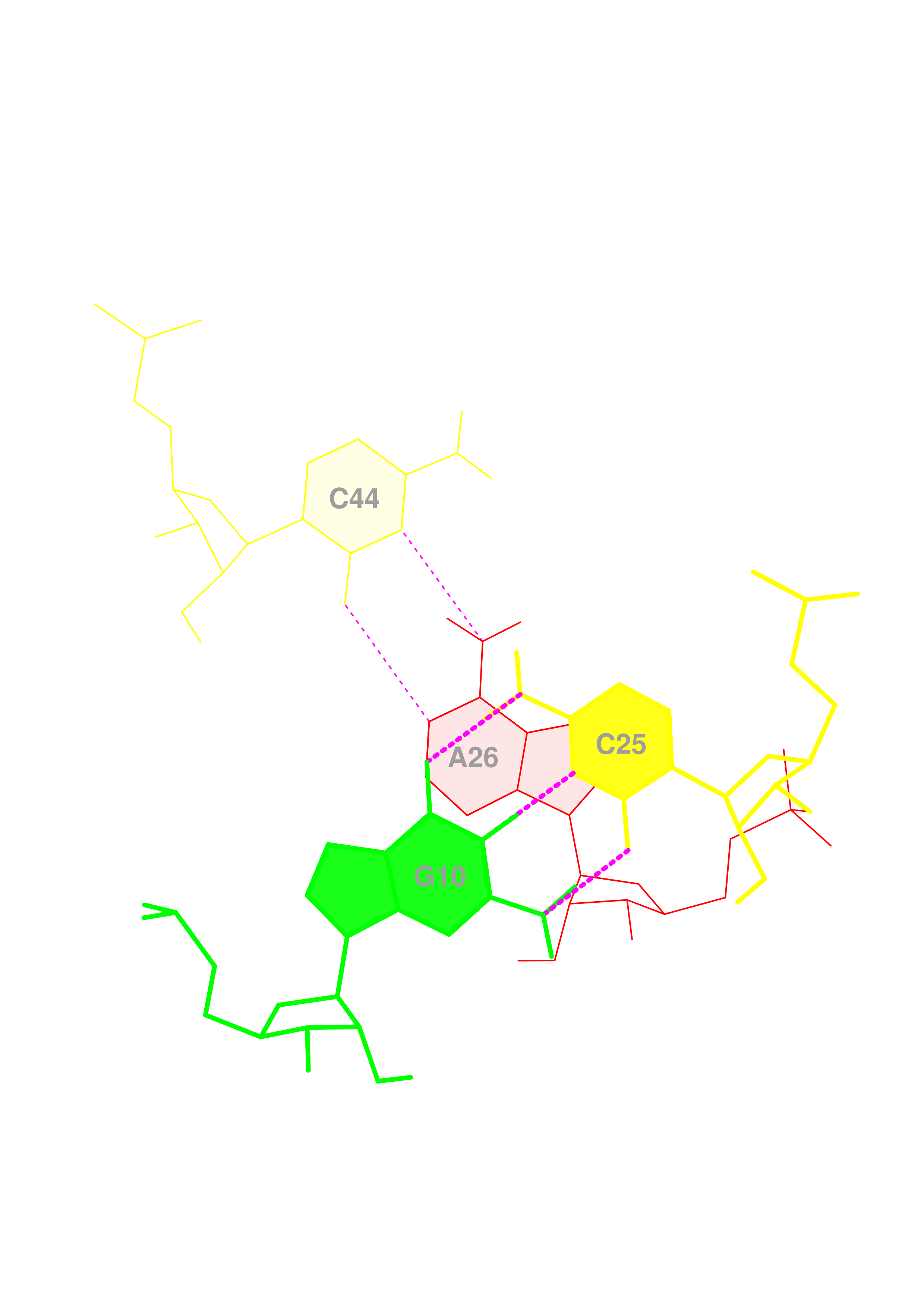
Для удобства работы, информация о водородных связях была вынесена в отдельный файл. Спаренные нуклеотиды, идущие подряд, образуют стебли (рис. 4): (2-7, 71-66), (49-54, 65-58), (37-44, 33-26), (10-12, 25-23), (37-44, 33-26). Во втором стебле наблюдается небольшое выпетливание с 61 по 58 нуклеотид.
Неканонических пар 9: U-**--A, U-**+-G, A-**--U, U-**--U, C-**--A, A-**+-A, A-**--A, G-**+-C, U-**+-U.
Некоторые комплементарные пары, не относящиеся к стеблям, образуют водорлдные связи: U55-G18, A13-A45, A14-A21,G15-C48, G19-C56, U46-U47. Эти связи стабилизируют третичную структуру тРНК. Интересно, что большинство этих связей образовано неканоническими парами нуклеотидов.