Т-РНК, комплексы ДНК-белок
Предсказание вторичной структуры тРНК
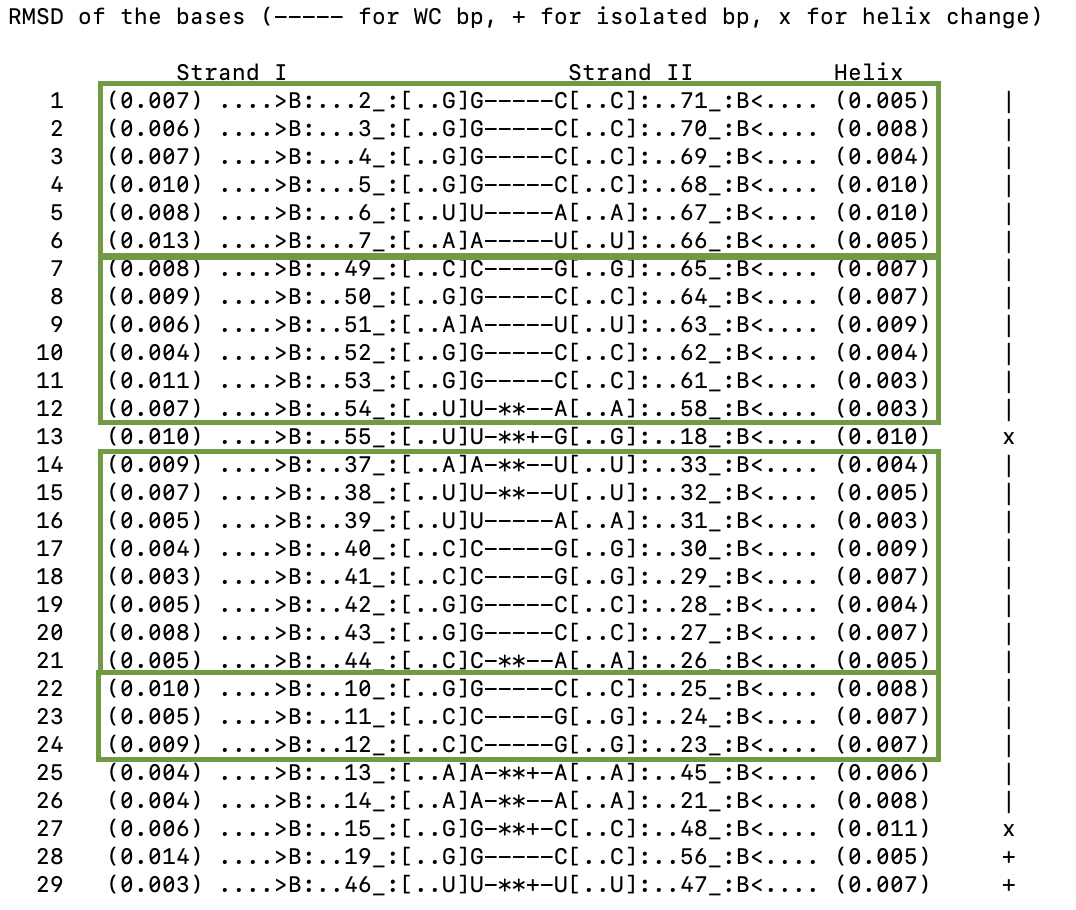
Предсказание вторичной струткуры мутанта глутаминил-тРНК-синтетазы (PDB: 1qrt) осуществлялось с помощью программ из пакета EMBOSS и Viena Rna Package. Результаты выдачи программ сравнивались с результатом выдачи find_pair из пакета 3DNA (табл. 1).
Программа einverted требует подбора параметров для более точного вывода стеблей тРНК. Наиболее точно стебли определены (рис. 1) при таких параметрах: Gap penalty: 10, Minimum score threshold : 8, Match score : 10, Mismatch score : -10. Результат выдачи программы с этими параметрами показан на рис. 1. Водородные связи на выходе этой команды строятся на основе смысловой цепи ДНК, поэтому все молекулы урацила заменены на тимин. При сравнении с выдачей find_pair (рис. 2)видно, что многие пары подобраны верно, однако эта программа не удобна за счет сложности подбора параметров.
Построение по алгоритму Зукера (программа RNAfold) дала более точный результат (рис. 3). Однако, программа не показывает неканонические пары нуклеотидов, поэтому различие между результатами все равно есть. Недостающие пары я указала на рисунке.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-2-71-3' - 5'-7-66-3' Всего 6 пар |
5'-1-71-3' - 5'-6-66-3' 6 пар, смещение на один нуклеотид назад (5 верных) |
7 пар - добавилась первая пара 5'-U1-72A-3', которой не было в find_pair, но была в выдаче einverted |
| D-стебель | 5'-10-25-3' - 5'-12-23-3' Всего 3 пары |
5'-10-25-3' - 5'-11-24-3' 2 пары |
3 пары |
| T-стебель | 5'-49-65-3' - 5'-54-58-3' Всего 6 пар, выпетливание в 1 нуклеотид |
3 пары | 5 пар |
| Антикодоновый стебель | 5'-37-33-3' 5'-44-26-3' Всего 8 пар |
6 пар, не построены неканонические пары | 5 пар |
| Общее число канонических пар нуклеотидов | 19 | 25, 16 верных | 20 |
Поиск ДНК-белковых контактов
В этой части работы будет рассмотрен комплекс ДНК-белок, участвующий в инициации репликации вируса папилломы (PDB: 1ksx). В PDB-файле представлены 2 молекулы: биомолекула 1 (C, G, A, B, E, F), биоимолекула 2 (K, O, I, J, M, N). Эти биомолекулы симметричны, поэтому для исследования была выбрана биомоелкула 1. Буквами C (5'-3') и G (3'-5') обозначены 2 цепи ДНК, A,B,E,F - цепи репликационного белка Е1.
Исследуем ДНК-белковые контакты в выбранной биоимолекуле 1, будем считать полярными атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы. Для полярного контакта расстояние между полярными атомами белка и ДНК должно быть меньше 3.5 ангстрем, для неполярного - меньше 4.5 ангстрем. Для нахождения связей мной был написан скрипт. Рузльтаты показаны в табл. 2.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 4 | 89 | 93 |
| остатками фосфорной кислоты | 74 | 96 | 170 |
| остатками азотистых оснований со стороны большой бороздки | 0 | 59 | 59 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 2 | 2 |
Большинство контактов между белками и ДНК образованы за счет сахаро-фосфатного остова, при этом в образовании полярных связей лидируют остатки фосфорной кислоты, потому что они ближе расположены к белку, чем атомы дезоксирибозы. Атомы азотистых оснований со стороны большой и малой бороздок не образуют полярных связей, а неполярных связей образуется больше со стороны большой бороздки, так как малая бороздка менее доступна.
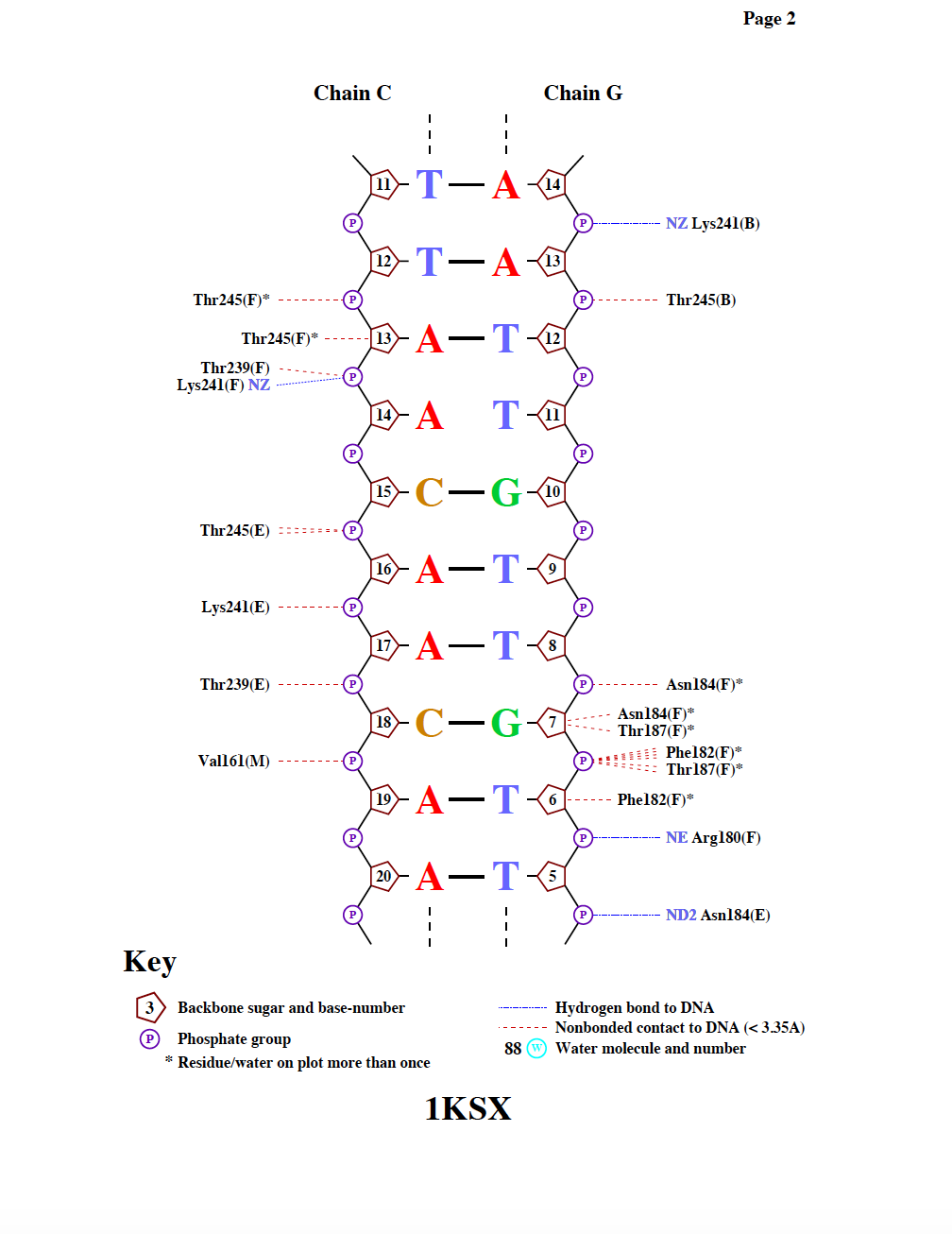
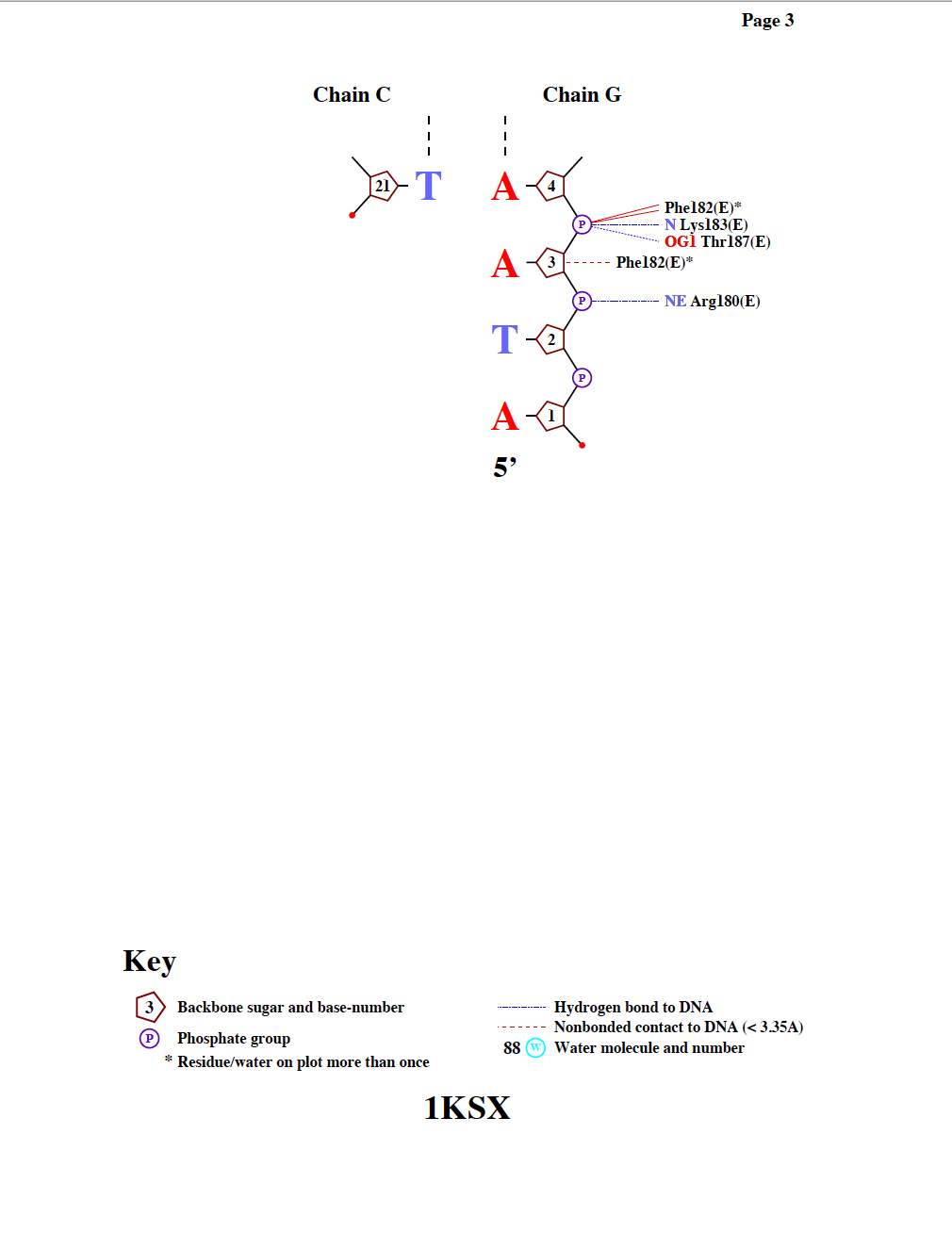
Для более подробного изучения ДНК-белковых контактов с помощью программы nucplot были построены схемы взаимодействия ДНК с белком (Рис. 4, 5, 6). На схемах отмечены контакты (несвязанные контакты длиной меньше 3.5А и водородные связи) различных аминокислотных остатков с сахаро-фосфатным остовом C и G цепей ДНК. Интересно, что рассматриваемая биомолекула 1 образует контакты не только с входящими в нее белками, но и с белками цепей белка M и I биомолекулы 2. Большинство контактов с ДНК образует Phe162(I) (6 несвязанных контактов), он также является наиболеее важным для распознавания последовательности ДНК, так как контактирует непосредственно с нуклеозидами (рис. 7). Возможно, он и отвечает за связывание двух биомолекул. Контакты с дезоксирибозой и нуклеотидами встречаются реже, чем с остатками фосфорной кислоты (табл. 1), так как нуклеозиды погружена вглубь ДНК, поэтому контакты белка с ними более специфичны. Контактирующие атомы были обнаружены в JMol при помощи команды select within (3.5, [phe]162:I) and (dna or water) (рис. 7).