Секвенирование по Сэнгеру
Задание 1. Восстановление последовательности ДНК по данным из капиллярного секвенатора
| Исходный файл с ридом прямой последовательности | 41_F.ab1 |
| Исходный файл с ридом обратной последовательности | 41_R.ab1 |
| Консенсная последовательность | Consensus.fa |
| Выравнивание прямой и обратной последовательностей | Alingment |
| Выравнивание прямой, обратной последовательности и консенсуса | Alignment_cons |
Редактирование прочтений было выполнено в программе UGENE. Сначала оба прочтения были выровнены относительно первого. С прямой последовательности удалены 14 нуклеотидов с конца (705-718) (еще несколько, помимо удаленных программой) и 24 нуклеотида в начале (1-24). В обратной последовательности было удалено 19 нуклеотидов с конца (702-720) (еще несколько, помимо удаленных программой). В прямой последовательности много ошибок хроматограммы, исправленных при сравнении с обратной.
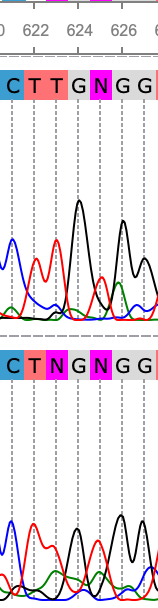
При исправлении ошибок хроматограммы были обнаружены ошибки нескольких типов (Рис. 1-3). На Рис.1 (левая N) из-за высоко уровня шума определить нуклеотид однозначно нельзя, поэтому было принято решение исправить на R (A или G); на Рис.1 (правая N) программа неправильно прочитала хроматограмму на "хвосте" обратного прочтения (высокий уровень шума), была произведена замена на C; на Рис.2 в обратном прочтении хроматограммы отсутствует пик, который есть на прямом прочтении, поэтому гэп был заменен на G, также неправильно определен 43 нуклеотид прямого прочтения: там должен быть T; на Рис.3 нулеотиды (625) обоих прочтений были неправильно определены программой, но по пикам видно, что там должен стоять T, также левая N неопределена, так как пик находится на нетипичном расстоянии от 622.
Приблизительное отношение сигнала к шуму - 10:1. Сигнал по всему прочтению очень неравномерный: в некоторых местах они составляет меньше половины высоты нормального пика. Шум намного менее равномерный: в некоторых местах соотношение максимальной и минимальной высоты шума превышает 20:1. Самый часто встречающийся нуклеотид - тимин.
Задание 2. Нечитаемый фрагмент хроматограммы
На рис. 4 показан нечиатемый фрагент хроматограммы из файла в папке bad. В прямом прочтении хроматограммы детектируется один большой сигнал гуанина, тогда как в обратном прочтении почти все сигналы (за исклбючением нескольких нетипичных пиков, рис. 5) хорошо различимы. Ошибка на рис. 4, вероятнее всего, возникла из-за проблем во время прохождения ddNTP через капилярный форез: возможно, произошел сбой в работе лазера или детектора, либо концентрация ddGTP значительно превышала концентрацию других помеченных дидезоксинуклеотидов, так что ddGTP нетипично присоединилась к оборванным концам цепи ДНК.