Трансмембранные белки
База данных OPM
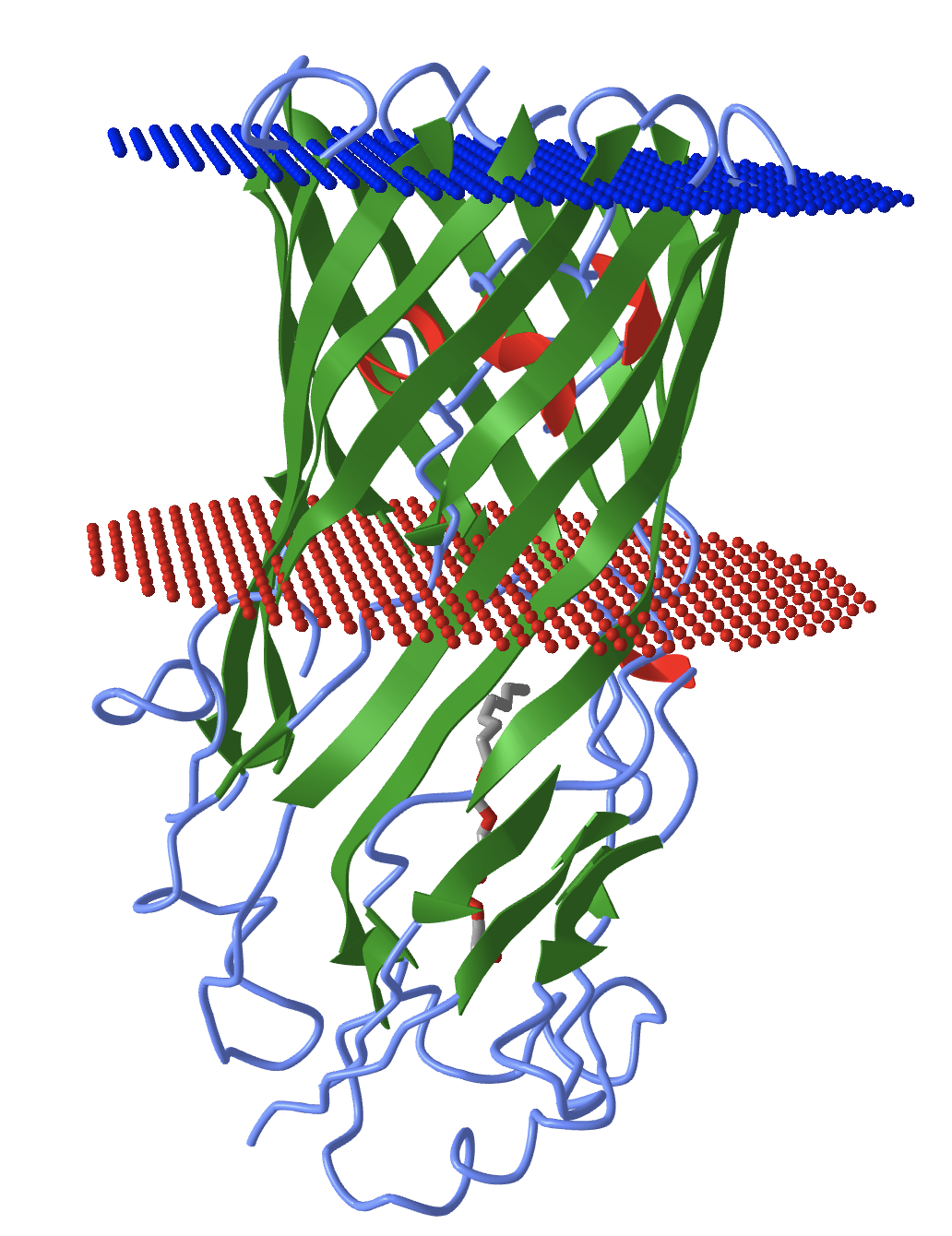
Выбранный белок с бета-листами в трансмембранной части - Toluene transporter TbuX (Транспортер толуола) бактерии Ralstonia pickettii.
| PDB | Q9RBW8_RALPI |
| Толщина трансмембранной части | 25.4 Å |
| Координаты трансмембранных участков | A - Tilt: 2 - TM segments: 1(45-53), 2(81-89), 3(95-103), 4(136-144), 5(150-159), 6(219-227), 7(233-241), 8(279-287), 9(293-301), 10(336-345), 11(350-359), 12(378-386), 13(393-402) |
| Среднее количество остатков в одном бета-тяже | 15 |
| Локализация | Внешнаяя мембрана грам-отрицательной бактерии |
DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
Альфа-спиральный белок: TatCo - бактериальный трансмембранный белок, отвечающий за перенос через мембрану больших белков с двойным аргининовым мотивом в сигнальном пептиде. Рассмотренный в данной работе белок TATCO_HALVD принадлежит архее Haloferax volcanii. Это архея - экстремофил, способная выживать в условиях повышенной солености.
Бета-листовой белок: Транспортер толуола, изученнный на предыдущем этапе работы.
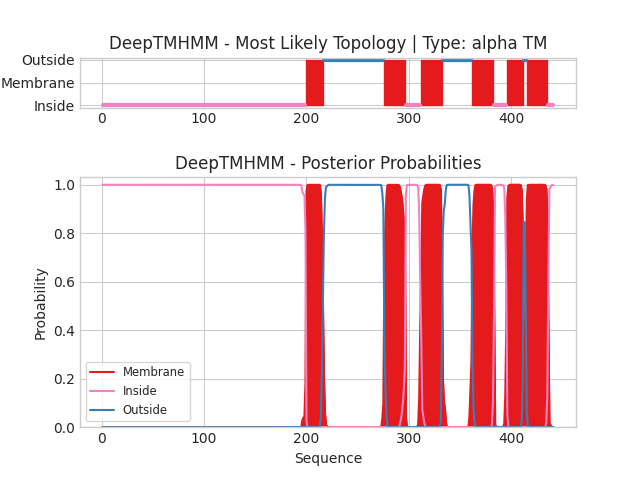
Аминокислотные последовательности белков были загружены в сервис DeepTMHMM для предсказания их вторичной структуры. Выдача для TATCO_HALVD: TATCO.gff3, TATCO.3line; для Q9RBW8_RALPI: RALPI.gff3, RALPI.3line.
Описание графиков: по оси Х расположениы номера аминокислотных остатков последовательности белка, по оси Y - вероятность принадлежности АК остатков к определенным частям белка.
Красный - часть белка внутри мембраны.
Розовый - часть белка со стороны цитоплазмы
Синий - часть белка с внешней стороны мембраны
Оранжевый - сигнальный пептид
Зеленый - часть белка со стороны периплазмы
Для выданного белка TATCO_HALVD выделилось 6 трансмембранных участков, что соответствует информации из Uniprot. У транспортера толуола TbuX выделилось 14 трансмембранных участков, тогда как по даным OPM их 13.
PPM: Предсказание положения выданного белка в мембране
С помощью сервиса PPM было определено положение альфа-спирального белка TATCO_HALVD в мембране.
Параметры:
Количество мембран: 1
Тип мембраны: Archaebacteria cell membrane (Haloferax volcanii - архея)
Allow curvature: no
Топология N-конца: in (в выдаче DeepTMHMM левый конец расположен внутри клетки, со стороны цитоплазмы)
Входной PDB файл. Выдача: TATCOout.pdb
| Толщина трансмембранной части | 26.8 ± 0.9 Å |
| Координаты трансмембранных участков | 1( 115- 123), 2( 200- 222), 3( 270- 290), 4( 312- 335), 5( 357- 376), 6( 395- 416), 7( 417- 432) |
| Среднее количество остатков в одной альфа-спирали | 23 |
| Локализация | Внешнаяя мембрана клетки |
Сравнение алгоритмов предсказания трансмембранных спиралей
Toluene transporter TbuX
OPM: 1(45-53), 2(81-89), 3(95-103), 4(136-144), 5(150-159), 6(219-227), 7(233-241), 8(279-287), 9(293-301), 10(336-345), 11(350-359), 12(378-386), 13(393-402)
DeepTMHMM: 1(68-77), 2(102-111), 3(117-129), 4(158-167), 5(173-183), 6(242-250), 7(257-266), 8(301-310), 9(317-326), 10(358-367), 11(374-383), 12(399-409), 13(416-425), 14(447-456). Предсказан один лишний лист.
Дело в том, что DeepTMHMM предсказывает структуру белка по его гену и не учитывает модификации белка при, например, встраивании в мембрану клетки. В данном случае (предположительно), сигнальный пептид, который использовался для транспортировки белка в мембрану, был отрезан после встраивания. При отрезании сигнального пептида изменилась нумерация аминокислот в белке.
TatCo
PPM: 1( 115- 123), 2( 200- 222), 3( 270- 290), 4( 312- 335), 5( 357- 376), 6( 395- 416), 7( 417- 432)
DeepTMHMM: 1(200-216), 2(276-296), 3(312-332), 4(362-382), 5(396-411), 6(415-435)
UniProt: 1(200-220), 2(276-296), 3(317-337), 4(357-377), 5(395-415), 6(416-436)
Все трансмембранные альфа-спирали, обнаруженные DeepTMHMM совпадают с предсказанием UniProt.DeepTMHMM был предсказан внутриклеточный N-конец, тогда как в UniProt и PPM он проходит через мембрану. Важно, что этот участок обладает низкой достоверностью (<50), что может говорить о взаимосвязи результатов предсказания PPM и достоверности UniProt, но это не так важно, так как в целом качество предсказания очень высокое.
База данных TCDB
TatCo был найдет в базе данных TCDB по идентификатору D4GZD0. TCDB код: 2.A.64.4.1:2 - класс электрохимические потенциал-зависимые транспортеры
A - подкласс унипортеры, антипортеры, симпортеры
64 - семейство распознающих двойной аргинин
4.1 - код белка