Паралоги, визуализация
Составление списка гомологичных белков, включающих паралоги
Для нахождения белков, гомологичных CLPX_ECOLI, протеомы выбранных бактерий были скачаны в отдельный файл. Из файла была создана база данных и проведен поиск гомологов с помощью blastp (выдача):
$ makeblastdb -in proteomes.fasta -dbtype prot
$ blastp -task blastp -query CLPX_ECOLI.fasta -db proteomes.fasta -evalue 0.001 -out result.txt
Реконструкция и визуализация
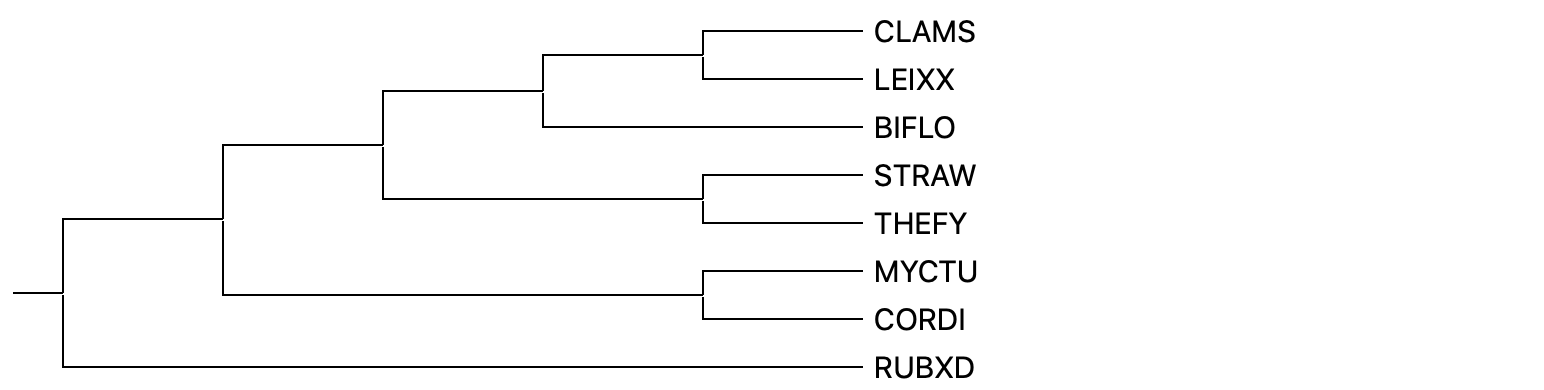
По названиям белков в UniProt были скачаны их последовательности, а затем выровнены с помощью Muscle. Выравнивание было импортировано в MEGA. Дерево (Рис. 2) было построенно методом наибольшего правдоподобия (Maximum Likelihood), Newick.
Большинство белков неаннотированны (у неготорых белков, функция лишь предполагается), поэтому невозможно точно определить, являются ли белки ортологами.
Пары ортологов: CLPX_LEIXX & CLPX_CLAMS; FTSH_RUBXD & FTSH_MYCTU; CLPX_STRAW & Q47MU4_THEFY.
Пары паралогов: CLPX_BIFLO & RUVB_BIFLO; Q1AY82_RUBXD & Q1AVT0_RUBXD; Q47MZ2_THEFY & Q47KU4_THEFY.

CLPX - ATP-dependent Clp protease ATP-binding subunit. В эту ортологичную группу попали все выбранные бактерии. Реконструированная филогения белков частично соответствует филогении бактерий: сохранены ветви CLAMS & LEIXX, STRAW & THEFY; но при этом MYCTU & CORDI вынесены в разные ветви, а также BIFLO вынесена далеко от CLAMS & LEIXX.
FTSH - ATP-dependent zinc metalloprotease. Сюда попали все бактерии, кроме CORDI. Филогения бактерий снова не совпадает с правильной: RUBXD & MYCTU объединены в одну ветвь, а THEFY & STRAW снова вынесены в разные ветви.