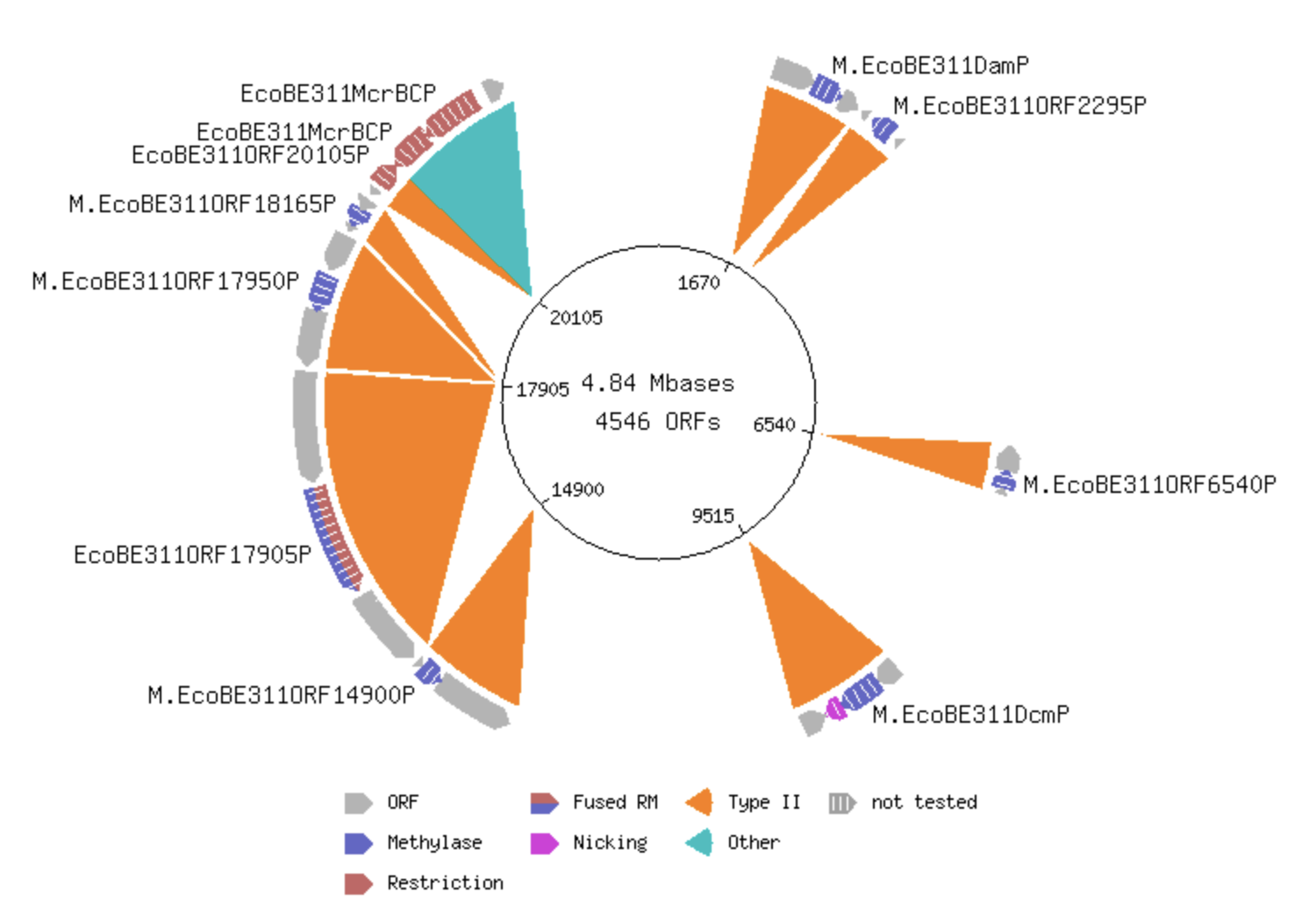
Недопредставленность сайта рестрикции в геноме Escherichia coli B311
В клектах бактерий есть механизм, направленный на борьбу с чужеродной ДНК - система рестрикции-модификации. Ферменты эндонкулеазы, закодированные в геноме бактерий, специфично узнают сайты рестрикции в последовательности нуклеотидов и делают двуцепочченый разрыв. Метилирование собственной ДНК предотвращает действие эндонуклеаз на собственные сайты рестрикции. В системах рестрикции-модификации II типа метилтрасферазная и нуклеазная активность обеспечивается независимыми белками, закодирвоанными в геноме.
Для исследования систем рестрикции-модификации II типа, в рамках задания, была выбрана бактерия Escherichia coli BE311, в геноме которой закодировано 8 эндонуклеаз (рис. 1, табл. 1).
| Рестриктаза II типа | Сайт рестрикции |
|---|---|
| M.EcoBE311DamP M.EcoBE311ORF18165P M.EcoBE311ORF14900P | GATC |
| M.EcoBE311ORF2295P | ATGCAT |
| M.EcoBE311DcmP V.EcoBE311DcmP | CCWGG |
| EcoBE311ORF17905P | CANCATC |
| M.EcoBE311ORF17950P | GGTACC |
Поиск представленности сайтов рестрикции
✓ Поиск представленности в геноме бактерии сайтов рестрикции, узнающихся закодированными в ее же геноме рестриктазами: cbcalc -K -s inrm.tsv -o myrm_in_genome.tsv BE311.fasta. На выходе был получен файл: myrm_in_genome.tsv с информации о представленности в геноме 5 сайтов рестрикции из табл. 1. .
✓Поиск всех сайтов рестрикции систем рестрикции-модификации II типа. Сначала из таблицы со всеми недопредставленными сайтами были выбраны известные сайты длины 3 и более нуклеотидов: RMsites.txt. Затем произведен поиск по геному: cbcalc -K -s RMsites.txt -o allrm_in_genome.tsv BE311.fasta Полученные результаты сохранены в файл.
Из файла со всеми встречающимися в геноме сайтами рестрикции были выбраны недопредставленные - сайты с O/E ratio менее 0,8 (по рекомендации преподавателя): файл. Затем с помощью скрипта на python из общего списка рестриктаз были выбраны те эксперементально проверенные рестрикатазы, которые могут узнавать найденные недопредставленные сайты: result.txt.
PSI-BLAST
Для выполнения задания выбран Ribosome hibernation promotion factor (O05886), выделенный из бактерии Mycobacterium tuberculosis strain ATCC25618. Этот белок димеризует 70S рибосомы с образованием 100S рибосом во время фазы роста бактерии[1].
Для составления семества гомологов выбранного белка был запущен PSI-BLAST по банку SwissProt с порогом E-value 0.005. Результаты интераций PSI-BLAST описаны в табл. 2.
| Номер итерации | Число находок выше порога (0.005) | ID худшей находки выше порога | E-value этой находки | ID лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 20 | P17161.1 | 0.003 | P171160.1 | 0.005 |
| 2 | 27 | P71346.3 | 1e-11 | - | - |
| 3 | 31 | P06727.4 | 0.003 | P33621.1 | 0.007 |
| 4 | 38 | P02651.2 | 2e-04 | O46409.1 | 0.007 |
| 5 | 46 | P32918.2 | 0.004 | O42296.1 | 0.008 |
| 6 | 136 | A0A6P6DKR7.1 | 0.005 | P0DKW8.2 | 0.005 |
Семейство гомологов выделяется после 2 итерации PSI-BLAST: на второй итерации нет находок ниже порога E-value, а на 3 возникают новые белки - аполипопротеины (медиаторы липидного обмена[2]). Аполипопротеины продолжают добавляться и в последующих итерациях (прием только они): скорее всего, этими итерациями мы захватываем близкое в искомому смейство гомологов. Если же после третьей итерации не включать в построение PSSM-матрицы нововошедшие аполипопротеины, ситуация стабилизируется и новые белки в последующих итерациях не находятся.
Можно считать, что семейство Ribosome hibernation promotion factor построенно хорошо: на второй идерации нет находок ниже порога, а в последующих итерациях порог растет. При исключении "лишних" белков в третьей итерации, поиски сходятся.
Литература:
1) Ueta M, Ohniwa RL, Yoshida H, Maki Y, Wada C, Wada A. Role of HPF (hibernation promoting factor) in translational activity in Escherichia coli. J Biochem. 2008 Mar;143(3):425-33. doi: 10.1093/jb/mvm243. Epub 2008 Jan 2. PMID: 18174192.
2) Deng X, Morris J, Dressmen J, Tubb MR, Tso P, Jerome WG, Davidson WS, Thompson TB. The structure of dimeric apolipoprotein A-IV and its mechanism of self-association. Structure. 2012 May 9;20(5):767-79. doi: 10.1016/j.str.2012.02.020. PMID: 22579246; PMCID: PMC3354570.