Построение HMM-профиля семейства белков Selenoprotein P
Селенопротеины - семейство белков, содержащих аминокислоту селеноцистеин. Селенопротеин Р - белок плазмы крови, который, предположительно, защищает клетки организма от токсичных веществ[1].
Селенопротеин Р состоит из двух доменов (рис.1): N-концевой с одним селеноцистеином (вероятно, учатсвующий в окислении) и C-концевой домен, включающий в себя 9 селеноцистеинов (скорее всего, отвечает за хранение и транспорт селеноцисетина)2.
Построение HMM-профиля
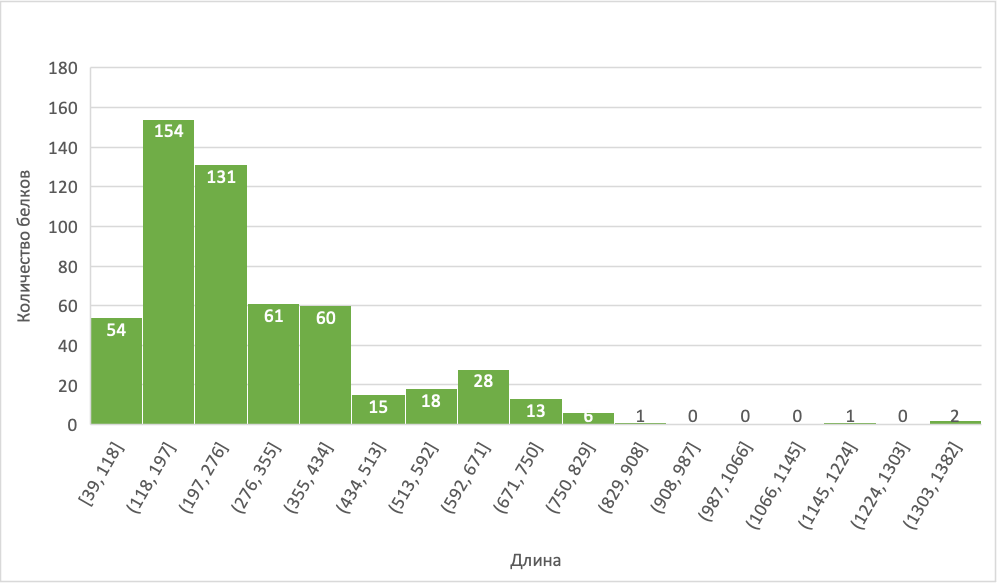
Для построения профиля был выбран N-концевой домен селенопротеина P - SelP_N (PF04592). Выбранная доменная архитектура содержит полные N-концевой и С-концевой (PF04593) домены селенопротеина Р (рис. 1). В нее входят белки, по длине относящиеся к 5 столбцу (рис. 3) - это не самая длинная доменная архитектура SelP_N.
| ID | Количество последовательностей | Средняя длина домена | Среднее сходство | Число доменных архитектур | Длина HMM-профиля |
|---|---|---|---|---|---|
| PF04592 | 8 (seed), 544 (full) | 166.2 aa | 32 % | 12 | 233 |
Для построения выравнивания были скачаны все последовательности (full), из которого были выбраны последовательности, содержащие нужную доменную архитектуру (domain). По последовательнсотям с доменной архитектурой было построено выравнивание алгоритмом Muscle, из которого был удален участок до первого домена и участок после второго домена. Я решила оставить короткие консервативные участки до первого и после второго домена, так как они, судя по выравниванию, тоже могут являться частью домена. Также были удалены высокосходные (Redundancy 90) и плохо выровненные последовательности. В результате осталось 27 последовательностей. Полученное выравнивание использовалось для построения HMM-профиля.
Построение HMM-профиля
hmm2build hmm alignment.fasta Построен HMM-профиль для поиска архитектуры
hmm2calibrate hmm Профиль откалиброван, посчитаны параметры для поиска. Длина профиля: 404
hmm2search --cpu=1 SelP_N.fasta > result_N.txt Поиск доменов во всех последовательностях
В результате поиска была получена табоица весов и E-value для некоторых последовательстей. Файл был импортирован в Excel и обработан: итоговая таблица. Были добавлены колонки о вхождении последовательностей в домен и в выборку, а также посчитаны 1-specificity и sensititvity для прошедших тестирование последовательностей.
Обсуждение результатов
График 1: Так как последовательностей много, а входит в домен всего лишь малая их часть, график очень стремительно падает. Однако можно заметить две "ступеньки" (помечены стрелками) - вторая из них соотвтетсвует минимальному score последовательностей, входящих в домен.
График 2: ROC-кривая резко переходит в стадию плато, однако можно выделить точку, соответствующую оптимальному соотношению 1-spec/sens (отмечена на графике).
График 3: По графику F1 можно также определить оптимальное соотношение specificity и sensitivity - оно находится в месте локального максимума и соотвествует весу 590,3. Это и есть пороговое значение веса для отнесения последовательности к выбранной двухдоменной архитектуре.
Найденное пороговое значение веса, 590,3, включает в двухдоменную архитектуру 56 последовательностей. 3 последовательности выходят за предельное значение и, по моим расчетам, могут не содержать два домена SelP_N и SelP_C.
Литература:
1) Volker Mostert, Selenoprotein P: Properties, Functions, and Regulation, Archives of Biochemistry and Biophysics, Volume 376, Issue 2, 2000, Pages 433-438, ISSN 0003-9861, doi.
2) Kryukov, G.V. and Gladyshev, V.N. (2000), Selenium metabolism in zebrafish: multiplicity of selenoprotein genes and expression of a protein containing 17 selenocysteine residues. Genes to Cells, 5: 1049-1060. doi
3) PFAM