Нуклеотидный BLAST
Задание 1. Определение функции и таксономии нуклеотидной последовательности.
При выполнения практикума 6 была получена консенсусная последовательность после анализа хроматограммы секвенирования по Сэнгеру с помощью программы UGENE. Для выполнения поставленной задачи был выбран именно blastn, потому что нам ничего было не известно про полученнную последовательность. Было необходимо найти последовательность, которая была бы похожа на полученную.
При поиске были использованы стандартные значение параметров: Database: Nucleotide collection (nr/nt), Max target sequences: 100, Expect threshold: 0.01, Word size: 16, Match/Mismatch Scores: 1,-2, Gap Costs: Linear. На выходе были получены следующие результаты.
Вывод о функции последовательности: данная последовательность, скорей всего, является участком гена субъединицы I цитохром оксидазы, так как именна эта находка имела E-value 0.0. Ген COI кодирует первую субъединицу мультимерного белка – цитохромоксидазы с. В дыхательной цепи эукариот этот белок образует IV, терминальный комплекс, катализирующий окисление кислорода и одновременный перенос протонов через мембрану. Субъединица COI является ключевой для осуществления белком его функции, поскольку именно в этом протеине локализуются гем- и медь-содержащие домены – активные сайты, в которых происходит окисление О2.
Вывод о таксономическом положении организма: исследуемая последовательность, вероятно, принадлежит виду-Loxosomella varians,роду-Loxosomella, семейству- Loxosomatidae, типу- Entoprocta(Внутрипорошицевые). Также необходимо добавить, что лучшие находки 2-9 принадлежат Cephalothrix simula.
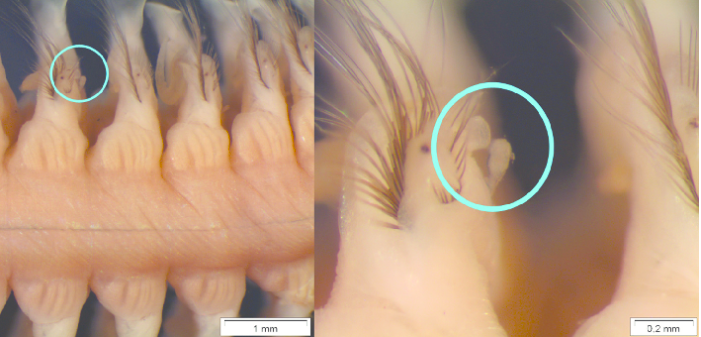
Задание 2. Поиск генов белков в неаннотированной нуклеотидной последовательности.
Для выполнения данного задания был выбран контиг из сборки генома Giraffa camelopardalis из предыдущего практикума фаста файл.
Для предсказания функции белка, закодированного в этом контиге, был выбран именно blastx, так как он переводит последовательность нуклеотидов в последовательность аминокислот и ищет сходства именно среди белков Swissprot. Парамеры поиска: database: swissprot (по достоверно существующим белкам), organism: exclude Giraffa camelopardalis (taxid:9894), expect threshold: 0.01 (находки с хорошей гомологией), word size: 6.
С результатом можно ознакомиться здесь: результаты

Первые три находки оказались ретротранспортным элементом белка ORF2, с этими находками процент индентичности был больше 60%, а E-value: 7e-105, 1e-99, 1e-71, из чего можно предположить, что часть последовательности кодирует этот белок. Затем следовали две находки, которые оказались белком черепно-лицевого развития, процент идентичности - 55%. Большинство находок принадлежали Homo sapiens и Mus musculus.
Задание 3. Интепретация карты локального сходства гомологичных хромосом двух бактерий.
При выполнении этой части практикума были выбраны две бактрии одного рода Mycobacterium: Mycobacterium tuberculosis и Mycobacterium cookii. Mycobacterium tuberculosis (палочка Коха) — возбудитель туберкулёза человека, Mycobacterium cookii - возбудитель неспецифической реакции гиперчувствительности к туберкулину у крупного рогатого скота.
Геном обеих бактерий состоит из одной полностью расшифрованной хромосомы. Для выполнения поставленной задачи использовался blastn. Использовался именно он, потому что мной были выбраны последовательности двух близкородственных бактерий(гомологи). Параметры при запуске никак не изменялись(использовались параметры, заданные по умолчанию). С результатом можно ознакомиться ниже:
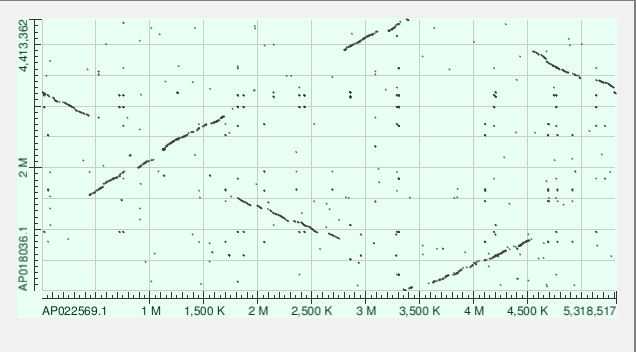
На карте видны инверсия(в самом начале 1М), также множественные индели(3,5М и 5,31М). Разрыв на 4,5М свидетельствует о том, что последовательности этих бактерий записаны, начиная с разных мест. О родственности бактерий говорят достаточно длинные схожие участки.