Трансмембранные белки
1. Знакомство с базой данных OPM:
На сайте базы данных OPM я решила выбрать трансмембранный белок с PDB ID 6x1k. Этот белок экспрессируется у Escherichia coli. Этот белок представляет собой разработанный de novo трансмембранный бета-бочонок. Белок локализирован во внешней мембране бактерии, а погруженная в мембрану часть структуры представляет собой бета-бочонок.
Координаты трансмембранных участков: 1( 9- 15), 2( 23- 31), 3( 38- 43), 4( 50- 60), 5( 66- 75), 6( 82- 93), 7( 100- 106), 8( 114- 122)
Среднее количество остатков в одном β-тяже белка: 14

| Таблица.1 Сводная таблица | |||
|---|---|---|---|
| Толщина трансмембранной части белка | 24.2 Å | ||
| Локализация | Внешняя мембрана грамм-отрицательной бактерии | ||
| Организм | Escherichia coli | ||
| PDB | 6x1k | Число трансмембранных структур | 8 |
2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка:
Я запустила сервис DeepTMHMM для выданного α-спирального белка и для выбранного мною в задании №1 β-листового белка. Результаты представлены ниже:
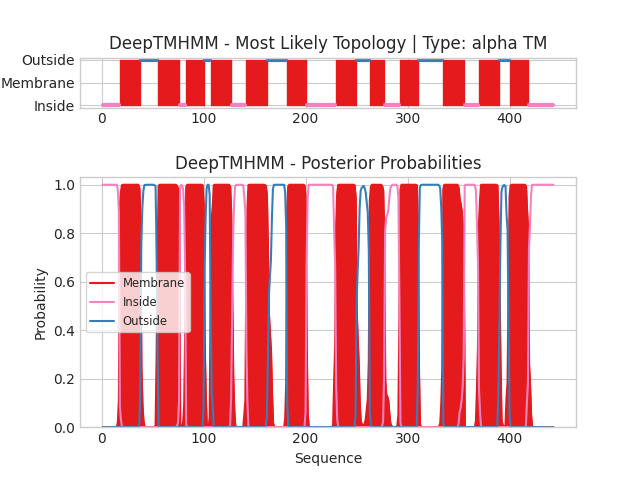
α-спиральный белок:
По оси абсцисс отложена последовательность аминокислотных остатков белка, а по оси ординат - вероятность принадлежности остатков к частям белка, расположенных: со стороны цитоплазмы(розовым), с внешней стороны клетки(синим) или же внутри мембраны(красным).
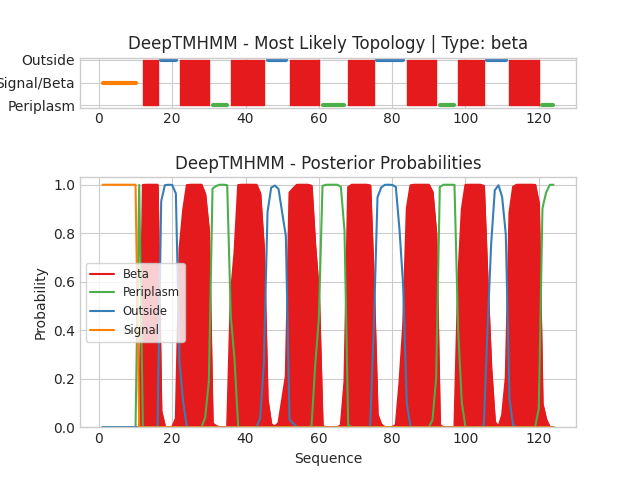
β-листовой белок:
Выдача полностью аналогична предыдущей, с тем лишь отличием, что показана принадлежность к трансмембранным частям белка(красным), периплазме(зёленым), сигнальному пептиду(оранжевым) или части, располагающейся со стороны цитоплазмы(синим).
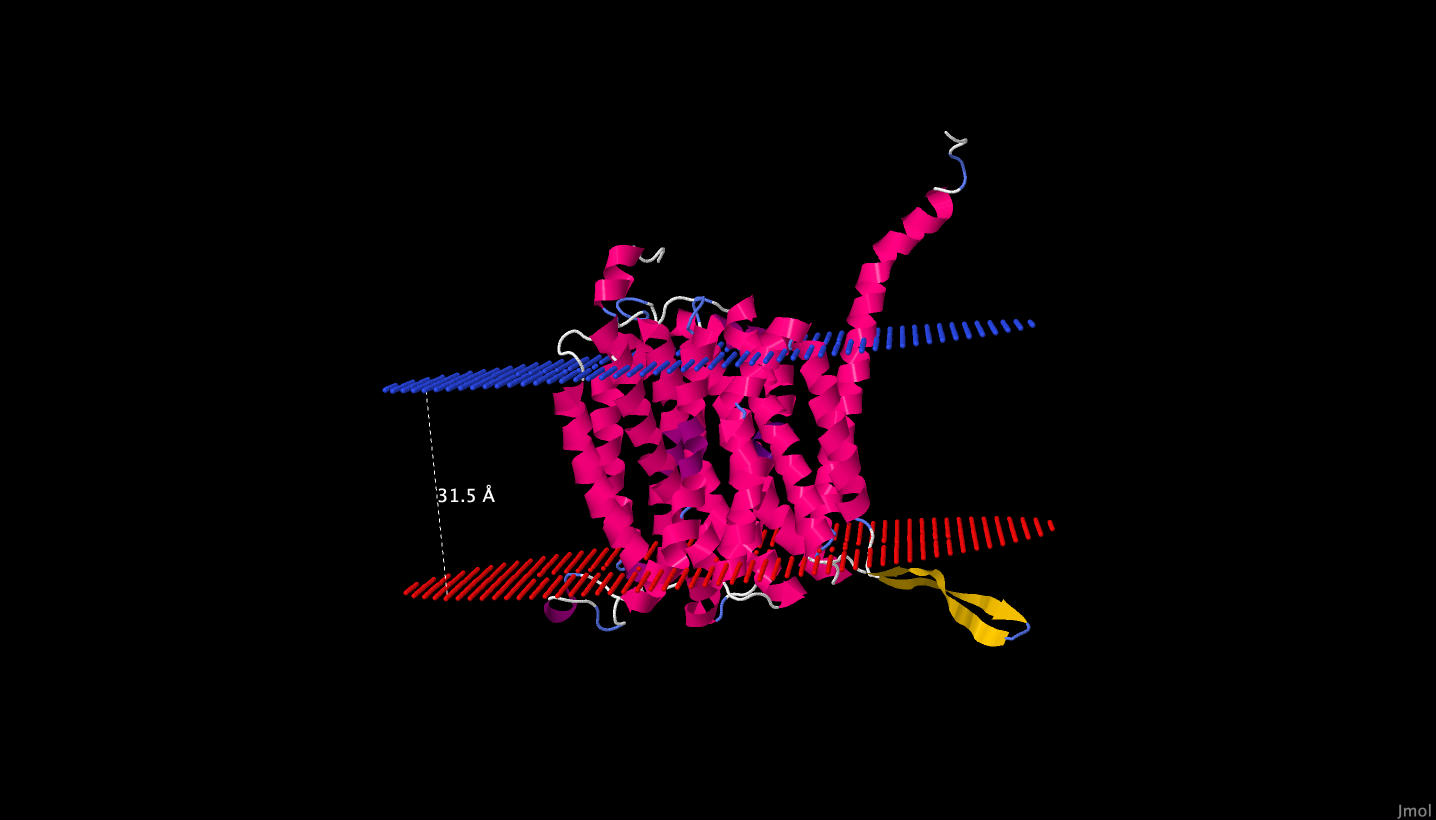
3. PPM: Предсказание положения выданного белка в мембране:
Мне был выдын белок Probable nitrate/nitrite antiporter NarK1 - Вероятный нитратно-нитритный антипортер NarK1, принадлежаший Thermus thermophilus. Идентификатор SwissProt: Q9RA46
Параметры запуска:
Number of Membranes - 1
Type of membrane - Gramm-negative bacteria inner membrane
Allow curvature - no
Topology (N-ter) - in
| Таблица.2 Выдача | |||
|---|---|---|---|
| Толщина трансмембранной части белка | 31.5 ± 0.8 Å | ||
| Локализация | Внутренняя мембрана грамм-отрицательной бактерии | ||
| Координаты трансмембранных участков | 1( 16- 40), 2( 54- 76), 3( 81- 101), 4( 107- 129), 5( 139- 160), 6( 182- 202), 7( 227- 249), 8( 261- 285), 9( 290- 309),10( 333- 355),11( 367- 391),12( 399- 421) | ||
| Число трансмембранных структур | 3 | Среднее количество остатков в одном β-тяже белка | 13 |
4. Сравнение алгоритмов предсказания трансмембранных спиралей:
Для белка 6x1k предсказания DeepTMHMM и PPM совпали почти идеально, несколько отличаются длины трансмембранных участков, вследствие чего границы тоже немного разные:
1( 9- 15), 2( 23- 31), 3( 38- 43), 4( 50- 60), 5( 66- 75), 6( 82- 93), 7( 100- 106), 8( 114- 122) - Данные PPM
1( 12- 16), 2( 22- 30), 3( 36- 45), 4( 52- 60), 5( 68- 75), 6( 84- 92), 7( 98- 105), 8( 112- 120) - данные DeepTMHMM
Сравнение предсказания для Q9RA46 в DeepTMHMM и PPM:
1( 16- 40), 2( 54- 76), 3( 81- 101), 4( 107- 129), 5( 139- 160), 6( 182- 202), 7( 227- 249), 8( 261- 285), 9( 290- 309),10( 333- 355),11( 367- 391),12( 399- 421) - Данные PPM
1( 18- 38), 2( 55- 76), 3( 83- 100), 4( 107- 127), 5( 142- 162), 6( 182- 200), 7( 230- 249), 8( 263- 277), 9( 293- 310),10( 335- 355),11( 370- 390),12( 400- 418) - данные DeepTMHMM
Различия в резульате DeepTMHMM и данных OPM связаны с различным началом отсчета, т. е. DeepTMHMM учитывает начальную сигнальную последовательность в 25 а. о. , тогда как данные из OPM начинают отсчет с первого а. о. первого бета-листа
PPM предсказывает трансмембранные участки а основе "встраивания" белка в определенную мембрану (в данном случае мембрана грам-отрицательной бактерии), тогда как DeepTMHMM делает предсказание только на основе свойств а. о. и не имеет никаких "представлений" об особенностях различных мембран. Этим мжно объяснить небольшие различия в гранцах трансмембранных участков, предсказанных этими методами.
5. База данных TCDB:
β-листовой белок:
TC accession: 2.A.1.8.8
2. Электрохимические переносчики, управляемые потенциалом
2.А Портеры (унипортеры, симпортеры, антипортеры)
2.A.1 Суперсемья главных фасилитаторов
2.A.1.8.8 NO2-экструзия, NO3-/NO2- обменная пермеаза