Сигналы и мотивы-2
Задание 1.
Для выполнения этого задания был выбран поиск последовательности сайта посадки сигма-субъединицы в хромосоме бактерии Escherichia coli str. K-12 substr. MG1655 (E. coli). Была выбрана именно эта бактерия, так как её очень часто используют в научных работах, она является, как мне кажется, одним из самых распространенных модельных организмов, хотелось провести исследование, чтобы вынести что-то новое для себя о столь изуенном.
С NCBI Genome был скачан геном бактерии и таблица генов в формате gff. Из файлов была удалена информация не относящаяся к хромосоме. Для поиска оперонов был использован сервис operon-mapper. Его выдача доступна по ссылке. После этого был использован скрипт Смирнова.М, принимающий на вход fasta-файл с хромосой и файл list_of_operons.txt, и выдающий 2 fasta-файла: один со всеми промоторами - 150-нуклеотидными участками перед первым страрт-кодоном оперона, другой с сотней случайных промоторов. Первый использовался в качестве входной последовательности для FIMO, а второй - для MEME (использовалась веб-версия). Параметры программы MEME настроены следующим образом: поиск 3 мотивов, встречающихся 0 или 1 раз в каждой последовательности, длина мотива от 25 до 60 нуклеотидов, поиск только по данной цепи. Выдача MEME доступна по ссылке.
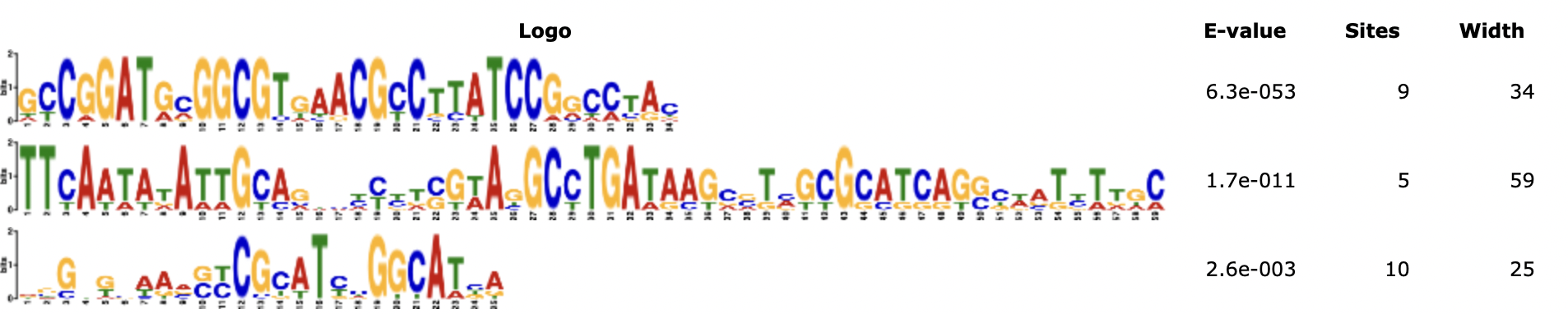
<
Было найдено 3 мотива с достаточно хорошим e-value. Скорее всего, сайт богатый аденинами соответсвует -35 боксу, а последующий малостабильный участок - спейсеру, а -10 бокс попал в мотив лишь частично. В качестве входного в FIMO(Выдача) был использован мотив с наилучшим e-value. Было найдено 444 находок, что свидетельствует о том, что либо этот мотив не имеет отношения к сайту посадки сигма-субъединицы, либо недостаточно полно и точно его описывает. При запуске FIMO был выставлен порог на p-value по умолчанию.