Внутримолекулярные взаимодействия в структуре 1YDM
Общая характеристика структуры 1YDM
Идентификаторы белка в PDB: 1YDMНазвание белка: Гипотетический белок yqgN (hypothetical protein yqgN)
Количество разных цепей в PDB-файле: 3 (A, B и C)
В структуре белка 1YDM содержится 3 цепи. Помимо собственно белка и воды в PDB-файле приведены координаты атомов трех сульфат ионов, находящихся по одному в каждой из цепей белка. Расположение сульфат-ионов в структуре 1YDM представлено на рис.1.
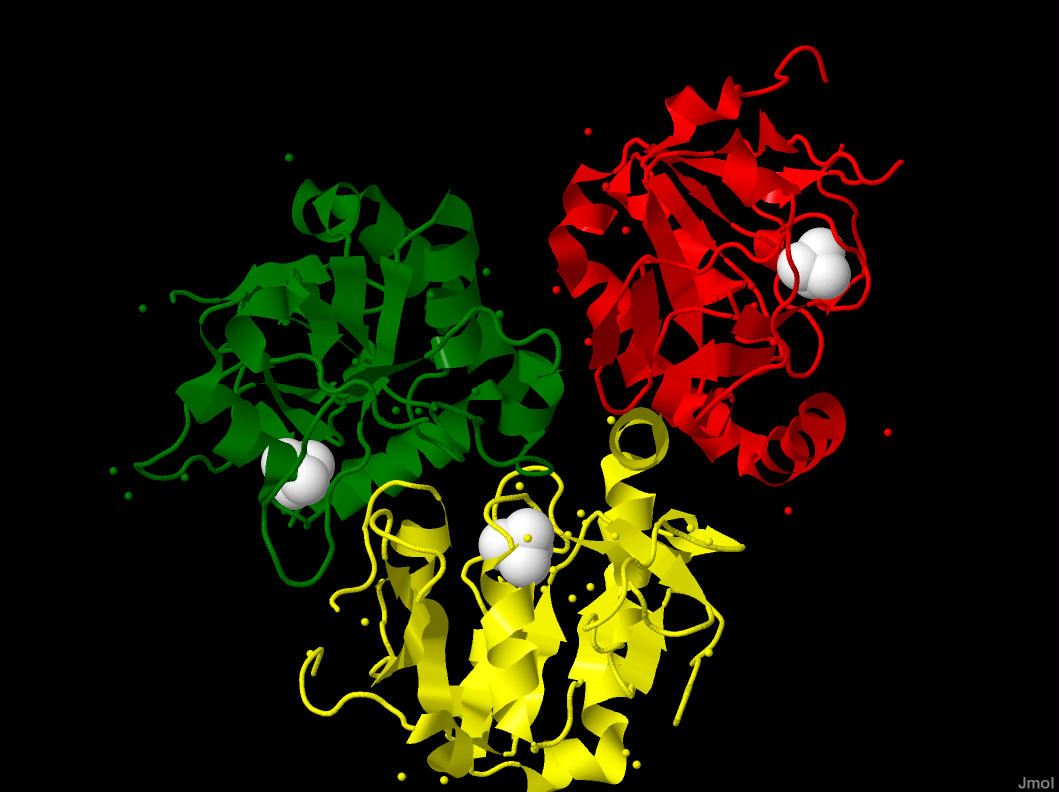
рис.1 Структура белка 1YDM.
Красным цветом выделена цепь А, зеленым цветом - С, а желтым цветом цепь В. Белыми "шариками" в каждой цепи выделены атомы сульфат иона. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)
Общий вид структуры 1YDM представлен на рис.2.
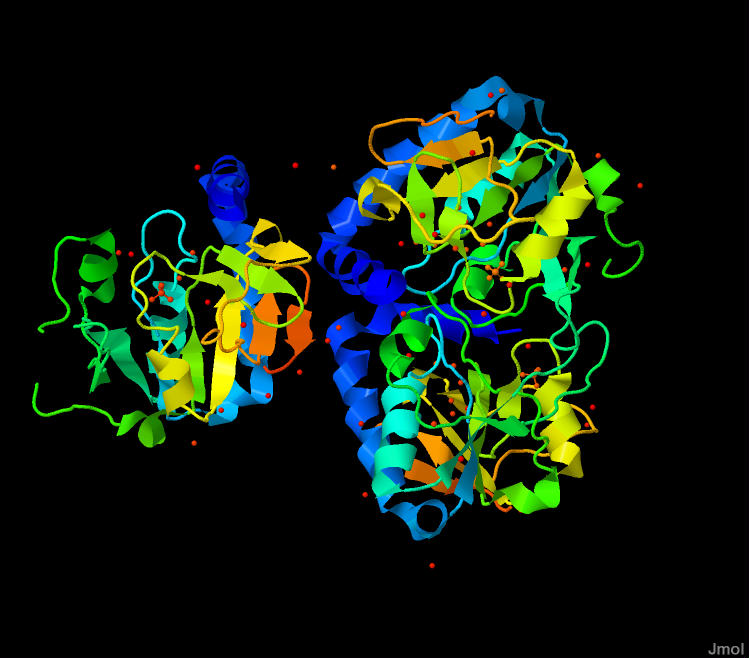
рис.2 Структура белка 1YDM.
Последовательность расположения различных элементом структуры 1YDM от N- до С-конца показаны при помощи цветового перехода от синего к оранжевому соответственно. В явном виде показана вторичная структура всех белковых молекул при помощи команды cartoon. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)

Анализ структуры альфа-спиралей и бета-листов в цепи A белка с PDB-кодом 1YDM
Для анализа структур альфа-спиралей и бета-листов в белке 1YDM выбрана A цепь. В ней выбрана альфа-спираль с 55 по 66 аминокислотный остаток и вета-лист с тремя бета-тяжами.
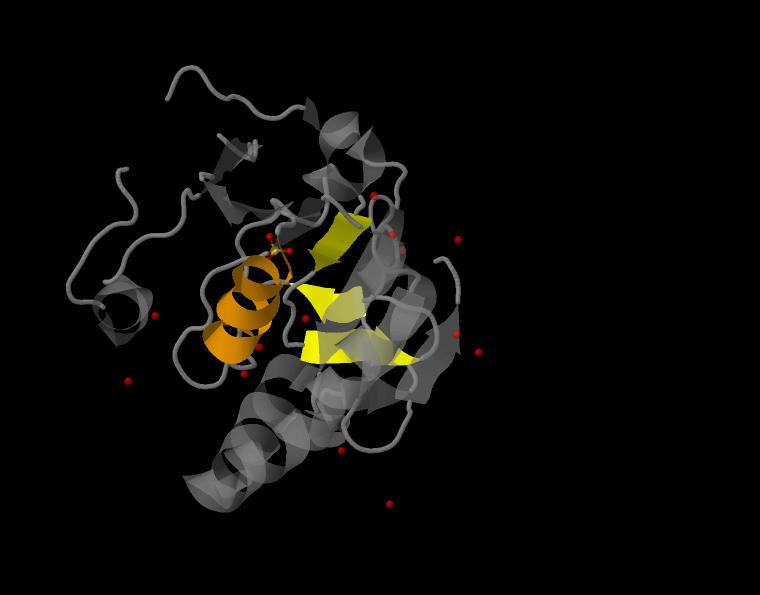
рис.3 На рисунке показано нахождение структуры альфа-спирали с 56 по 66 аминокислотный остаток и структуры бета-листа с 3 бета-тяжами (118-120, 125-128, 132-134 аминокислотные остатки) белка 1YDM. Изображение получено с помощью программы Jmol.
(увеличенное изображение при клике на картинке)
На рис.4 отдельно представлена α-спираль с 56 по 66 аминокислотный остаток цепи A белка 1YDM.
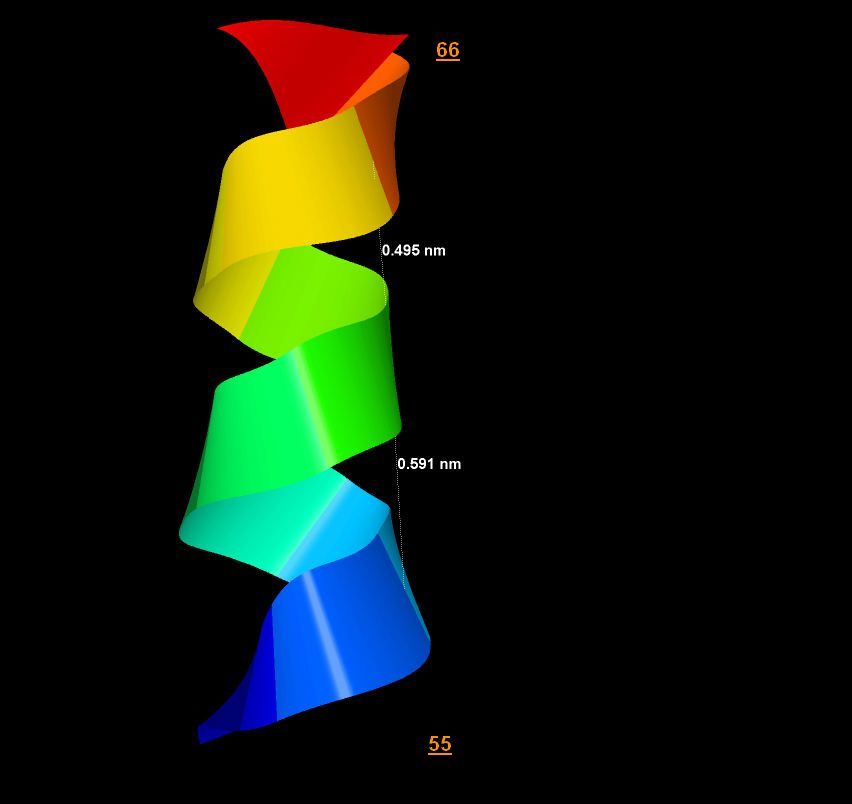
рис.4 Структура альфа-спирали с 56 по 66 аминокислотный остаток белка 1YDM в программе Jmol. Все аминокислотные остатки покрашены в разные цвета. На рисунке подписаны витки спирали в нанометрах(nm).
Средний виток спирали равен
(4.95Å + 5.91Å)/2 = 5.43Å
(Увеличенное изображение при клике на картинке)
На 3 витка альфа-спирали приходится 11 аминокислотных остатка. Из этого следует, что число остатков спирали на один виток равно 11/3, то есть приблизительно 3.7 . Таким образом, остаток с номером n в середине спирали образует водородные связи с n+4 и n-4 аминокислотным остатком. На рис.5 показано, что остаток с номером 61 (n) образует водородные связи с 57 (n-4) и 65 (n+4) остатком.
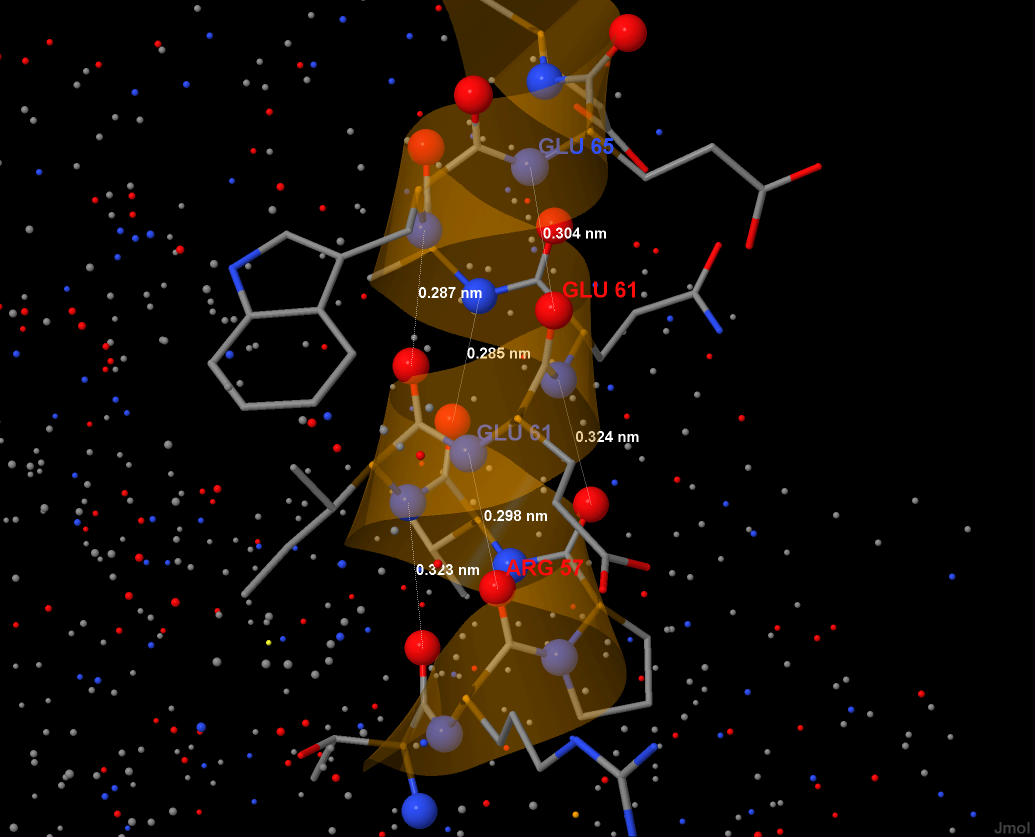
рис.5 Структура альфа-спирали с 56 по 66 аминокислотный остаток белка 1YDM. На рисунке подписаны длины водородных связей в нанометрах (nm).
Для некоторых атомов, участвующих в образование водородных связей подписаны аминокислотные остатки и их номер. Явно видно, что остаток с номером 61 образует водородные связи с 57 и 65 остатком. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)
Посчитаем среднюю длину водородных связей в альфа- цепи: (3.04Å + 2.87Å + 2.85Å + 3.24Å + 3.23Å + 2.98Å)/6 = 3.04Å .
Рассмотрим β-лист с тремя β-тяжами (118-120, 125-128, 132-134 аминокислотные остатки).

рис.6 Бета-лист с тремя бета-тяжами (118-120, 125-128, 132-134 аминокислотные остатки) цепи A белка 1YDM. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)
Тяжи 125-128 и 132-134 аминокислотных остатков антипараллельны, так как "стрелочки", то есть направление от N-конца к С-концу смотрят в разные стороны.
Внутримолекулярные взаимодействия боковых групп белка в цепи A структуры 1YDM
В белке 1YDM есть по 5 цистеиновых остатков (Cys) в каждой из трех цепей. Нахождение Cys в белке представлено на рис.7
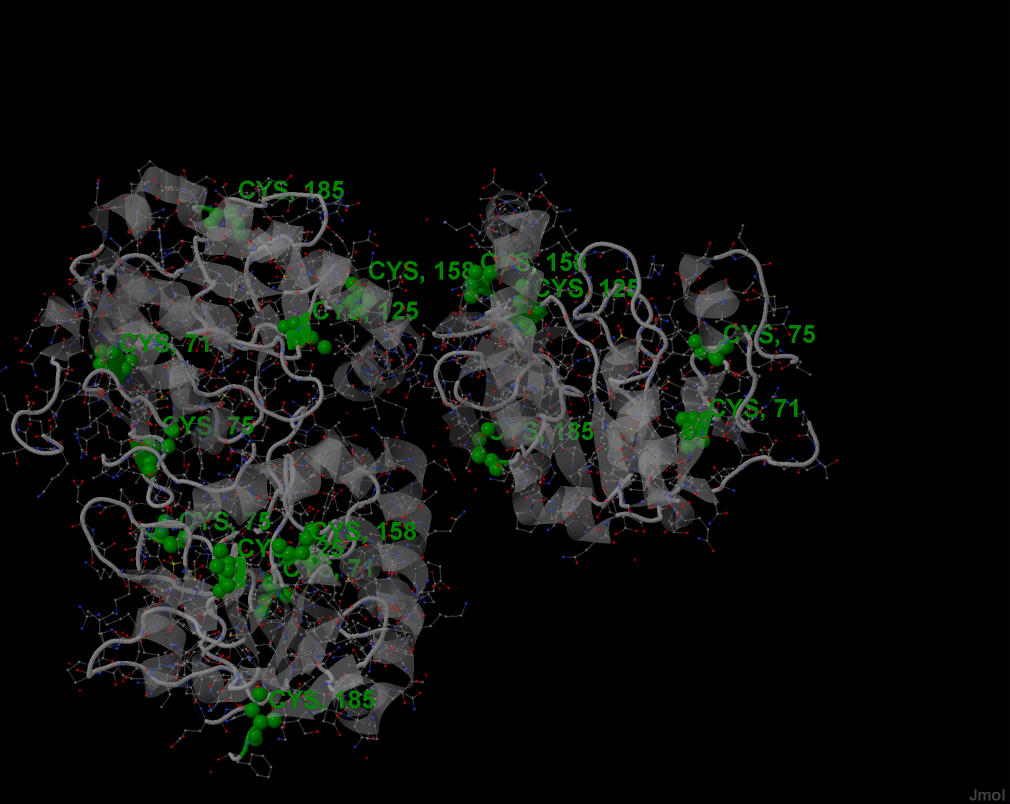
рис.7 Полная структура белка 1YDM. Для удобства и понятности изображения вторичная структура белка покрашена в серый цвет и сделана прозрачной.
Зеленым цветом выделены и подписаны цистеины с порядковами номерами. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)
Для рассмотрения в подробностях возможных дисульфидных связей между цистеинами была выбрана цепь A. Были померяны расстояния между атомами серы соседних цистеинов. Результаты приведены на рис.8.
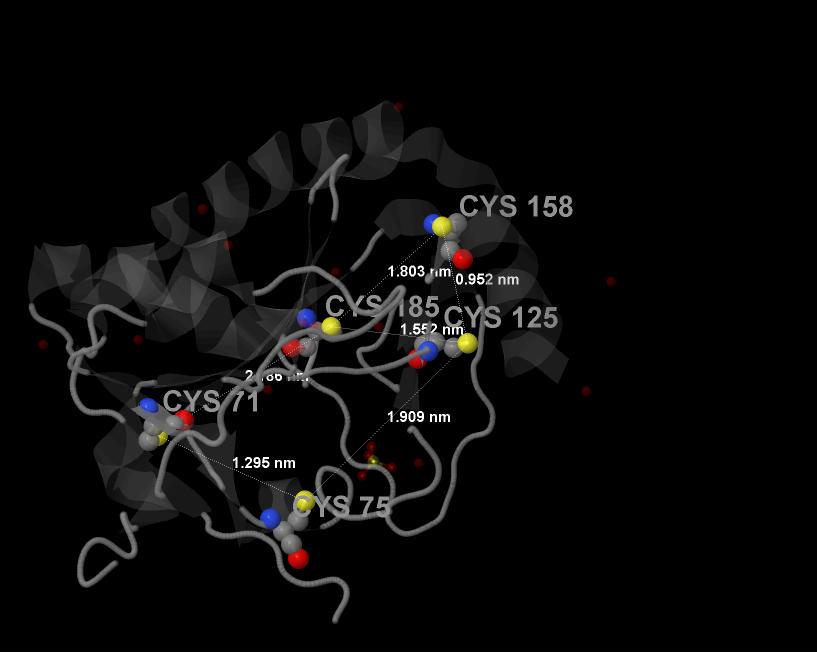
рис.8 Полная структура белка 1YDM. Для удобства и понятности изображения вторичная структура белка покрашена в серый цвет и сделана прозрачной.
Цистеин подписан его трехбуквенным обозначением (Cys), а так же порядковым номером. Атомы цистеина представлены в виде сфер и покрашены по их химической природе: атомы серы - в желтый, азота - в синий, кислорода - в красный, углерода - в серый. На рисунке в явном виде подписаны расстояния в нанометрах (nm) между атомами серы соседних цистеинов. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)
Из рис.8 видно, что минимальное расстояние между атомами серы соседних цистеинов равно 9.52 Å , что довольно много для образования дисульфидных связей. Для полной уверенности в этом была использована встроенная в Jmol команда ssbonds, которая подтвердила, что в белке 1YDM нет дисульфидных связей.
Солевые мостики образуются между отрицательно(Asp, Glu) и положительно заряженными аминокислотами (Lys, Arg и His). Для того чтобы найти солевые мостики, при помощи команды within найдем те заряженнные аминокислоты, расстояние между которыми меньше 3.5 Å друг от друга. Результаты показаны на рис.9.
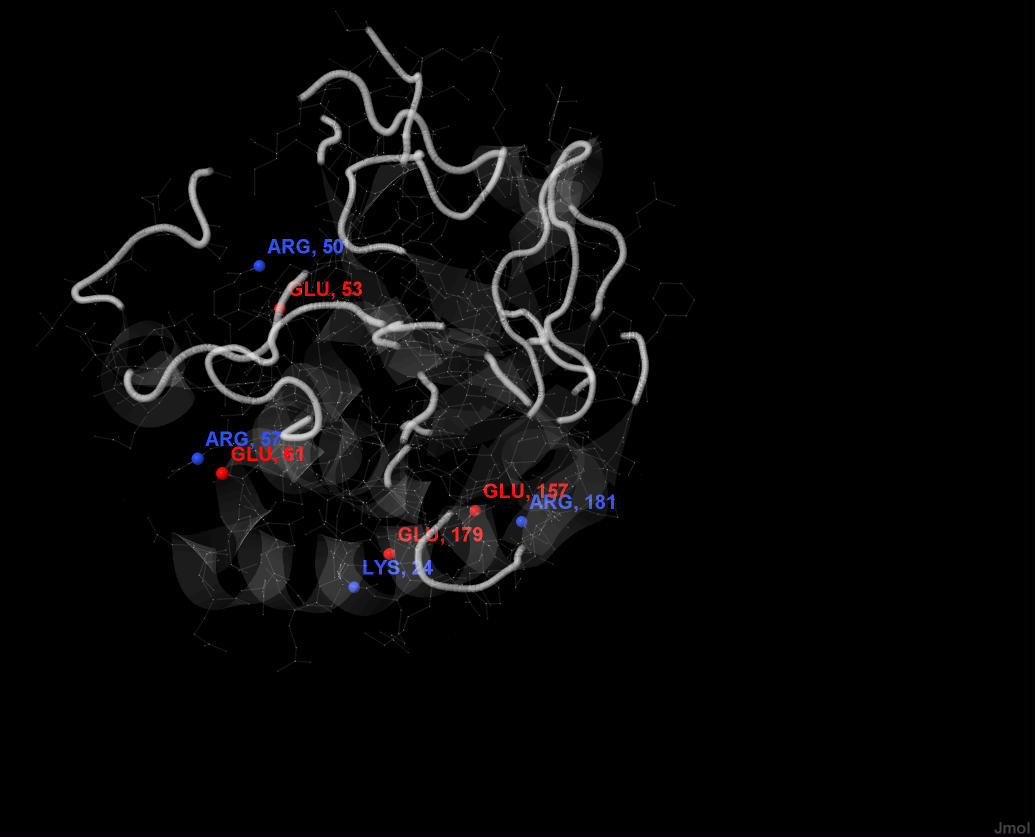
рис.9 Цепь А структуры белка 1YDM. Выделены атомы кислорода у отрицательно заряженнных аминокислоты и атомы азота у положительно заряженных аминокислот, расстояние между которыми меньше 3.5 Å друг от друга. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)
Отдельный солевой мостик показан крупно на рис.10.
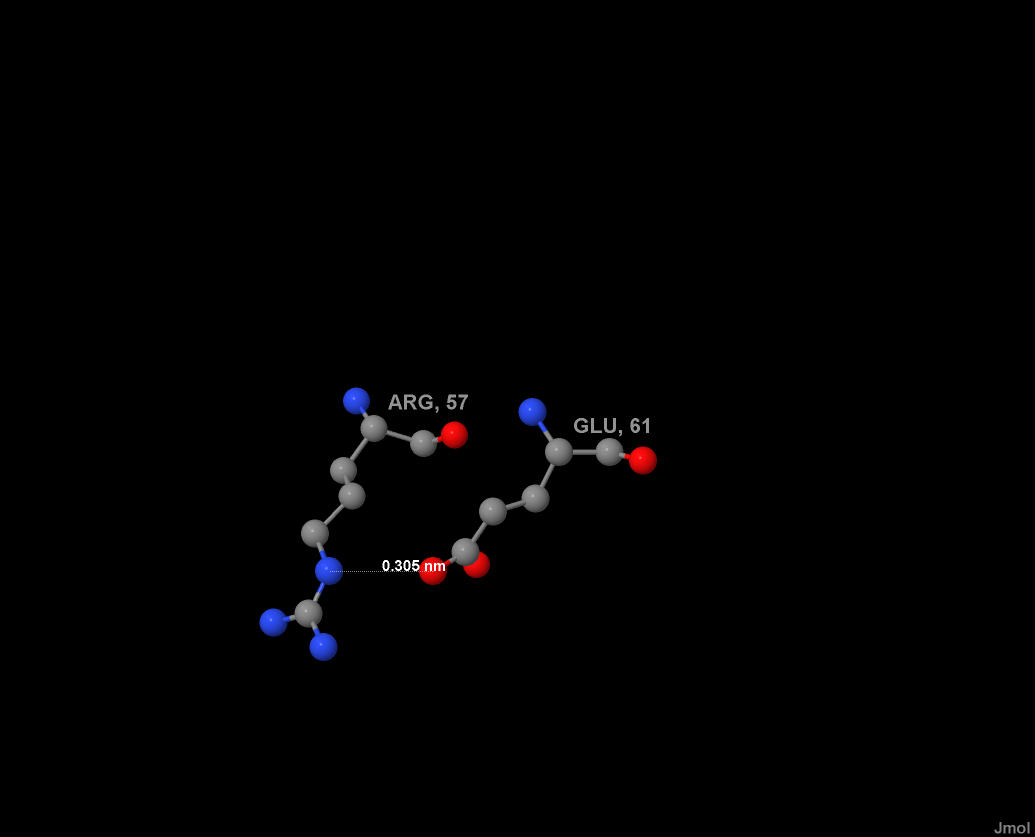
рис.10 Солевой мостик между 57 остатком аминокислоты аргинина (Arg) и 61 остатком глутаминовой кислоты (Glu). Атомы аминокислот представлены в виде сфер и покрашены по их химической природе: атомы азота - в синий, кислорода - в красный, углерода - в серый. На рисунке в явном виде подписано расстояние в нанометрах (nm) между атомами кислорода и азота Glu и Arg соответственно. Изображение получено с помощью программы Jmol.
(Увеличенное изображение при клике на картинке)
Таким образом, в структуре белка 1YDM есть четыре солевых мостика.
© Nuzhdina Ekaterina, 2012
