BLAST
Поиск гипотетических гомологов изучаемого белка в разных банках
Рассмотрим web- интерфейс программы BLAST. В разделе "Basic BLAST" переходим по гиперссылке "protein blast".В поле "Enter Query Sequence" можно ввести accession number, gi, или FASTA последовательность. Для нашей задачи введем в это поле accession нашего белка: P54491.
Далее мы можем варьировать параметры нашего запроса: Database - можем менять базу данных, в которой будем искать наши варавнивания с запрошенным белком. В дополнительной вкладке "Algorithm parameters" можем поменять максимальное колличество выводимых выравниваний ("Max target sequences"), максимальное значение e-value ("Expect threshold"), а так же еще много интересных вещей, но в данном задании мы пользоваться ими не будем.
Будем искать выравнивания в трех базах данных: swissprot, PDB, nr.
Банк nr (Non-redundant protein sequences) — своеобразный "виртуальный" банк, существующий только как область поиска программой BLAST на сайте NCBI. Включает в себя все белковые последовательности из всевозможных источников (в первую очередь Swiss-Prot и аннотации кодирующих участков генов в GenBank).
После того, как мы сформировали запрос нажимаем кнопку "BLAST" и ждем некоторое время, пока у нас не появится список находок. Чтобы узнать номер находки в списке описаний, наведем курсор мыши на "Accession" (гиперссылку в правом столбце таблицы). В "строке состояния" (внизу окна браузера) отобразится гиперссылка, в которой будет указано "blast_rank=...", значит, курсор на ...-ой находке.
(Результаты находок для белка YQGN_BACSU в трех базах данных приведены в табл.1.)
Табл. 1. Результаты поиска гипотетических гомологов белка YQGN_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession | P54491.1 | 1YDM_A | NP_390369.1 |
| E-value | 3e-138 | 4e-136 | 2e-136 |
| Вес (в битах) | 390 | 382 | 390 |
| Процент идентичности | 100% | 98% | 100% |
2. Число находок с E-value < 10–10 |
11 | 5 | 2325 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 16 | 10 | 3916 |
| Accession | A5FIV1.1 | 3MA6_A | YP_003177494.1 |
| E-value | 0.84 | 0.95 | 1.0 |
| Вес (в битах) | 33.5 | 30.0 | 38.1 |
| % идентичности | 27 | 32 | 25 |
| % сходства | 50 | 48 | 41 |
| Длина выравнивания | 81 | 56 | 161 |
| Координаты выравнивания (от-до, в запросе и в находке) | В запросе: 48-125 В выравнивании: 443-519 |
В запросе: 17-64 В выравнивании: 236-290 |
В запросе: 38-187 В выравнивании: 49-204 |
| Число гэпов | 7 | 9 | 16 |
Число явных гомологов (E-value < 1e-10) при поиске по разным БД существенно различаются, как мы видим, что вполне объяснимо. Банк nr содержит в себе данные и из swiss-prot и из PDB, самое большое колличество находок именно в нем. В swiss-prot нашлось больше находок, чем в PDB, так как не для всех белков есть данные рентгеноструктурного анализа и соответсвующие 3-D структуры.
Рассмотрим колличество находок для разных баз данных и E-value самой последней находки, в каждой из них. (Результаты в табл.2.)
Табл. 2. Исследование результатов поиска гипотетических гомологов белка YQGN_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
| Всего находок | 28 | 19 | 4080 (порог на число находок 5000, на e-value 10, если его увеличить находки еще будут) |
| E-value последней находки | 9.5 | 8.9 | 9.7 |
| Чем лимитировано? | e-value(так как по умолчанию оно выводит находки с <10) , а число находок ограничено лишь 100. После изменения e-value на <40 последней находкой стала с e-value 23. |
e-value | e-value, так как находок <<5000, а expect немногим меньше 10. |
Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
Табл. 3. Поиск гипотетических гомологов белка YQGN_BACSU с фильтром по таксонам: Eukaryota. Матрица BLOSUM 62.| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
Гипотетические гомологи (E-value < 1e-3) |
|||
| Accession | Q9P7W2.1 | 3HXT_A | CCU99308.1 |
| E-value | 6e-18 | 1e-14 | 9e-21 |
| Вес (в битах) | 80.9 | 68.6 | 93.6 |
| % идентичности | 29 | 26 | 29 |
| % сходства | 50 | 51 | 48 |
| Длина выравнивания | 197 | 190 | 216 |
| Координаты выравнивания (от-до, в запросе и в находке) | В запросе: 2-183 В выравнивании: 5-201 |
В запросе: 2-180 В выравнивании: 10-199 |
В запросе: 2-184 В выравнивании: 8-223 |
| Число гэпов | 15 | 11 | 33 |
BLAST двух последовательностей
С помощью программы BLAST сделаем парное выравнивание белка 1YDM и его гомолога, найденного в предыдущей части(CCU99308.1).Сравнение результатов поиска с различными матрицами BLOSUM
Так как цифры, стоящии после BLOSUM означают порог идентичности(%) на котором кластеризуют последовательности, то становится очевидным, что для наиболее похожих последовательностей целесообразней использовать матрицу с меньшим порогом, а для более различающихся - с меньшим.Табл. 4. Поиск гипотетических гомологов белка YQGN_BACSU с фильтром по таксонам: Eukaryota. Матрица BLOSUM 90.
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
Гипотетические гомологи (E-value < 1e-3) |
|||
| Accession | Q9P7W2.1 | 3HXT_A | CCU99308.1 |
| E-value | 4e-17 | 7e-14 | 2e-19 |
| Вес (в битах) | 83.2 | 70.7 | 95.4 |
| % идентичности | 29 | 26 | 29 |
| % сходства | 44 | 43 | 43 |
| Длина выравнивания | 197 | 190 | 216 |
| Координаты выравнивания (от-до, в запросе и в находке) | В запросе: 2-183 В выравнивании: 5-201 |
В запросе: 2-180 В выравнивании: 10-199 |
В запросе: 2-184 В выравнивании: 8-223 |
| Число гэпов | 15 | 11 | 33 |
E-value(E): E = kmne-λs, где λ и k - это нормировка на матрицу BLOSUM, m - длина запроса, n - длина банка данных, s - вес выравнивания.
Bit score(S'):
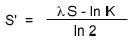
E = mn2-s'
Из Табл.5. видно, что e-value при переходе от матрицы BLOSUM 62 к матрице ВLOSUM с порогом кластеризации 90 возрастает. Возрастает так же вес в битах, а % сходства напротив уменьшается.
Табл. 5. Сравнение значения для гипототетических гомологов по матрице BLOSUM 90 и 62.
| Параметр | BLOSUM 62 | BLOSUM 90 | |
Поиск по Swiss-Prot |
|||
| E-value | 6e-18 | 4e-17 | |
| % сходства | 50 | 44 | |
| вес в битах | 80.9 | 83.2 | |
Поиск по pdb |
|||
| E-value | 1e-14 | 7e-14 | |
| % сходства | 51 | 43 | |
| вес в битах | 68.6 | 70.7 | |
Поиск по "nr" |
|||
| E-value | 9e-21 | 2e-19 | |
| % сходства | 48 | 43 | |
| вес в битах | 93.6 | 95.4 | |
Полезные ссылки:
© Nuzhdina Ekaterina, 2012