Эволюционные домены. Банки Pfam и InterPro.
Доменная структура белка YQGN_BACSU по данным Pfam.Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов | Положение в последовательности белка | Клан |
| 1. | PF01812 | 5-FTHF_cyc-lig | Семейство доменов названо по ферменту 5-формилтетрагидрофолату цикло-лигазе, катализирующему соединение двух молекул с образованием новой химической связи между азотом и углеродом ( превращение 5-формилтетрагидрофолата (5-FTHF) в 5-10-метенилтетрагидрофолат). | 2–179 | Клан ISOCOT_Fold (CL0246), содержит 11 семейств: 5-FTHF_cyc-lig, AcetylCoA_hyd_C, AcetylCoA_hydro, CitF, CoA_trans, DeoRC, DUF162, Glucosamine_iso, IF-2B, Rib_5-P_isom_A, Sugar-bind. |
Данные о домене 5-FTHF_cyc-lig
Домен входит в 19 архитектур. Для 4810 белков, содержащих домен, известна последовательность.
Для 6 разных белков, содержащих домен, определена пространственная структура (домена или всего белка).
Сохраним выравнивание "seed" фрагментов белков, соответствующих домену. Для этого в
меню "Alignments", выбераем формат MSF, Seed (а не Full — все последовательности), "Generate" и "Download".
Выравнивание "seed" фрагментов белков, соответствующих домену доступно для скачивания в формате MSF.
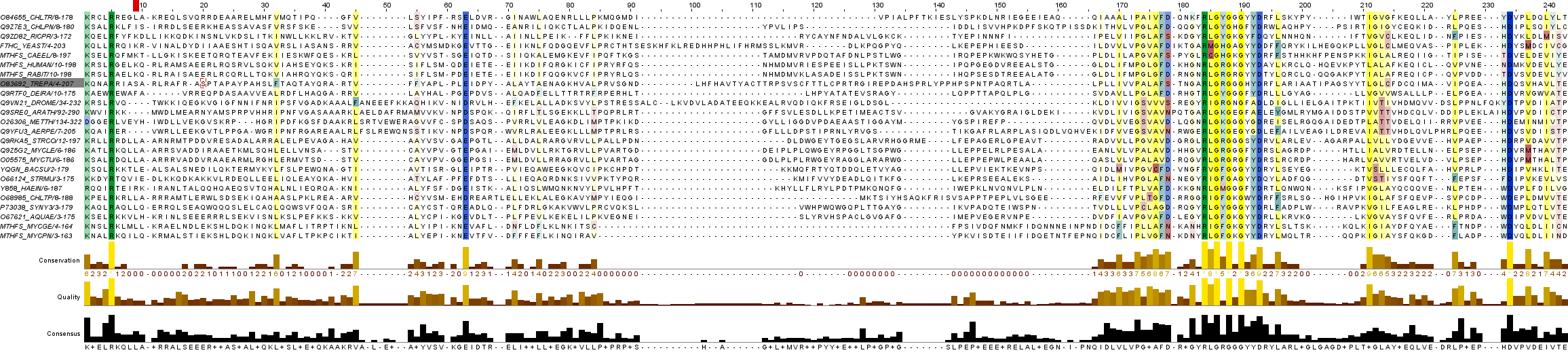
Рис.1. Визуализация множественного выравнивания "seed" фрагментов белков в JalView
(Увеличенное изображение при клике на картинке)
(Увеличенное изображение при клике на картинке)

Из рис.1. мы видим, что выравнивание очень похоже на достоверное, так как основные участки белков выравнялись достаточно хорошо (особенно те, которые участвуют в связывание лиганда SO4 - подребнее о нем можно узнать здесь в разделе "Анализ 3D-структуры белка исходя из выравнивания"). На основание этого можно с большой уверенностью предположить гомологию.
Данные о доменной архитектуре
Теперь рассмотрим доменную архитектуру, состоящую из двух доменов : IATP, 5-FTHF_cyc-lig из Drosophila sechellia (Fruit fly). Для того, чтобы получить данные о структуре домена нужно кликнуть по нему и перейти на страницу
с описанием. Далее переходим по ссылке
"Species" => "Tree". Выбираем "Expand to depth" = 2.
Для того, чтобы получить данные о структуре домена нужно кликнуть по нему и перейти на страницу
с описанием. Далее переходим по ссылке
"Species" => "Tree". Выбираем "Expand to depth" = 2.
Табл.2. Представленность домена PF04568 в организмах разных таксонов( http://pfam.sanger.ac.uk//family/PF04568#tabview=tab7)
|
Таксон
|
Количество белков с доменом PF04568
|
|
| Эукариоты | Зеленые растения | 15 |
| Грибы | 111 | |
| Животные | 92 | |
| Остальные эукариоты | 5 | |
| Археи | 0 | |
| Бактерии | 3 | |
| Вирусы | 0 | |
Табл.3. Представленность домена PF01812 в организмах разных таксонов( http://pfam.sanger.ac.uk//family/PF01812#tabview=tab7)
|
Таксон
|
Количество белков с доменом PF01812
|
|
| Эукариоты | Зеленые растения | 25 |
| Грибы | 127 | |
| Животные | 92 | |
| Остальные эукариоты | 33 | |
| Археи | 85 | |
| Бактерии | 4113 | |
| Вирусы | 0 | |
Как видно из табл.3. домен PF04568 почти не представлен у бактерий и совсем отсутствует у вирусов и архей. Из табл.4. видно, что домен PF01812 совсем не представлен у вирусов и очень распростарнен среди бактерий, в противоположенность домену PF04568. Оба домена примерно одинаково распределены по таксонам Эукариот.
Сравнение описания мотивов в разных банках семейств, по данным InterPro
Рис.2 Подписей (signatures), интегрированные в InterPro
(Увеличенное изображение при клике на картинке)

(Увеличенное изображение при клике на картинке)

| Самый короткий мотив? В каком банке он описан? | (Семейство IPR002698) 5-FTHF_cyc-lig, accessesion PF01812 - описан в банке Pfam и MTHFS_bact, accession TIGR02727 - описан в банке TIGRFAMs |
| Как называется самый длинный мотив? В каком банке он описан? | (Семейство IPR002698) PTHR23407:SF1 - Описан в банке PANTHER |
Рис.3 Структурные подписи, интегрированные в InterPro
(Увеличенное изображение при клике на картинке)
(Увеличенное изображение при клике на картинке)

Список литературы:

