Множественное выравнивание
Создание репрезентативной выборки гомологов белка YQGN_BACSU
В поле Organism для blastp введем название филума, к которому принадлежит бактерия, в которой
содержится белок YQGN_BACSU: Firmicutes. Далее выставим галочку напротив "Exclude", тем самым
запретим BLAST выдавать сходные белки из того же филума.
Добавим еще одно поле Organism, щелкнув на "+" справа от первого поля. Исключим еще Eukaryota.
Порог e-value оставим пока высоким: 1. В качестве базы данных будем использовать
RefSeq (выберем ее в поле "Database"). В поле "Max target sequences" выставим 5000.
В итоге мы получили 1866 хитов у которых e-value ≤ 9e-6 (следующее за ним 10e-5,
что по просмотренным выравниваниям уже много для гомологии), поэтому еще раз запустим BLAST
с теми же параметрами, но поставим "Expect threshold" на 9e-6 (следующее за ним 10e-5).
После этого в секции Descriptions окна выдачи BLAST выберем все последовательности (Select: All) и
получим о них выдачу GenBank (ссылка GenPept прямо под графическим изображением хитов). Но у нас ничего
не получится, так как GenPept не может обрабатывать такие большие запросы. Поэтому скопируем из
строки адреса сам запрос. Там будут содержаться accessions всех наших гомологов.
Разобъем теперь этот список на несколько
частей и прогоним их по базе http://www.ncbi.nlm.nih.gov/protein/.
Щелкая по названиям таксонов в правой стороне экрана, получим только те хиты, которые им принадлежат.
В итоге мы получим 6 белков, принадлежащих филумам архей, и 23 - бактериям. Записываем индентификаторы полученных
в базе RefSeq белков. Теперь можем скопировать их в строку запроса базы данных RefSeq и скачать последовательности
в fasta-формате всех белков сразу. Файл c последовательностями гомологов YQGN_BACSU из Prokaryota
в fasta-формате также можно найти в приложениях внизу страницы*1.
Повторим такой же поиск гомологов, но уже только по Eykaryota. Мы получим 144 хита с e-value ≤ 10e-5.
С таким количеством последовательностей GenPept уже справится, поэтому можем воспользоваться им при поиске белков из разных царств.
В итоге мы получим 10 белков, принадлежащих разным царствам Eukaryota.
Файл c последовательностями белков гомологов YQGN_BACSU из Eykaryota
в fasta-формате также можно найти в приложениях внизу страницы*2.
В Табл.1 приведены параметры запросов для разных доменов.
Параметры BLAST |
|||||
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | protein blast (blastp) | RefSeq | Исключаем таксоны: Eukaryota Firmicutes |
9e-6 | 1866 |
| По эукариотам | protein blast (blastp) | RefSeq | Только по Eukaryota | 10e-5 | 144 |
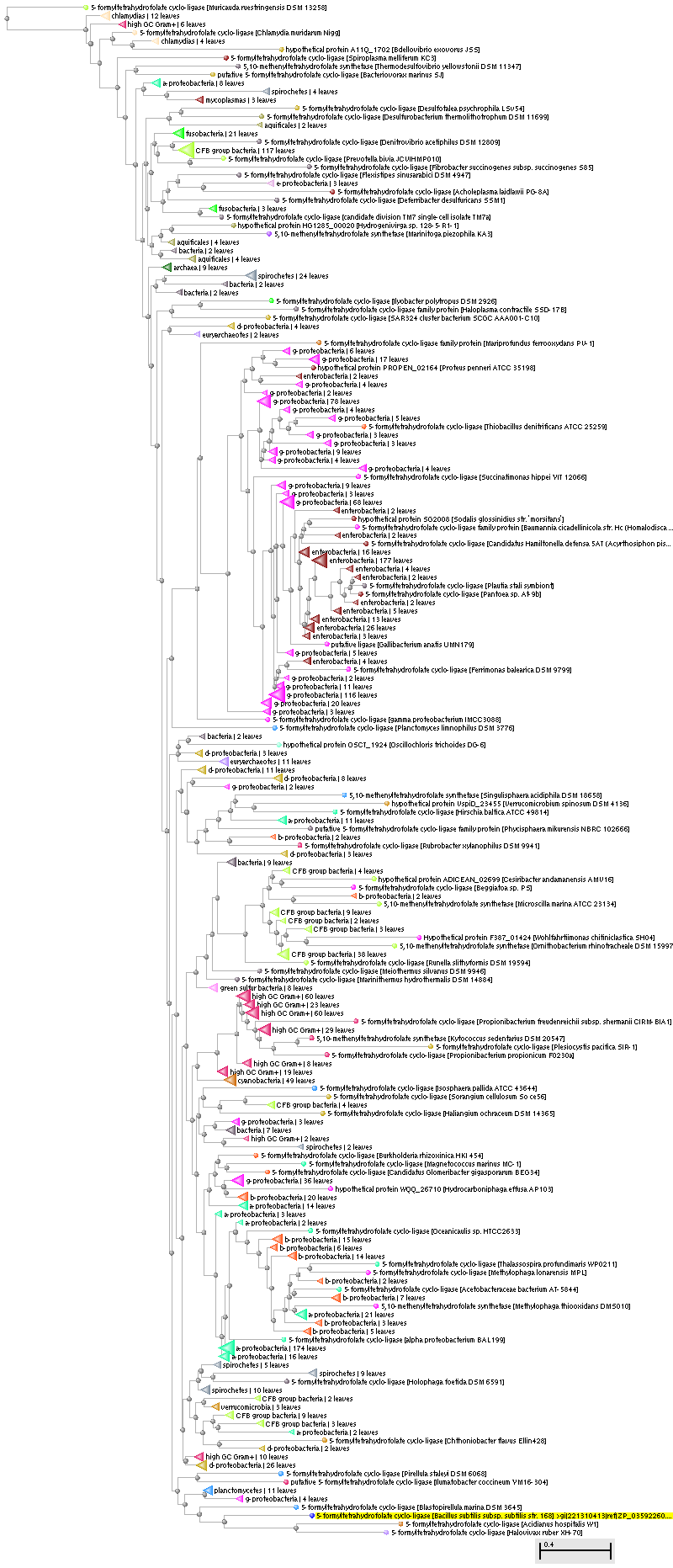
На Рис.1 приведено филогенетическое дерево для гомологов (1866) из Prokaryota.
(Увеличенное изображение при клике на картинке)

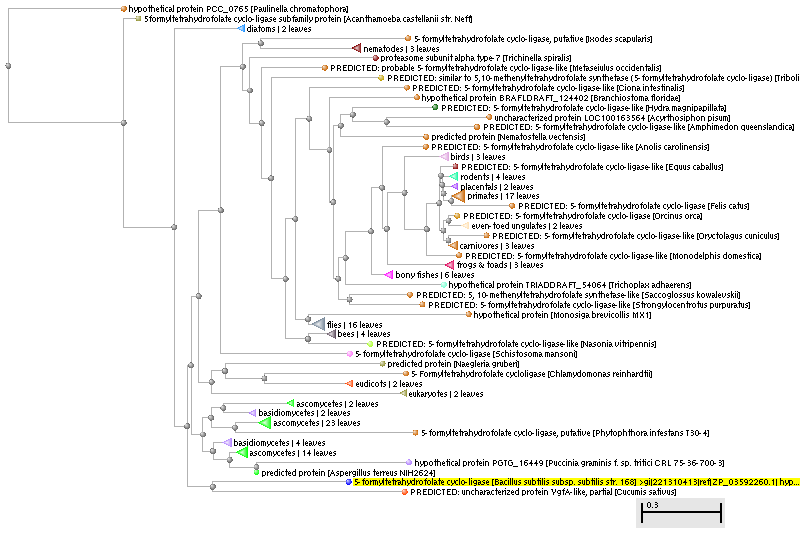
(Увеличенное изображение при клике на картинке)
В Табл.2 представлена выборка для гомологов белка YQGN_BACSU и сам белок (выделен красным цветом). Выписаны 3 домена, по которым велся поиск, в каждом из них выделены филумы/царста и белки, которые к ним принадлежат. Для каждого гомолога приведены организмы, из которых они выделены. Так для прокариот было найдено 29 белков, для эукариот - 10.
Табл. 2. Организмы из разных таксонов, в которых присутствует белок YQGN или его гомологи
| Домен | Филум/Царство | Название организма |
| Archaea | • Cenarchaeales | [Cenarchaeum symbiosum A] |
| • Euryarchaeotes | [Thermoplasmatales archaeon SCGC AB-539-N05] | |
| • Nitrosopumilales | [Candidatus Nitrosopumilus sp. AR2] | |
| • Nitrososphaerales | [Candidatus Nitrososphaera gargensis Ga9.2] | |
| • Methanomicrobiaceae | [Methanoculleus bourgensis MS2] | |
| • Methanoregulaceae | [Methanoregula formicicum SMSP] | |
| Bacteria | • Actinobacteria | [Nocardiopsis alba ATCC BAA-2165] |
| • Aquificales | [Persephonella marina EX-H1] | |
| • CFB group bacteria | [Prevotella saccharolytica F0055] | |
| • Chlamydias | [Chlamydia trachomatis A/HAR-13] | |
| • Cyanobacteria | [Leptolyngbya sp. PCC 6406] | |
| • Deferribacterales | [Denitrovibrio acetiphilus DSM 12809] | |
| • Deinococcales | [Deinococcus maricopensis DSM 21211] | |
| • Dictyoglomales | [Dictyoglomus thermophilum H-6-12] | |
| • Firmicutes (YQGN_BACSU) | [Bacillus subtilis subsp. subtilis str. 168] | |
| • Fusobacteria | [Ilyobacter polytropus DSM 2926] | |
| • GNS bacteria | [Oscillochloris trichoides DG-6] | |
| • Green sulfur bacteria | [Chlorobaculum parvum NCIB 8327] | |
| • Haloplasmatales | [Haloplasma contractile SSD-17B] | |
| • Mycoplasmas | [Ilyobacter polytropus DSM 2926] | |
| • Nitrospinales | [Nitrospina gracilis 3/211] | |
| • Nitrospirales | [Thermodesulfovibrio yellowstonii DSM 11347] | |
| • Planctomycetes | [Rhodopirellula sallentina SM41] | |
| • Proteobacteria | [Vibrio harveyi CAIM 1792] | |
| • Spirochetes | [Treponema primitia ZAS-1] | |
| • Thermotogales | [Marinitoga piezophila KA3] | |
| • Thermales | [Thermus scotoductus SA-01] | |
| • Thermodesulfobacteriales | [Thermodesulfobacterium geofontis OPF15] | |
| • Verrucomicrobia | [Methylacidiphilum infernorum V4] | |
| Eukaryotes | • Acanthamoeba | [Acanthamoeba_castellanii_str._Neff] |
| • Animals | [Homo_sapiens] [Macaca_mulatta] |
|
| • Cercozoans | [Paulinella chromatophora] | |
| • Choanoflagellates | [Monosiga brevicollis MX1] | |
| • Diatoms | [Thalassiosira pseudonana CCMP1335] | |
| • Fungi | [Saccharomyces cerevisiae S288c] | |
| • Green plants | [Cucumis sativus] | |
| • Oomycetes | [Phytophthora infestans T30-4] | |
| • Perkinsida | [Perkinsus_marinus_ATCC_50983 | |
| • Schizopyrenida | [Naegleria gruberi] |
Для удобства просмотра, таблица 2 находится также в файле.
Множественное выравнивание гомологов белка YQGN_BACSU
При помощи Jalview было визуализированно предварительное множественное выравнивание, построенное программой Muscle. Результаты представлены на Рис.3.Как видно из Рис.3, начало последовательности Acanthamoeba_castellanii_str._Neff совсем ни на что не похоже, а выравнялись только концевые блоки. Поэтому в дальнейшем исключаем данную последовательность из выравнивания.
Еще пришлось исключить последовательность Cucumis_sativus, так как гомология для белка этого организма представляется сомнительной: в начале последовательности нет большого куска, а в конце видны достаточно случайные совпадения.
Немного выбивается последовательность Denitrovibrio_acetiphilus_DSM_12809, так как в нем нет первого блока, но дальше она хорошо выравнивается.
Последовательность Thalassiosira_pseudonana_CCMP1335 имеет в начале достаточно большй "хвост" и периодические вставки между прдеполагаемые консервативными участками, что дает основание ее тоже исключить из выборки.
(Увеличенное изображение при клике на картинке)
 На рисунке представленно множественное выравнивание для предполагаемых гомологов белка YQGN_BACSU из полученной ранее выборки.
На рисунке представленно множественное выравнивание для предполагаемых гомологов белка YQGN_BACSU из полученной ранее выборки.Идентификаторы и названия организмов, из которых были выделены белки, представлены в виде блоков, покрашенных разными цветами: Археи - красным, Бактерии - зеленым, Эукариоты - синим.
Файл данного проекта в Jalview доступен по ссылке.
Обозначения:
"SECONDARY" - строка, содержащая информацию о вторичной структуре YQGN_BACSU со стандартными "заготовками" для альфа-спиралей и бета-тяжей.
"BLOCKS" - строка, содержащая буквы "B" на участках выранивания, похожих на блоки.
"LIGAND" - строка, содержащая буквы "L" под аминокислотными остатками, участвующими в связывании лиганда.
(Увеличенное изображение при клике на картинке)
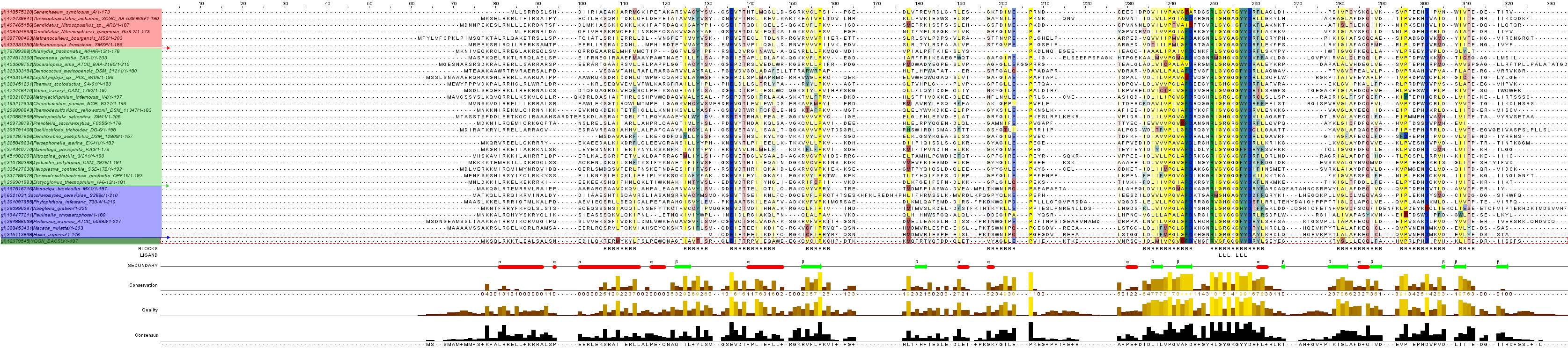 На рисунке представленно множественное выравнивание для оставшихся гомологов белка YQGN_BACSU.
На рисунке представленно множественное выравнивание для оставшихся гомологов белка YQGN_BACSU.Идентификаторы и названия организмов, из которых были выделены белки, представлены в виде блоков, покрашенных разными цветами: Археи - красным, Бактерии - зеленым, Эукариоты - синим.
Параметр color by conservation - 30%
Аминокислоты покрашены в соответсnвии с их функциями:
1. Положительно заряженные – Lys(K), Arg(R), His(H) - светло-зеленым цветом
2. Отрицательно заряженные – Asp(D), Glu(E) – синим цветом
3. Полярные – Ser(S), Thr(T), Cys(C), Met(M), Asn(N), Gln(Q) – красным цветом
4. Неполярные – Gly(G), Ala(A), Val(V), Leu(L), Ile(I), Pro(P) – желтым
5. Ароматические – Phe(F), Tyr(Y), Trp(W) – серо-зеленым
Обозначения аналогичные рис.3. Файл данного проекта в Jalview доступен по ссылке.
Как видно из рис.4., данное множественное выравнивание существенно лучше, чем на рис.3. Можно предположить, что все белки, чьи последовательности остались в данном выравнивании, действительно являются гомологами белка YQGN_BACSU.
По ссылке доступен файл в FASTA формате с гомологами белка YQGN_BACSU.
Результаты анализа множественного выравнивания гомологов белка YQGN_BACSU
Анализ 3D-структуры белка исходя из выравнивания
Сделаем так, чтобы в выравнивание остатки были покрашены в соответствии с их функциональными группами, поставим галочку в
меню Colour->By Conservation и установим порог - 30%. После этого выведем 3D структуру для белка YQGN_BACSU
(правой кнопкой по последовательности белка, а там в меню Structure->View structure for....
Теперь цвета остатков, которые видно на выравнивании, будут наложены на остатки структуры.
Весь белок представлен в виде cartoons, остатки, связывающие лиганд, выделены в виде толстого wireframe и подписаны.
(Увеличенное изображение при клике на картинке)
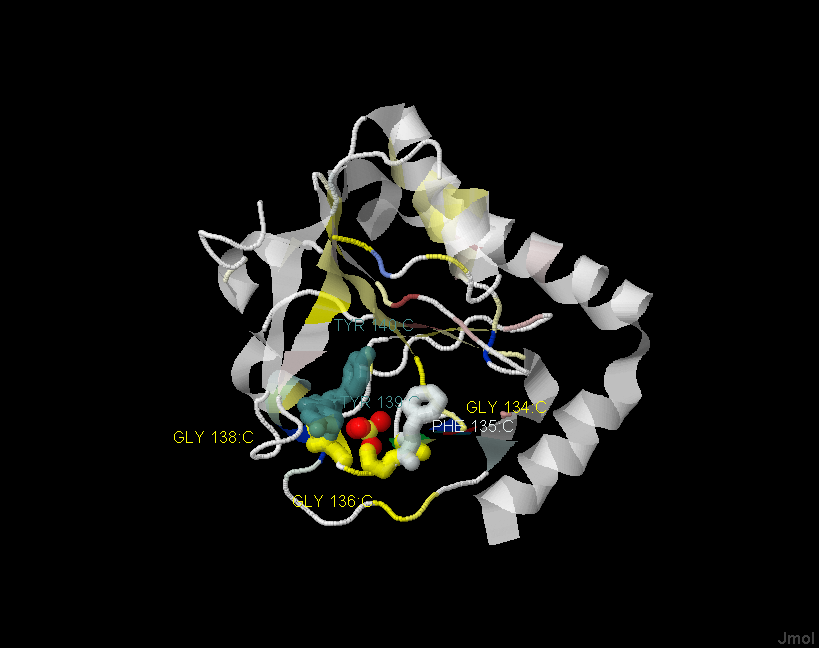 На рисунке подписаны аминокислотные остатки, которые участвуют в связывание с лигандом SO4:
На рисунке подписаны аминокислотные остатки, которые участвуют в связывание с лигандом SO4:GLY 134, PHE 135, GLY 136, GLY 138, TYR 139, TYR 140
Выделены отдельно и сами связывающие аминокислотные остатки.
Сам лиганд выделен "шариками" (атомы кислорода - красные, серы - желтые).
Консервативность выравнивания в целом можно оценить как среднее, так как идеально (80-100%) идентичных участков достаточно мало (14 и все они находятся
ближе к концу, начало белковых последовательностей разнообразно, как по длине, так и по аминокислотным остаткам).
Некоторые консервативные участки совпадают по положению с элементаим вторичной структуры: альфа спиралями, бета-листами.
Вторичная структура фунцкционально важна, поэтому происходит консервативность.
Качественно на "колонки-гэпов" (то есть колонки, в которых стоит знак гэпа у всех последовательностей
кроме пары-тройки) элементов вторичной структуры по сравнению с другими участками белка почти не приходится. Колонки
-гэпов разбивают только одну альфа-спираль (с 18 по 33 остаток) из-за последовательности белка из Rhodopirellula salentina.
"Блоки", выделенные мной соответствуют участкам вторичной структуры не сильно. Блоки были выбраны по следующим критериям: видимая продолжительная
идентичность в последовательностях при colour by conservation 20%.
На основании данного множественного выравнивания можно сказать, что все найденные белки способны связывать лиганд, так как участок
связывания очень консервативен.
GLY 134(G) - абсолютно консервативен для всех последовательностей
GLY 136(G) и GLY 138(G)- абсолютно консервативны для всех последовательностей
PHE 135(F) - на его месте чаще всего встречается Y, так же могут встречаться: R, H. Замена на M происходит лишь в одном белке из Vibrio_harveyi
TYR 139(Y) - на F, W; H - в одной
TYR 140(Y) - в двух последовательностях заменен на F:
*Приложения:
- Файл c последовательностями белков гомологов YQGN_BACSU из Prokaryota
- Файл c последовательностями белков гомологов YQGN_BACSU из Prokaryota
Список литературы:
© Nuzhdina Ekaterina, 2012

