PSI-BLAST
Составление семейства гомологов с помощью PSI-BLAST
Зайдем на сайт BLAST, выберем "protein blast". В качестве объекта исследования
используем белок с идентификатором Q246C0. Будем искать по базе данных "refseq_protein".
На странице с результатом два блока - хорошие находки (верхний) и плохие находки (нижний). А также два столбца для галочек, левый и правый.
В правом отмечаются те последовательности, по которым будет выполнена очередная итерация поиска PSI-BLAST: построены PSSM
(позиционные матрицы весов) и выполнен поиск.
При выборе последовательностей для очередной итерации будем обращать внимание на:
1) E-value находок, точнее на ступеньку в значениях E-value (резкий переход более, чем на 6 порядков).
2) Таксономию находок: если все, кроме одной, принадлежат роду Bacillus, а одна - из эукариот, то ее стоит исключить.
После первого полученного результата увидим, что все "хорошие" находки можно использовать для дальнейшей итерации. Найдем строчку
"Run PSI-Blast iteration ... with max" (на месте ... будет стоять номер итерации) => нажмем "Go".
И так несколько раз пока у нас не получаться сходящиеся результаты для худшей находки выше порога (0.005) и для лучшей находки
ниже порога двух подряд идущих итераций. Основные параметры каждой интерации до сходящегося результата представлены в табл.1.
Табл.1. Основные параметры итераций.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 58 | XP_002020128.1 | 0.003 | XP_004374902.1 | 0.010 |
| 2 | 72 | XP_002648497.1 | 0.001 | XP_003223763.1 | 0.038 |
| 3 | 72 | NP_001171930.1 | 4e-05 | XP_003223763.1 | 0.035 |
| 4 | 72 | XP_003387958.1 | 2e-04 | XP_003223763.1 | 0.062 |
| 5 | 72 | XP_003387958.1 | 6e-05 | XP_002635539.1 | 0.017 |
| 6 | 72 | XP_003387958.1 | 7e-05 | XP_002635539.1 | 0.017 |
| 7 | 72 | XP_003387958.1 | 7e-05 | XP_002635539.1 | 0.017 |
После выполнения последней итерации в левом столбце для галочек отметим все найденные последовательности после окончательной итерации. Нажмем кнопку Multiple alignment сверху блока и получим выравнивание последовательностей с помощью программы "Cobalt". В вверху страницы найдем андпись "Download"=> "Fasta plus gaps" и сохраним. По данной ссылке доступен файл в FASTA-формате с выравниванием последовательностей, найденных PSI-BLAST. Теперь визуализируем выравнивание при помощи программы JalView. Результаты можно увидить на рис.1.
(Увеличенное изображение при клике на картинке)
Выравнивание последовательностей-гомологов белка Q246C0, найденных с помощью PSI-BLAST. Визуализация выполнена с помощью JalView.
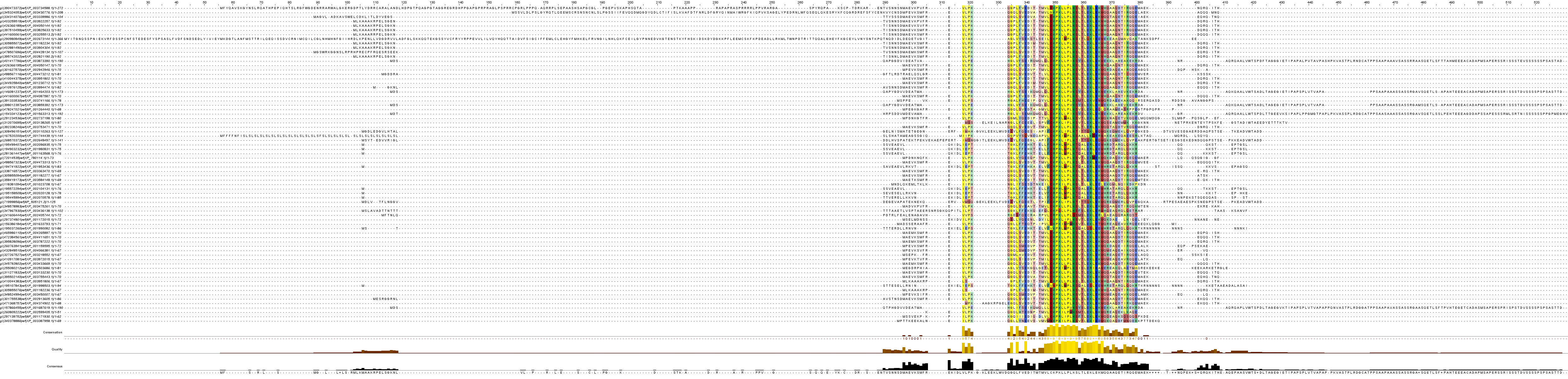
Из рис.1. очевидно, что найденные последовательности белков являются гомологами Q246C0, так как они они хорошо выравниваются.

