EMBOSS
Программа getorf пакета EMBOSS
Для изучения программы getorf пакета EMBOSS была взята запись банка EMBL с ID D89965. Полная запись была получена с помощью команды:
Сама запись представлена в файле d89965.entret.
Получим набор трансляций всех открытых рамок данной последовательности, которые определены при использовании стандартного кода
и одновременно удовлетворяют следующим условиям:
Используем код:
Где -table 0 - стандартный генетический код, -minsize 90 - минимальная длина orf 90 нуклеотидов,
-find 1 - нужный тип поиска: orf начинается со старт-кодона и заканчивается стоп-кодоном.
В результате был получен следующий файл d89965_orfs.fasta:
>D89965_1 [66 - 155] Rattus norvegicus mRNA for RSS, complete cds. MQFHPRLPAVLQVCAACDRYASLLPAQRRL >D89965_2 [56 - 169] Rattus norvegicus mRNA for RSS, complete cds. MISDAVSSATASSASSLRSMRSVRQSFASSTAALTRWP >D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGA >D89965_4 [218 - 3] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MLLRCSNCLNVNWKCIRAIWSKPPLSWQKTGVPIACCANLKHCWQSRMKLHRLSSPVTVT WCSQKTILLLSA >D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Определим, какая из найденных открытых рамок соответствует (полностью или частично) приведённой в поле FT кодирующей
последовательности (CDS).
Кодирующая последовательность приведена ниже:
MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHY GIAQRGLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA
Этой последовательности полностью соответствует третья найденная ORF (D89965_3).
Теперь создадим файл с последовательностью записи Swiss-Prot, на которую ссылается данная запись EMBL.
Теперь выясним, какой из полученных открытых рамок соответствует эта последовательность. Нетрудно заметить, что последовательность соответствует пятой ORF D89965_5 (можно скопировать небольшой кусок из файла sw_prot.fasta и найти его при помощи ctr+F в файле d89965_orfs.fasta). Для подтверждения соответствия приведем выравнивание ORF с последовательностью (для удобства полное выравнивание со всеми ORF приведено в файле - algn_all.fasta, а ниже представлено только выравнивание с D89965_5):
########################################
# Program: needle
# Rundate: Sat 14 Dec 2013 20:02:26
# Commandline: needle
# [-asequence] sw_prot.fasta
# [-bsequence] d89965_orfs.fasta
# -outfile algn.fasta
# Align_format: srspair
# Report_file: algn.fasta
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: HSLV_ECOLI
# 2: D89965_5
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 176
# Identity: 98/176 (55.7%)
# Similarity: 98/176 (55.7%)
# Gaps: 78/176 (44.3%)
# Score: 490.0
#
#
#=======================================
HSLV_ECOLI 1 MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGG 50
|||||||||||||||||||||||
D89965_5 1 ---------------------------MKGNVKKVRRLYNDKVIAGFAGG 23
HSLV_ECOLI 51 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 100
||||||||||||||||||||||||||||||||||||||||||||||||||
D89965_5 24 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 73
HSLV_ECOLI 101 ETASLIITGNGDVVQPENDLIAIGSGGPYAQAAARALLENTELSAREIAE 150
|||||||||||||||||||||||||
D89965_5 74 ETASLIITGNGDVVQPENDLIAIGS------------------------- 98
HSLV_ECOLI 151 KALDIAGDICIYTNHFHTIEELSYKA 176
D89965_5 98 -------------------------- 98
#---------------------------------------
#---------------------------------------
Как видно из результатов, найденный белок отностся к E.coli, в то время как данные ORF относятся к Rattus norvegicus. Это можно объяснить тем, что в процессе пробоподготовки к секвенированию скорее всего произошло загрязнеие образца посторонними мРНК (так секвенировались мРНК эпителия желудка, то вероятность попадания кишечной палочке довольно велика). А потом в автоматическом режиме аннотировали эту запись.
Файлы-списки
Теперь скачаем в файл adh.fasta в fasta-формате все доступные в Swissprot последовательности алкогольдегидрогеназ (их идентификаторы описываются выражением adh*_*) при помощи команды:
Получим файл с универсальными адресами (USA) этих последовательностей.
То, что получится – пример файла-списка ("листфайла").
Используем программу infoseq с параметрами -only и -usa:
Получим из этого файла-списка другой, меньший, с адресами только тех последовательностей, которые взяты из организмов:
Для этого используем программу grep с параметром -f, чтобы подать ей на вход список слов для поиска:
Получили файл с адресами последовательностей из нужных организмов.
На основе этого файла-списка получим fasta-файл с последовательностями дегидрогеназ наших организмов, используя программу seqret:
Итого полученные файлы:
EnsEMBL
Портал EnsEMBL предназначен для визуализации известной информации о геномах человека и животных.
Получим последовательность всего гена, кодирующего белок NINJ2_HUMAN. При помощи сервера srs мы нашли какая мРНК соответствует
нашему белку (AF205633).
>AF205633 AF205633.1 Homo sapiens ninjurin2 (NINJ2) mRNA, complete cds. cccacgcagtctgttcccggcacccggtgcgtgtgaagggacttgagggcagcgagatgg aatcagcaagagaaaacatcgaccttcaacctggaagctccgaccccaggagccagccca tcaacctgaaccattacgccaccaagaagagcgtggcggagagcatgctggacgtggccc tgttcatgtccaacgccatgcggctgaaggcggtgctggagcagggaccatcctctcatt actacaccaccctggtcaccctcatcagcctctctctgctcctgcaggtggtcatcggtg tcctgctcgtggtcattgcacggctgaacctgaatgaggtagaaaagcagtggcgactca accagctcaacaacgcagccaccatcttggtcttcttcactgtggtcatcaatgttttca ttacagccttcggggcacataaaacagggttcctggctgccagggcctcaaggaatcctc tctgaatgcagcctgggacccaggttctgggcctggaacttctgcctccttcctccgtga tctgccaggctcgtgggcactttccacagcccaggagagcttctgaaaggacagtatagc tgcccttgctccctacccacagcacctgagttaaaaagtgatttttatgttattggtcta agggacttccatcttggtctgaagtcctgagctcagacgcaggtactgccagccatacct tcctggtagcatctgctggacctaagtaaggcatgtctgtctaaggccaagtctgcccgg cttaaggatgctggttctgactctaccccactgcttccttctgctccaggcctcaatttt cccttcttgtaaaatggaatctatatctataaaggtttcttcaaatccaaaaaaaaaaaa aaaaaaa
При помощи портала EnsEMBL найдем информацию о гене человека NINJ2_HUMAN, который мы выбрали ранее (см. семестр 3--> online BLAST). Для этого в строку на главной странице "for" впишем NINJ2_HUMAN. Перейдем по ссылке, найденной после ввода. На странице мы найдем основную информацию о гене, кодирующем белок.
И print screen сайта видно, что у гена есть 4 транскрипта (т.е. варианта трансляции), 4 экзона, длина транслируюнмой области 188 остатка.
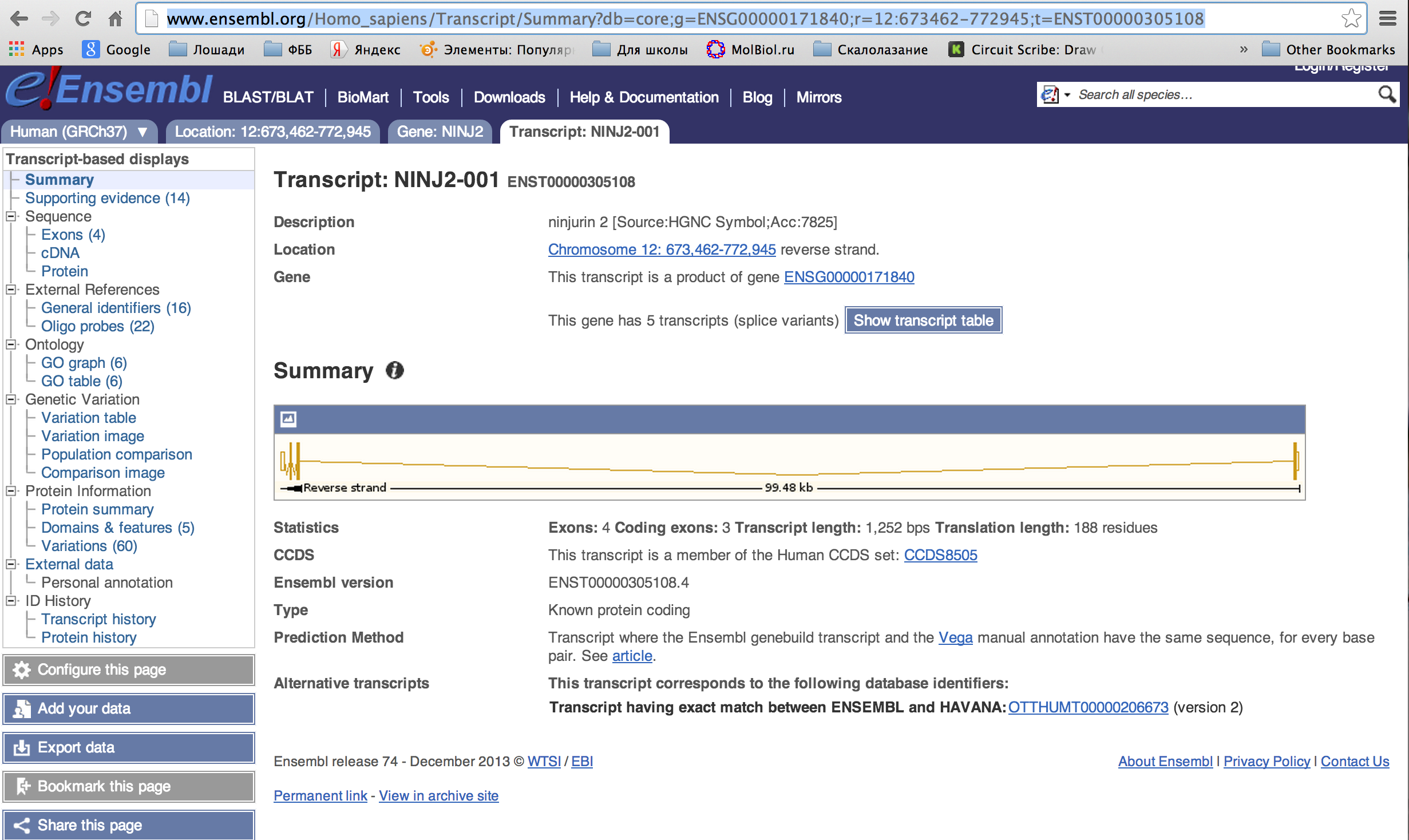
Далее определим при помощи сервиса BLAST/BLAT (гиперссылка вверху заглавной страницы) местоположение гена в геноме человека.
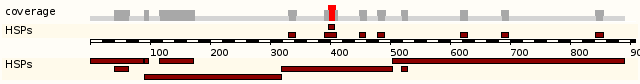
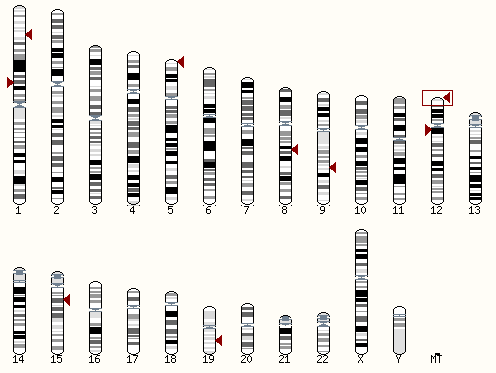
Результаты представлены на рис.2.
Из рисунка видно, что ген NINJ2_HUMAN локализован в 12 хромосоме (местоположение выделено рамкой).
На странице результата в самом низу дана информация о найденнных совпадениях гена в геноме человека.
Таблица 1. Лучший хит, найденный при помощи BLAT
Query |
Chromosome |
Stats |
||||||||
Start |
End |
Ori |
Name |
Start |
End |
Ori |
Score |
E-val |
%ID |
Length |
504 |
889 |
- |
Chr:12 |
673462 |
673847 |
+ |
1939 |
0.0e+00 |
100.00 |
386 |
Осталось не очень понятным, что означает пункт %ID, так как даже у хитов, в которых совпало всего 7-13 нуклеотидов %ID тоже равно 100.00.
Далее пройдем по гиперссылке Contig view, обозначенной "[C]" левее первой находки
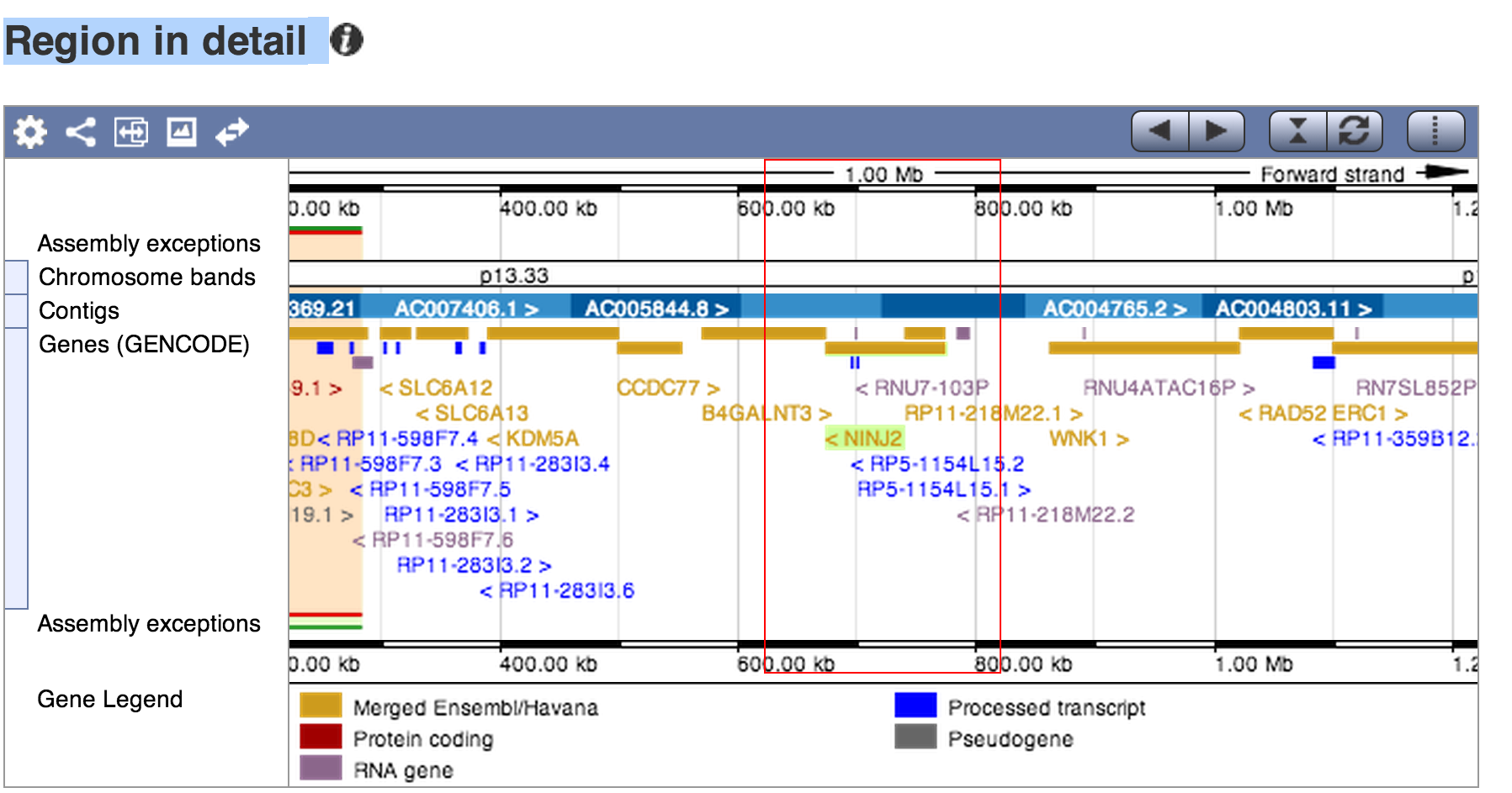
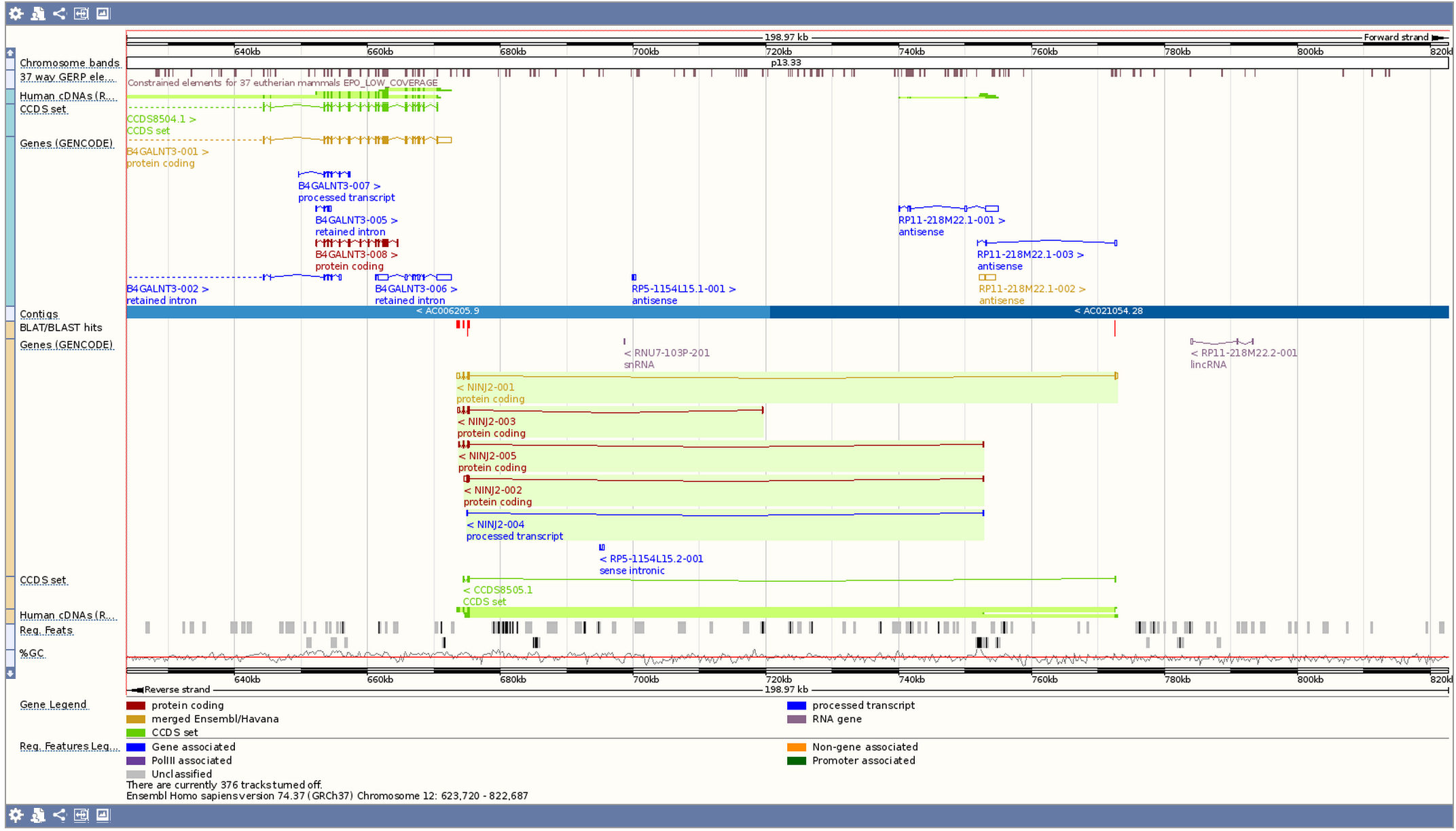
(в таблице находок "Alignment Summary" внизу страницы). Откроется страница "Region in detail", посвящённая участку человеческого генома.
На ней есть несколько полностью интерактивных картинки: мы можем рассмотреть детальное расположение хита в хромосоме
(Рис. 3), в контиге (Рис. 4) и детальное окружение хита (Рис. 5).
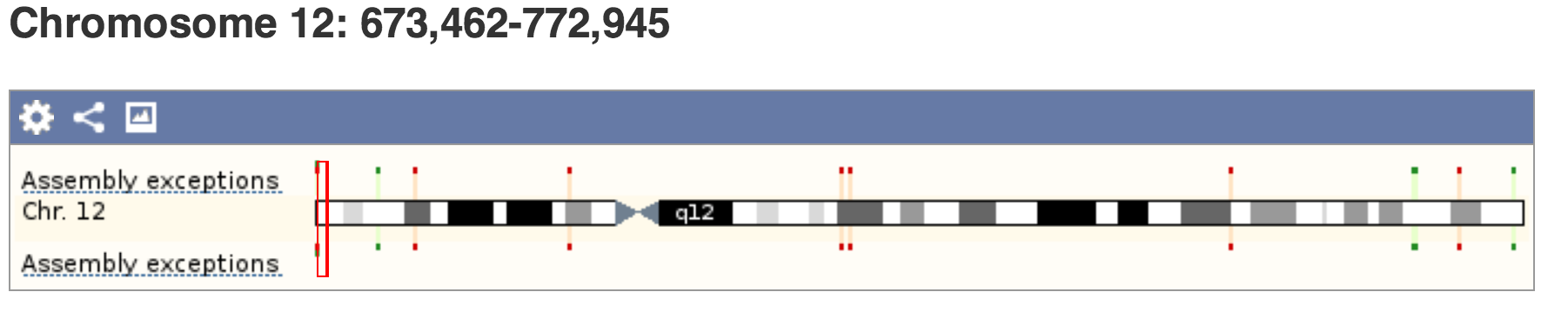
К сожалению, непосредственный эспорт изображений хита в хромосоме через сайт убирает рамочку, показывающую его местоположение.
Поэтому следующее изображение получено просто print screen.
Красной рамкой выделен участок 673 462-772 945, который кодирует белок NINJ2_HUMAN.
На странице можно настроить разрешение картинки, приблизить или отдалить, прокрутить или экспортировать ее как файл
в широком выборе форматов.
Сам сервис EnsEMBL довольно информативен.