Геномные браузеры
UCSC
Найдем выбранный нами ранее белок человека NINJ2_HUMAN (см. 3 семестр--> Онлайн BLAST) в UCSC браузере.
Таблица 1. Результаты поиска в UCSC браузере по белку человека NINJ2_HUMAN
Короткое и полное имя гена |
NINJ2, Homo sapiens ninjurin 2 |
Закодирован на цепи |
Обратной (-) |
В какой хромосоме находится |
Хромосома 12 |
К каким плечу и полосе принадлежит участок |
chr12:p13.33 |
Координаты гена в последовательности хромосомы |
673 462 - 772 755 |
Сколько альтернативных продуктов закодировано в гене |
4 |
Число экзонов и длина аминокислотной последовательности |
2, 172 |
Распишем по-подробней, как мы собственно это все получили. Сначала переходим на сайт UCSC genome browser. Под надписью "Human (Homo sapiens) Genome Browser Gateway" находится поле "search term", в него вписываем идентификатор нашего белка NINJ2_HUMAN, информацию о котором мы хотим найти. Нажимаем "submit". Получаем выдачу браузера, представленную на рис.1.
(для увеличение кликните на картинку)
В красной рамке находится информация о том, в какой хромосоме находится ген (chr), кодирующий белок, и его координаты в последовательности хромосомы (после ":").
В фиолетовой рамке находится информация о том, к какому плечу и какой полосе принадлежит участок.
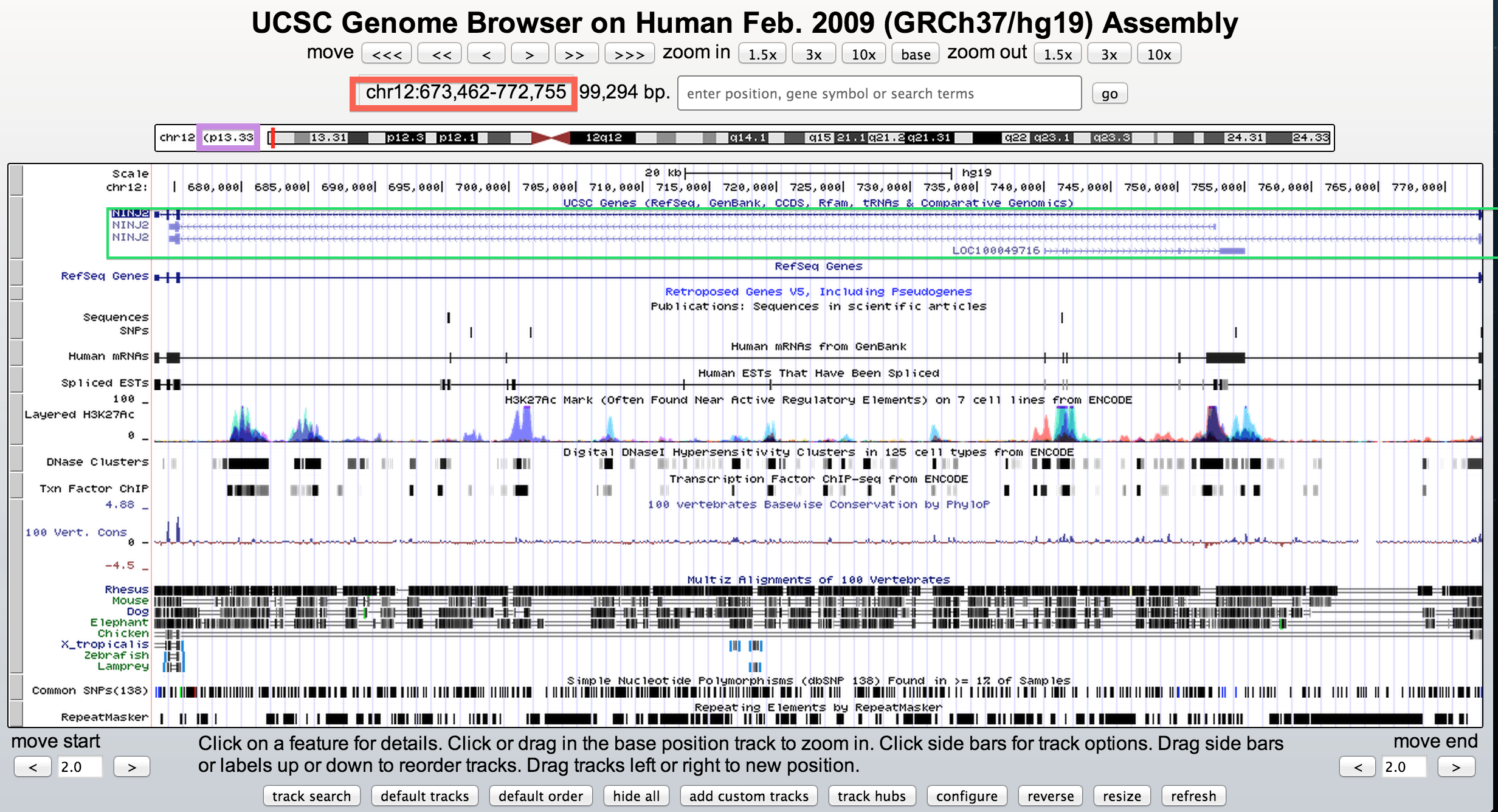
Если мы кликнем по области, обозначенной на рис.1 зеленой рамочкой, то попадем на страницу (рис.2) с описанием гена, кодирующего белок NINJ2_HUMAN. Там мы сможем найти информацию о коротком и полном имени гена, на какой цепи он закодирован; сколько альтернативных продуктов закодированно в гене - для каждого из них число экзонов и длину аминокислотной последовательности.
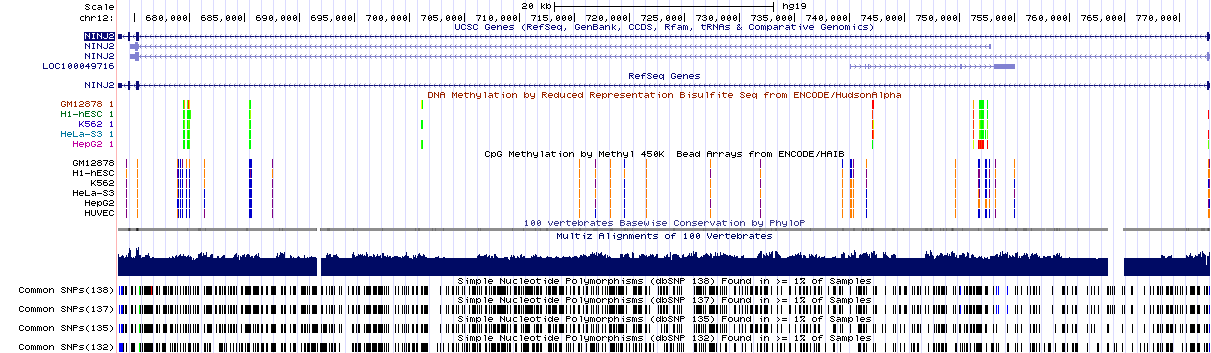
Теперь сохраним картинку окрестности гена из Genome Browser c треками (рис.3):
гены USCS и RefSeq (USCS Genes и RefSeq Genes),
метилирование ДНК (ENC_DNA_Methylation),
консервативность последовательности среди млекопитающих (Conservation),
полиморфизмы (Common SNPs)
Для этого пролистаем страницу браузера чуть ниже. Для полей USCS Genes, RefSeq Genes,
ENC_DNA_Methylation, Conservation, Common SNPs зададим параметры изображения (dense, pack, full или squish), а для всех
остальных треков укажем hide. Чтобы сохранить картинку, нажмаем правой кнопкой на трек и выбераем пункт "View image".
(для увеличение кликните на картинку)
Параметры изображения, использованные для получения картинки:
для USCS Genes, RefSeq Genes - full,
для ENC_DNA_Methylation - show,
для Conservation - squish,
для Common SNPs - dense.
Ensembl
Найдем заданный ген в поиске Ensembl
(ссылка
на найденный ген). В меню слева выберем Comparative Genomics - Genomic alignments.
В выпадающем списке "Alignment" выберем выравнивание с шимпанзе. Сохраним полученное выравнивание (export data - слева).
Выравнивание в формате CLUSTAL доступно по
ссылке.
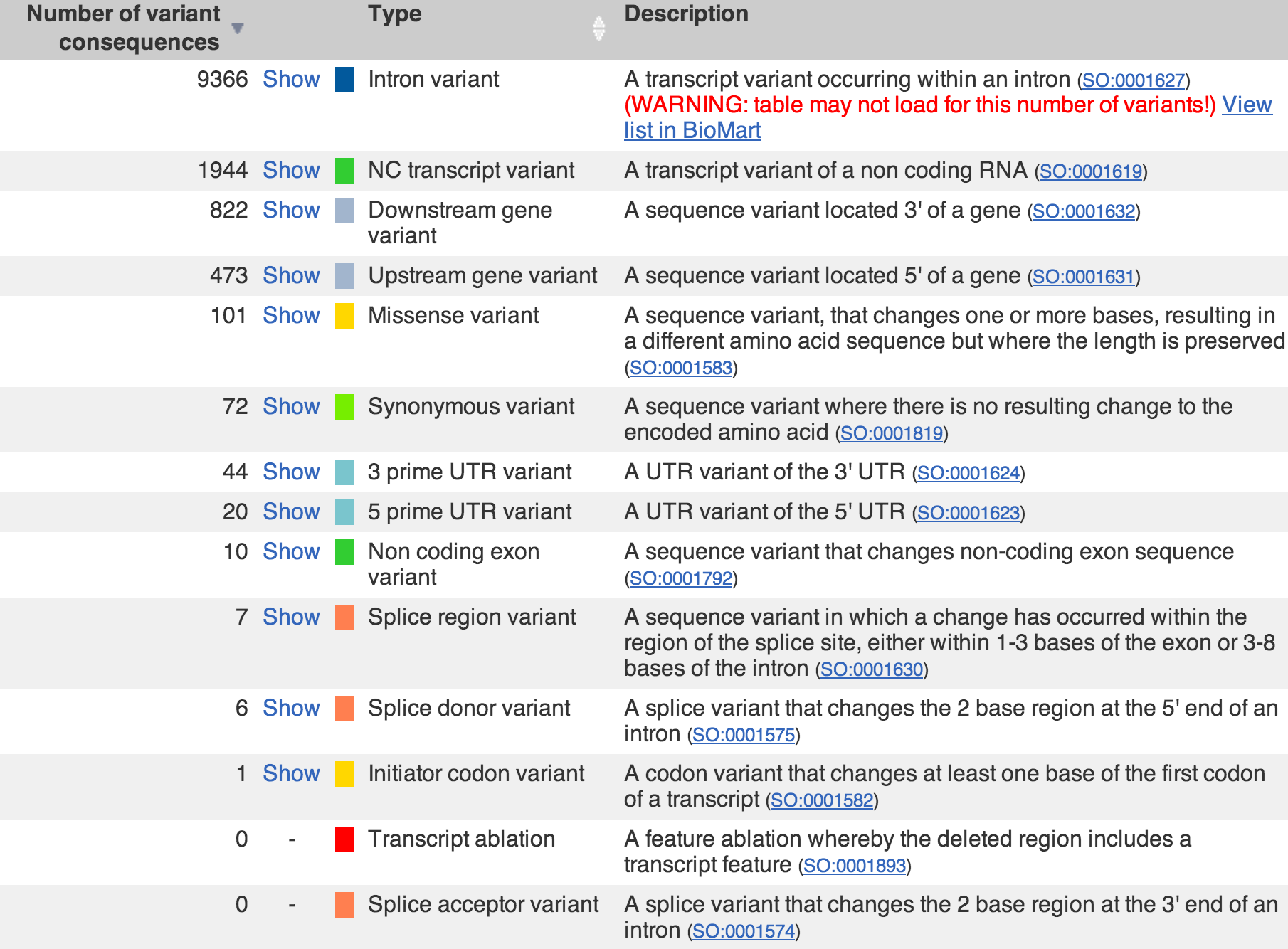
Узнаем число полиморфизмов, выбрав в меню слева Genetic Variation - Vatiation table (рис.4).
Используем команду distmat для подсчета числа различий между геном NINJ2 человека с гомологичным геном шимпанзе. В результате получаем матрицу, где для каждой пары последовательностей показано эволюционное "расстояние" в количестве замен на 100 пар оснований.