Алгоритмы реконструкции деревьев. Укоренение и бутстреп
Укоренение в среднюю точку
Укореним в среднюю точку дерево, построенное при выполнении предыдущего занятия методом Neigbour Joining Tree Using
BLOSUM62 в JalView (рис.2).
Алгоритм укороенения в среднюю точку - находим на дереве самый длинный путь от листа к листу
и за корень принимаем середину этого пути.
К сожалению, программа MEGA по не совсем понятным причинам отказывается переукоренять деревья, построенные JalView.
Поэтому воспользуемся программой retree пакета PHYLIP:
- Скопируем файл с деревом в Newick-формате (.nwk) в файл с именем intree
- Запустим программу retree
Мы увидим окно, в котором, для начала, мы должны проверить правильность параметров под буквами. В нашем случае все верно, поэтому мы печатаем 'Y' и нажиамем Enter. Если же надо что-то изменить - вводим букву той строки, которую хотим поменять.
После того, как мы ввели 'Y' программа покажет нам изображение дерева (пока без изменений) и строчку с вопросом 'NEXT?'.
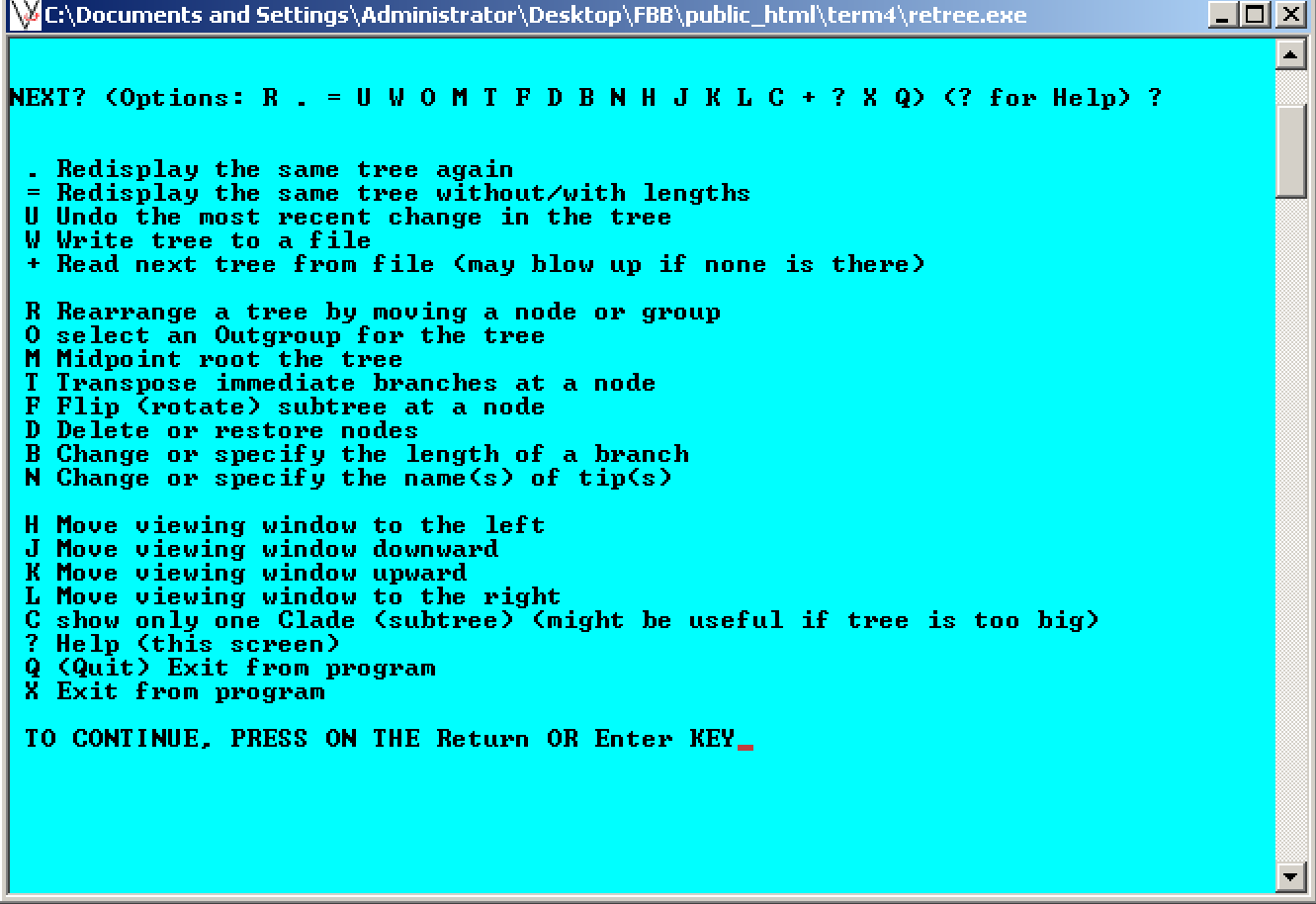
- Что бы получить информацию о том, что значат все эти буквы введем '?'.
Появившееся окно представлено на рис.1.
Нужное нам действие — укоренение в среднюю точку ("Midpoint root the tree"), которое находится под буквой 'M'. Так же нам пригодится возможность записи дерева в файл ("Write tree to a file") под буквой 'W' (дерево запишется в файл под названием "outtree").
(Увеличенное изображение при клике на картинке)
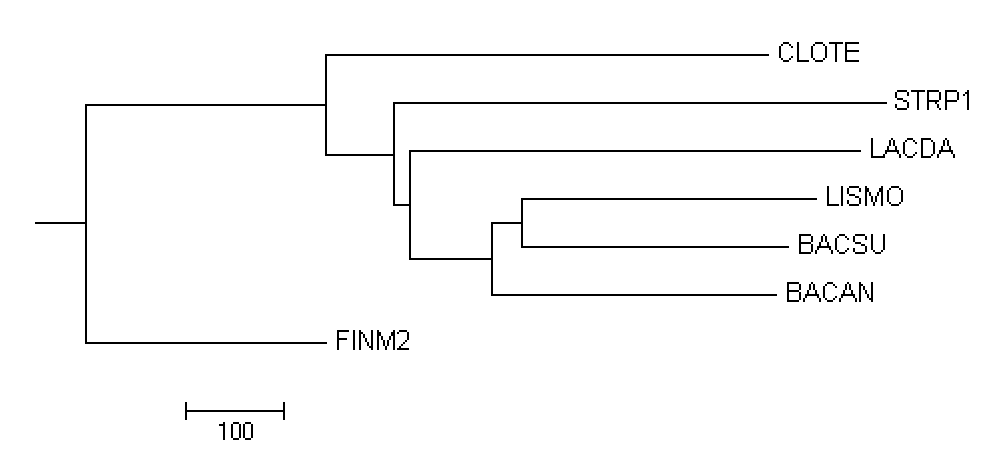
Укоренение произошло в ветвь {FINM2} против {BACAN, BACSU, LISMO, LACDA, STRP1, CLOTE}
(Увеличенное изображение при клике на картинке)
(Увеличенное изображение при клике на картинке)
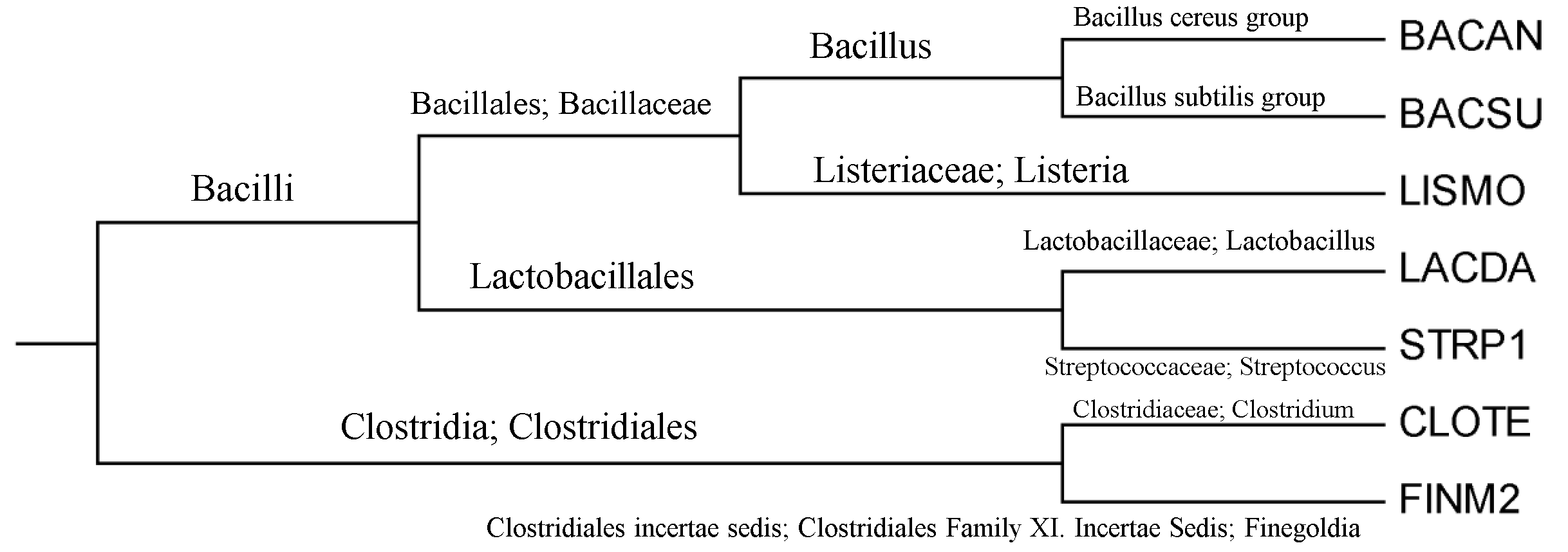
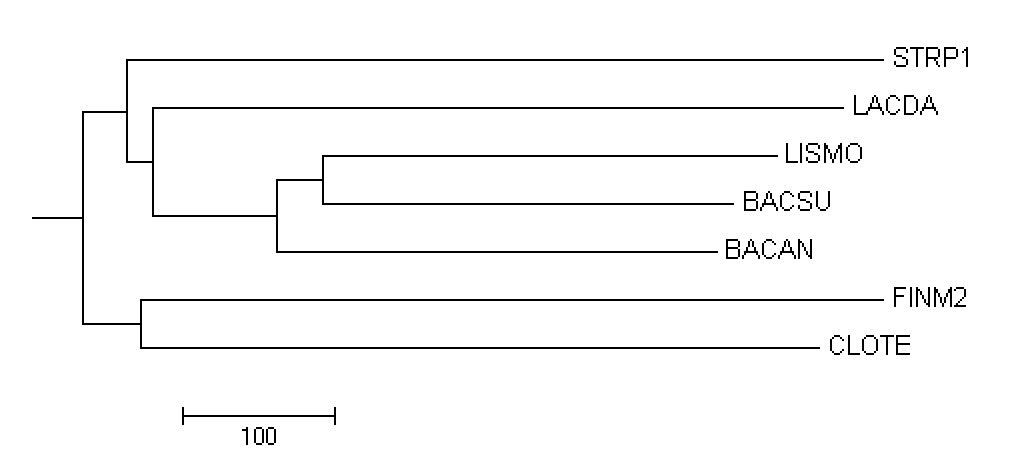
Укоренение можно считать правильным, так как оно соотносится с правильным корнем. Но вот само дерево сильно отличается по ветвям от представленного на рис.4.
Использование внешней группы
Метод Maximum Parsimony определяет оптимальное дерево так, чтобы свести к минимуму количество мутаций. Расстояний между листьями нет, поэтому нельзя выбрать самый длинный путь и провести укоренение в среднюю точку. Но мы можем воспользоваться укоренением с помощью внешней группы.
Реконструируируем методом максимальной экономии укоренённое дерево отобранных нами бактерий, используя в качестве внешней группы белок того же семейства из кишечной палочки (Escherichia coli, ECOLI).
Результат импортируем в программу MEGA. После реконструкции дерева нужно в меню Subtree выбрать Root и указать в качестве корня ветвь, ведущую к ECOLI. Наконец, чтобы получить изображение укоренённого дерева без ECOLI, нужно воспользовавться кнопкой "Show Subtree Separately" (изображение голубой лупы на фоне дерева на левой панели окна MEGA)
(Увеличенное изображение при клике на картинке)
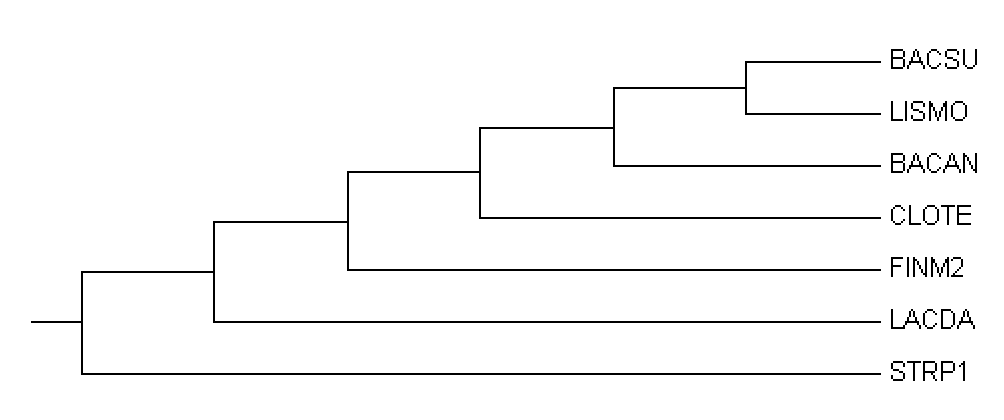
Укоренение произошло в ветвь {STRP1} против {BACAN, BACSU, LISMO, LACDA, FINM2, CLOTE}. Такое укоренения сильно отличается по сравнению с правильным деревом ({FINM2, CLOTE} {BACAN, BACSU, LISMO, LACDA, STRP1}), представленным на рисунке 4. Так же мы видим большие различия в самих ветвях. Из этого можно сделать вывод, то метод с использованием внешней группы не очень удачен для построения дерева, по крайне мере для данной выборки.
Бутстрэп
Проведем бутстрэп-анализ филогении наших белков, используя один из методов, доступных из программы MEGA.
Импортируем в MEGA файл с выравнеными последовательностями. Далее выберем в меню "Phylogeny" пункт
"Construct/Test Neighbour-Joining tree". В появившемся окошке в строке "Test of Phylogeny" выберем "Bootstrap method", а
в строке "No. of Bootstrap Replication" выберем число реплик равное 100 и нажмем "Compute".
(Увеличенное изображение при клике на картинке)
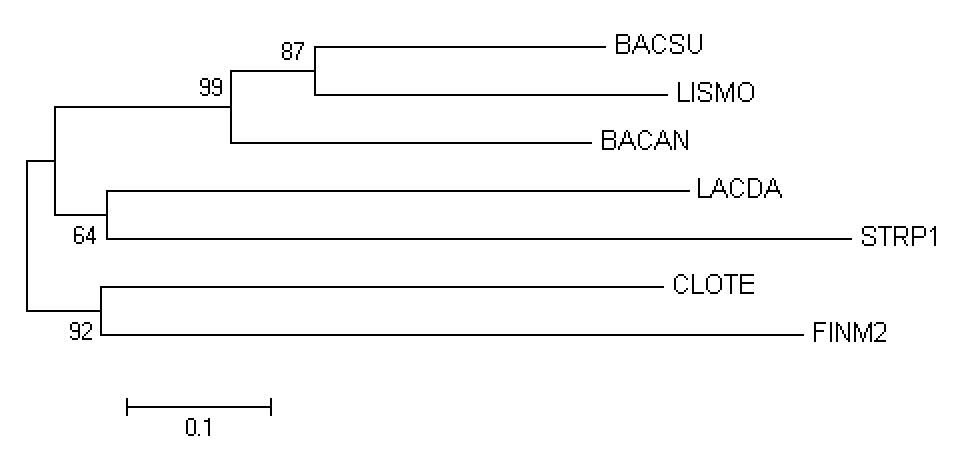
* деревья, названные "Original tree" и "Bootstrap consensus tree" не отличаются.
Как видно из рис.5 неправильные ветви не имеют меньшую поддержку, чем правильные. Полученное дерево аналогично
правильному, кроме ветви {BACSU,LISMO}, вместо {BACSU,BACAN}.