Реконструкция деревьев по нуклеотидным последовательностям.
Деревья, содержащие паралоги.
Построение дерева по нуклеотидным последовательностям
Последовательности были выравнены методом MUSCLE (MUltiple Sequence Comparison by Log- Expectation)
Методом реконструкции Minimum evolution в программе MEGA построим филогенетическое дерево.
Таблица 1.
Название |
Мнемоника |
AC записи EMBL |
Координаты 16S rRNA в записи |
Bacillus anthracis |
BACAN |
AE016879 |
9335..10841 |
Bacillus subtilis |
BACSU |
D26185 |
73411..74963 |
Clostridium tetani |
CLOTE |
AE015927 |
complement(8715..10223) |
Finegoldia magna |
FINM2 |
AP008971 |
197837..199361 |
Lactobacillus delbrueckii |
LACDA |
CR954253 |
45160..46720 |
Listeria monocytogenes |
LISMO |
AL591974 |
37466..39020 |
Streptococcus pyogenes |
STRP1 |
AE004092 |
17170..18504 |
(Увеличенное изображение при клике на картинке)
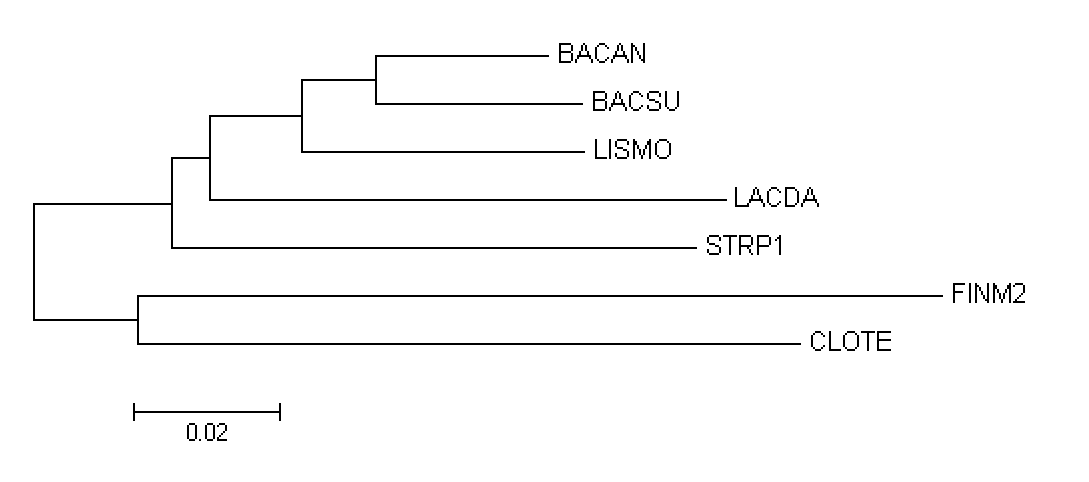
(Увеличенное изображение при клике на картинке)
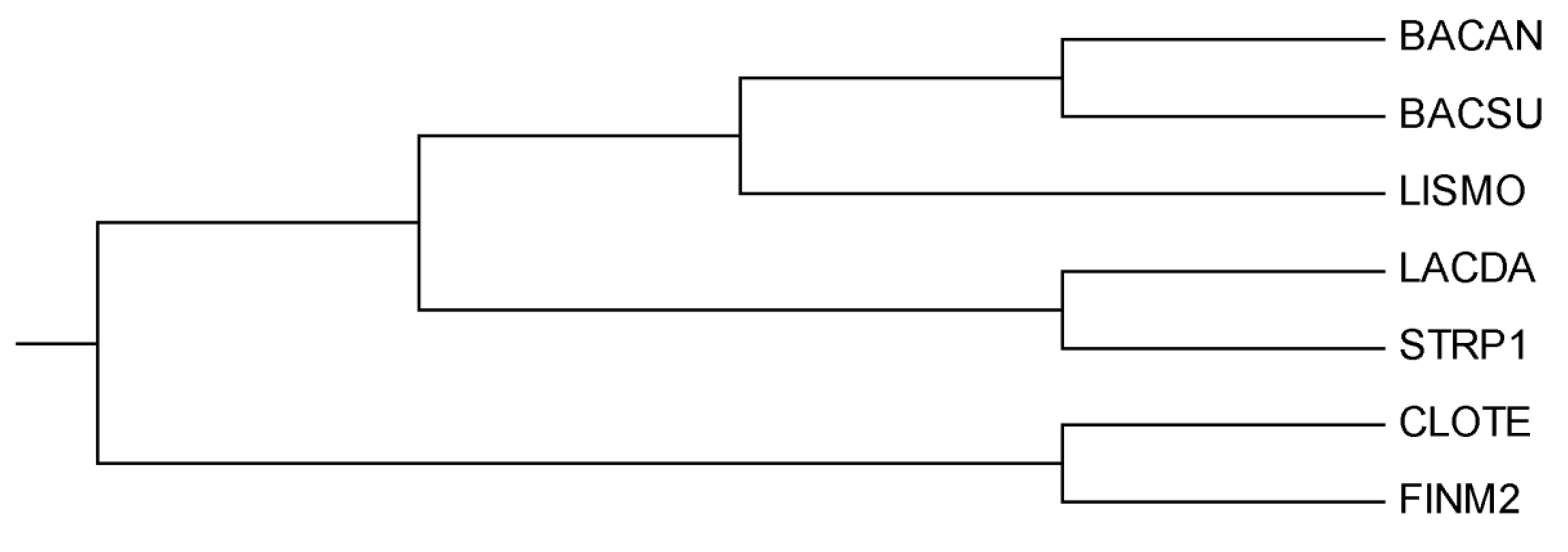
Теперь проведем анализ полученного дерева (рис.1) с правильным деревом (рис.2):
мы видим, что оба дерева практически совпадают. Отличие есть только в одной ветке STRP1.
По сравнению с деревьями, построенными по белкам качество реконструкции гораздо выше.
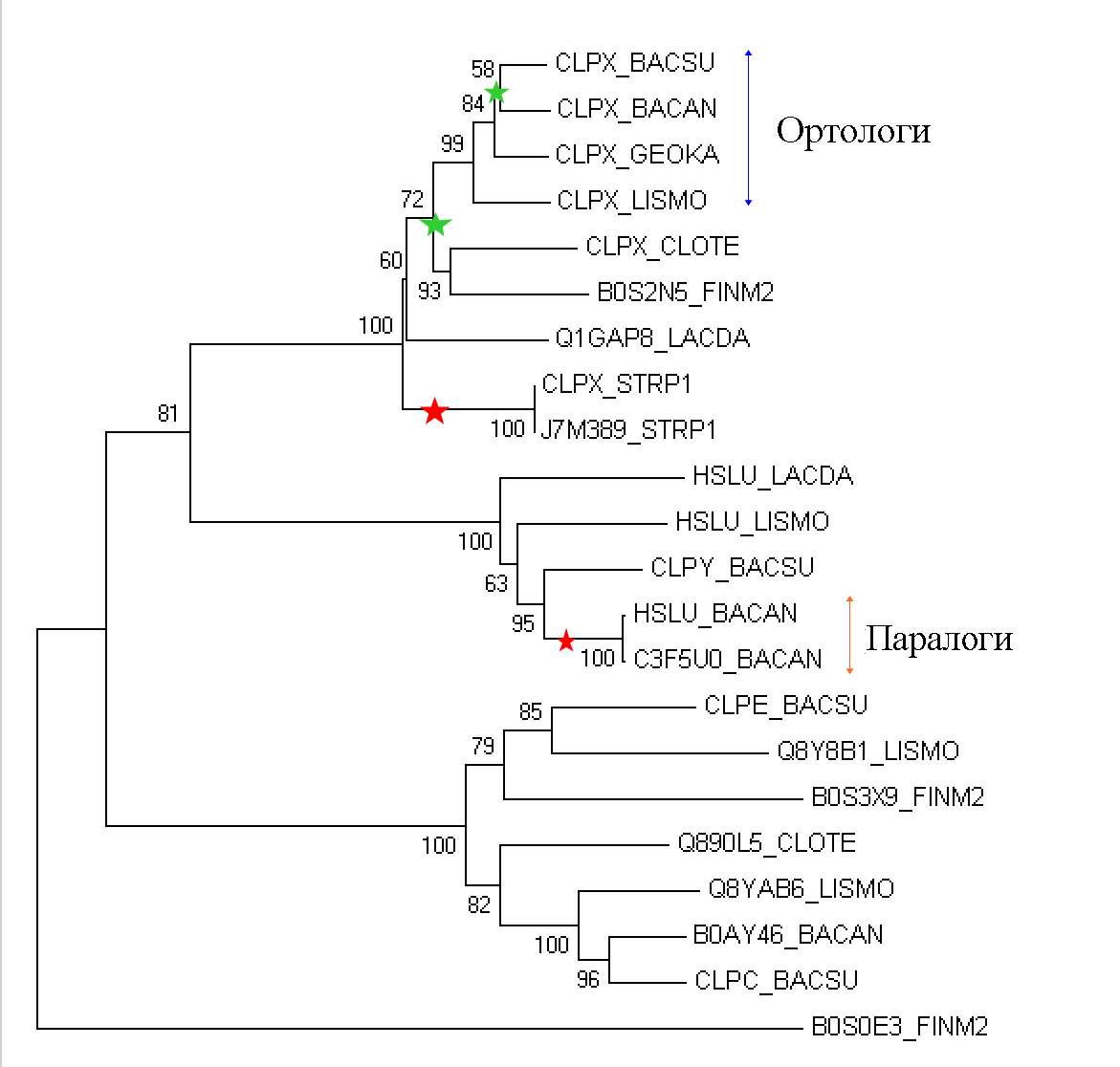
Построение и анализ дерева, содержащего паралоги
Найдем в выбранных бактериях достоверные гомологи белка CLPX_BACSU.
Чтобы найти гомологов в заданных организмах, воспользуемся файлом proteo.fasta на диске P,
там лежат записи банка UNIPROT, относящиеся к бактериям, перечисленным в таблице к заданию 1.
Проведем поиск программой blastp гомологов с порогом на E-value = 0,001.
blastp -task blastp -query ./CLPX_BACSU.fasta -db proteo.fasta -evalue 1e-5 -outfmt "6 sseqid" -out homologs.txt
blastdbcmd -db proteo.fasta -entry_batch homologs.txt -outfmt "%f" -out proteins_for_tree.fasta
Теперь отберем по мнемонике видов только те находки из файла proteins_for_tree.fasta
, которые относятся к отобранным нами бактериям. Отобранные записи представлены в файле
tree_prot.fasta. Эти белки выравним
программой Muscle.
Далее в программе MEGA в меню "Phylogeny" выберем пункт "Construct/Test Neighbour-Joining tree".
В появившемся окошке в строке "Test of Phylogeny" выберем "Bootstrap method" (500 реплик). Полученное дерево
представлено на рис.3.
Считая дерево реконструированным верно, укажем несколько пар ортологов и несколько пар паралогов.
Приведем примеры отражённых на дереве эволюционных событий двух типов:
1) дупликация гена - красная звезда
2) разделение путей эволюции белков в результате видообразования - зеленая звезда
Два гомологичных белка будем называть ортологами, если они а) из разных организмов;
б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. На рис.3.
примеры ортологов обозначены синей стрелкой.
Два гомологичных белка из одного организма будем называть паралогами. На рис.3.
примеры паралогов обозначены оранжевой стрелкой.