Восстановление кристалла из PDB файла
Стабильность кристалла
В структуре белка глюкансукразы Streptococcus mutans (PDB ID 3AIC) опишем взаимодействия с белками из соседних ячеек, определяющие стабильность кристалла из белков.
Кристаллографические характеристики можно найти в записи PDB (поле CRYST1): длины направляющих векторов кристалла, углы между ними, кристаллографическую группу и число молекул в ячейке. Для структуры 3AIC поле выглядит так:
Длины направляющих векторов кристалла - 295.520, 214.410, 220.660 Å, углы между ними все равны 90°. Значит, задана ортогональная система координат. Кристаллографическая группа кристалла - P 21 21 2 (ромбическая). А молекул в ячейке 32.
На самом деле на рисунке мы видим не сам мономер глюкансукразы, а видим "единицу структуры", состоящую из 8 мономеров глюкансукразы с разными мутациями. Структура представлена радужными переходами между мономерами.
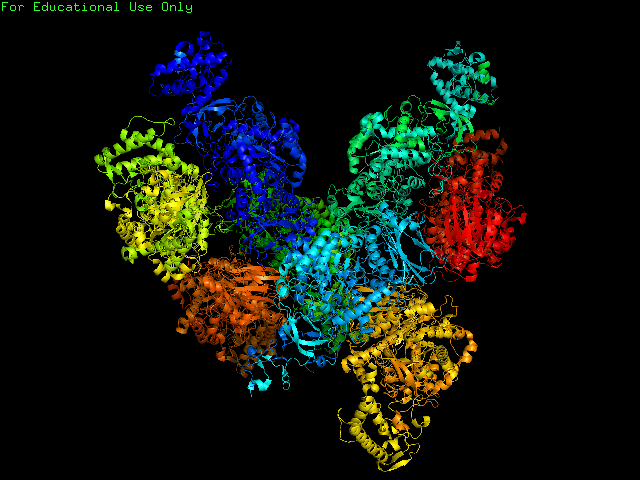
Чтобы восстановить по симметрии соседнюю ассиметрическую единицу в PyMOL воспользуемся командой symexp:
Мы отобразим все образы, находящиеся не далее чем на 5 Å от структуры 3AIC.
На расстояние не далее чем 5 Å от структуры 3AIC находится 6 ассиметрических единиц, которые выделены зеленым цветом. Центральная ячейка показана радужным цветом.
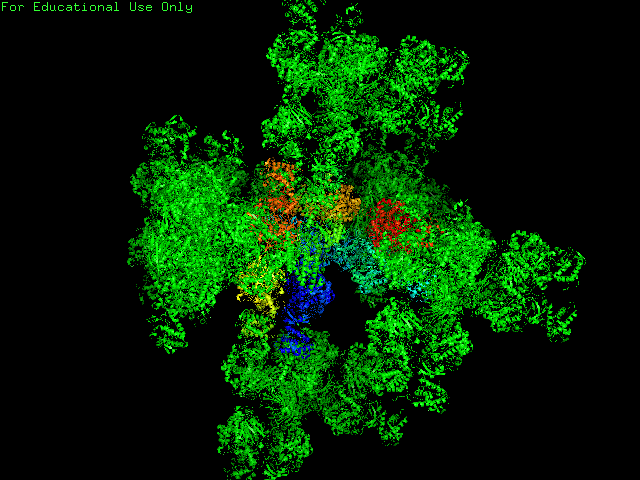
Для удобства будем считать, что взаимодействия центральной ячейки с 6 ассиметрическими единицами являются "идентичными" и рассмотрим взаимодействия только с одной единицей (по правде говоря, связи могут немного отличаться для 6 единиц, так как в них присутствуют разные мутации и они могут приходиться на зону контакта).
Гидрофобными можно считать контакты между остатками: Ala, Ile, Val, Leu, Phe, Met, Trp, Pro, - находящимися на расстоянии в пределах 4 Å. Водородные связи могут образовываться, если акцепторный атом и донорный атом находятся на расстоянии < 3,5Å.
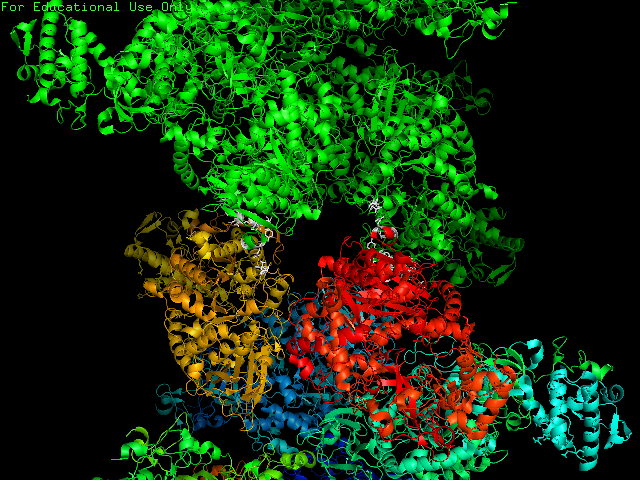
Белым цветом отмечена область остатков, которые находятся на расстоянии < 3,5 Å друг от друга. То есть мы видим, что всего есть 2 разных зон контакта между белками из соседних ячеек.
Водородная связь между остатками TYR-790 центральной ячейки и LYS-1018 соседней.
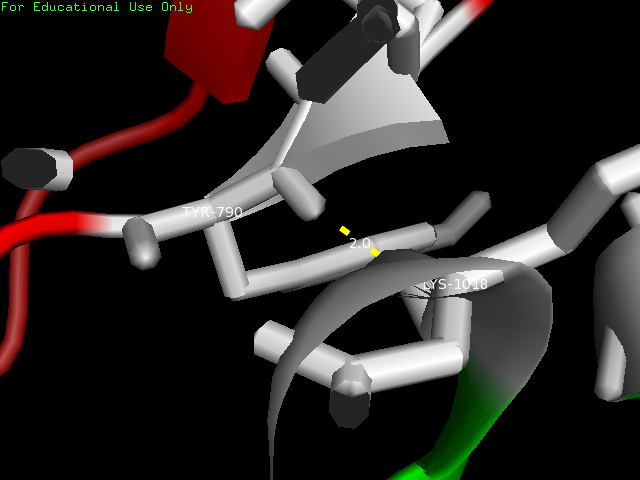
Показанные взаимодействия между соседними ячейками играют важную роль в формировании периодической структуры кристалла. Однако в природе такие взаимодействия скорее всего не характерны.
Cтранное расположение белковых цепей в структуре ДНК-белкового комплекса
Теперь объясним странное расположение белковых цепей в структуре ДНК-белкового комплекса 3HDD. Это гомеодоменный комплекс с ДНК из Drosophila melanogaster. Структура получена при разрешении 2.2 Å. Белок состоит из двух цепей А и В.
ДНК представлена в остовной модели (цепочки покрашены в орагжевый цвет), цепь А желтого цвета, а цепь В зелеая.
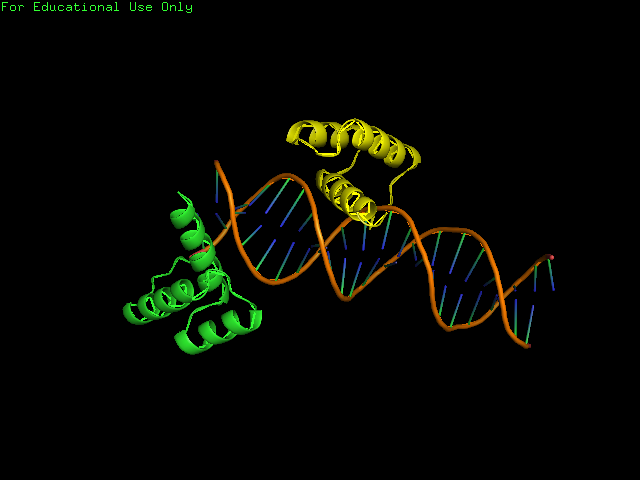
Видно, что цепь В находится «на краю» двойной спирали ДНК. Это довольно странное, на первый взгляд, расположение белковой цепи. Такую странность можно объяснить тем, что на Рис. 5 представлена только часть кристалла. После восстановления соседней ячейки (Рис. 6) видно, что цепь В контактирует с ДНК и в рассматриваемой ячейке, и в соседней.
ДНК представлена в остовной модели (цепочки покрашены в орагжевый цвет), цепь А желтого цвета, а цепь В зелеая.
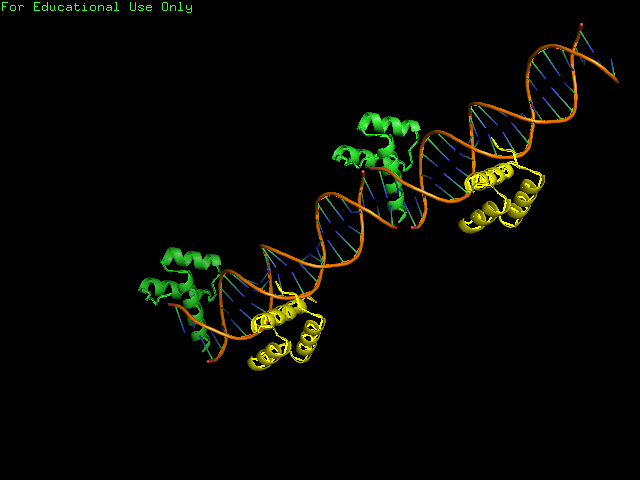
Цепь В располагается как раз на месте разделения спирали ДНК по двум соседним ячейкам, поэтому при рассмотрении только одной ассиметричной единицы, цепь В оказывается «на краю» ДНК.
ДНК представлена в остовной модели (цепочки покрашены в орагжевый цвет), цепь А желтого цвета, а цепь В зелеая. Контакты показаны белым цветом.
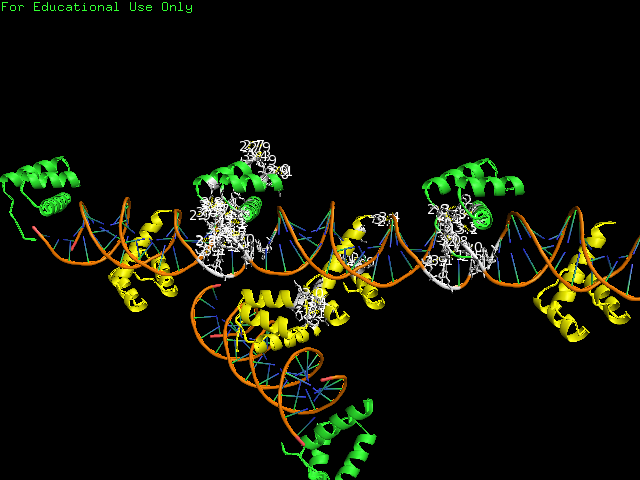
На рис.7 показаны контакты цепи В с продолжающейся в соседней ассиметричной ячейке спиралью ДНК (их длина в среднем составляет 3 Å), а также контакты цепи A.
Взаиомодействия показаны белым цветом, цепь А - желтым цветом.
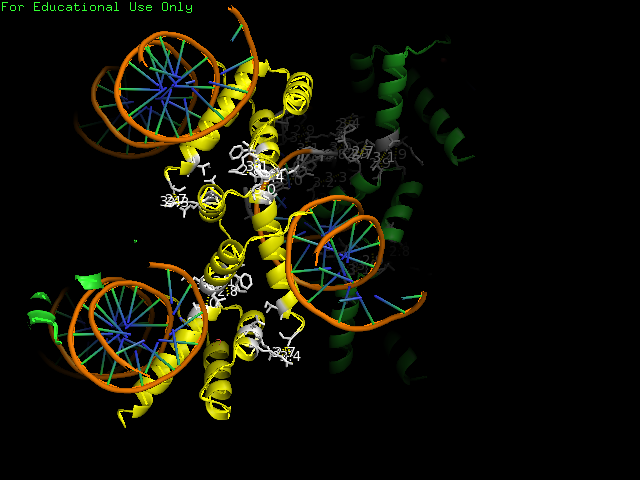
На рис.8 более подробно показаны контакты цепи А с соседней ассиметричной ячейкой (их длина в среднем составляет 3 Å).
