Cовмещение структур
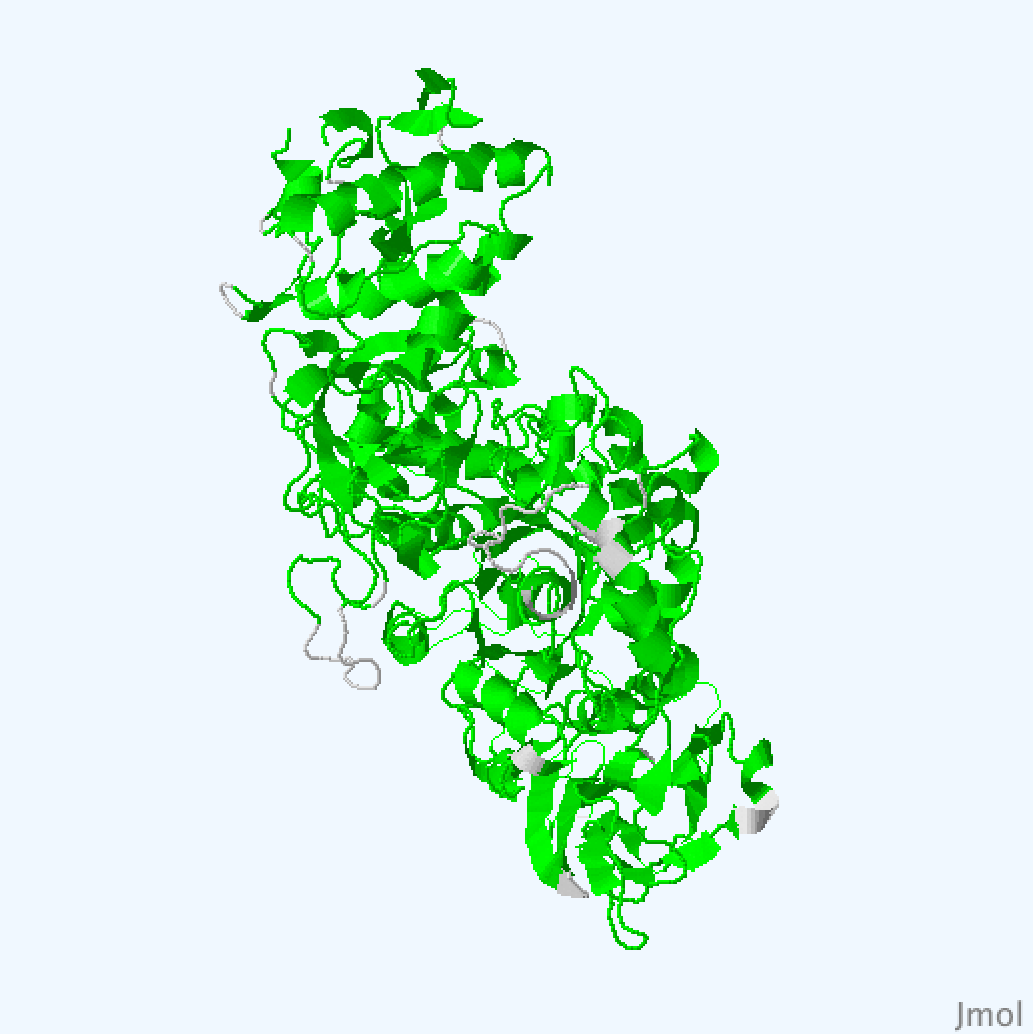
Совмещение структур 3aic белка глюкансукразы и четырех структурных гомологов
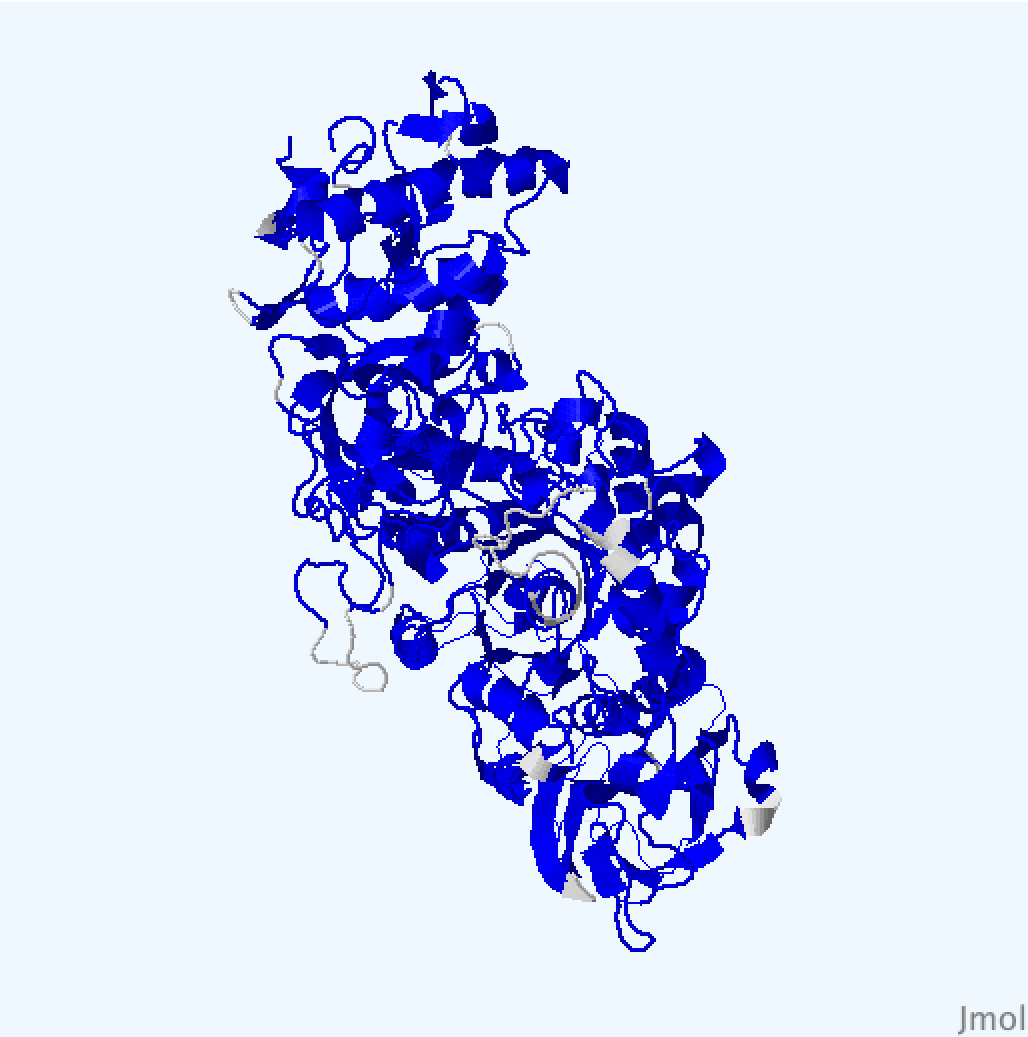
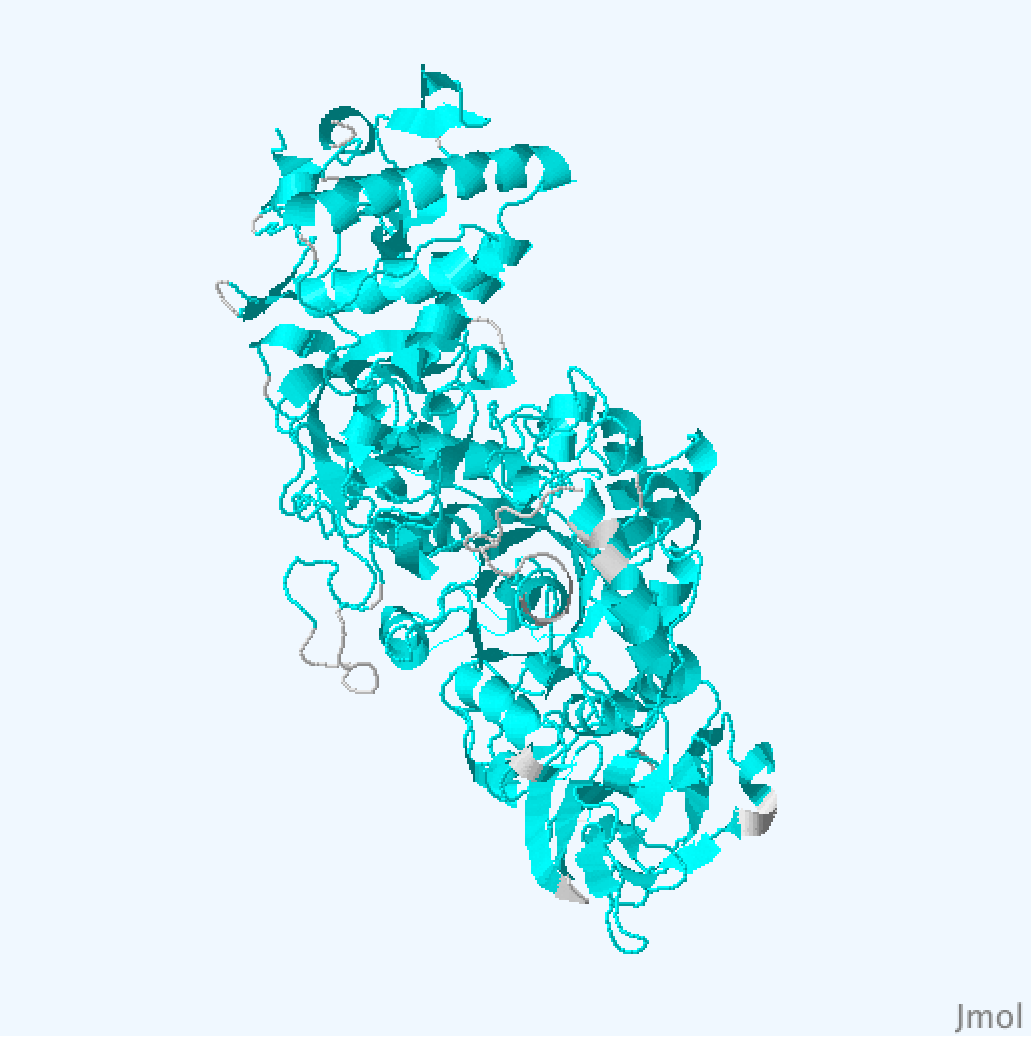
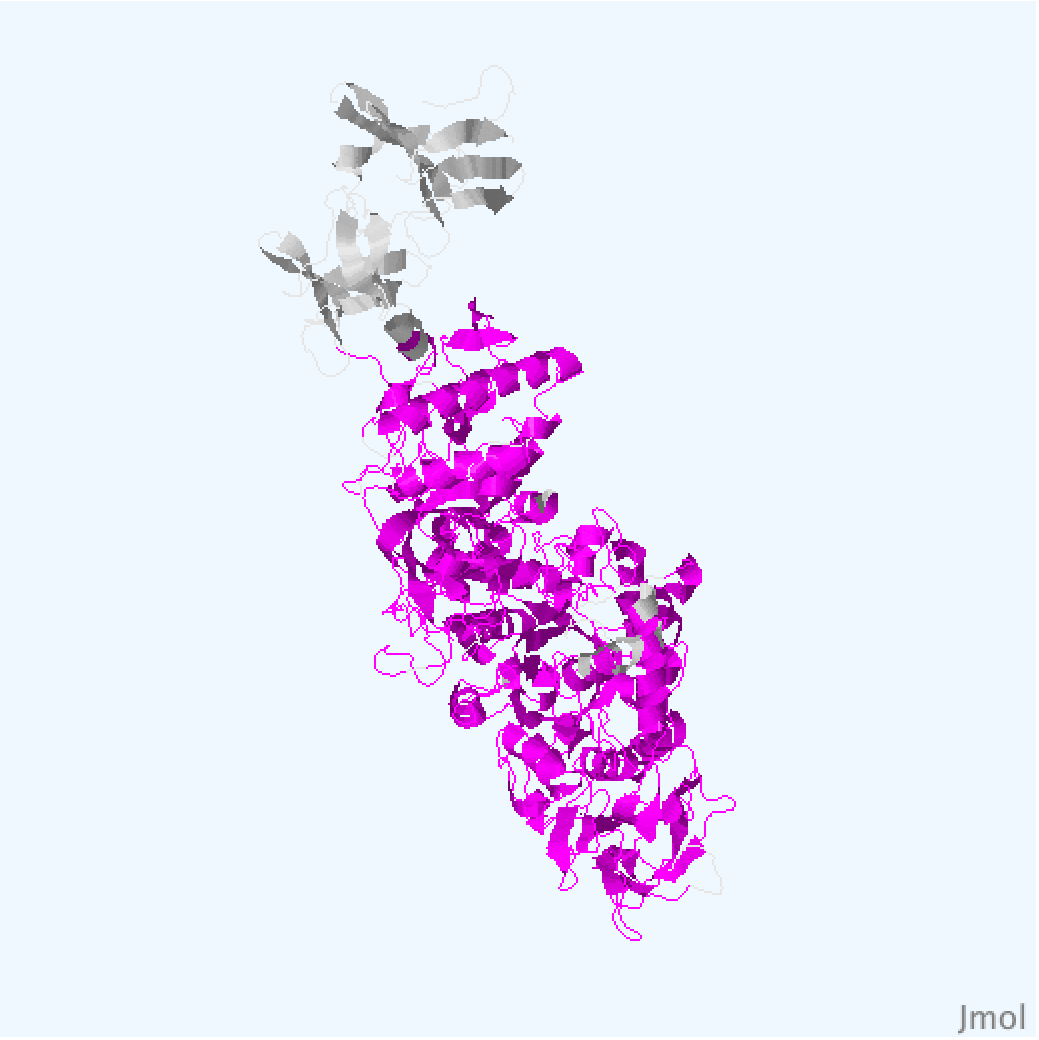
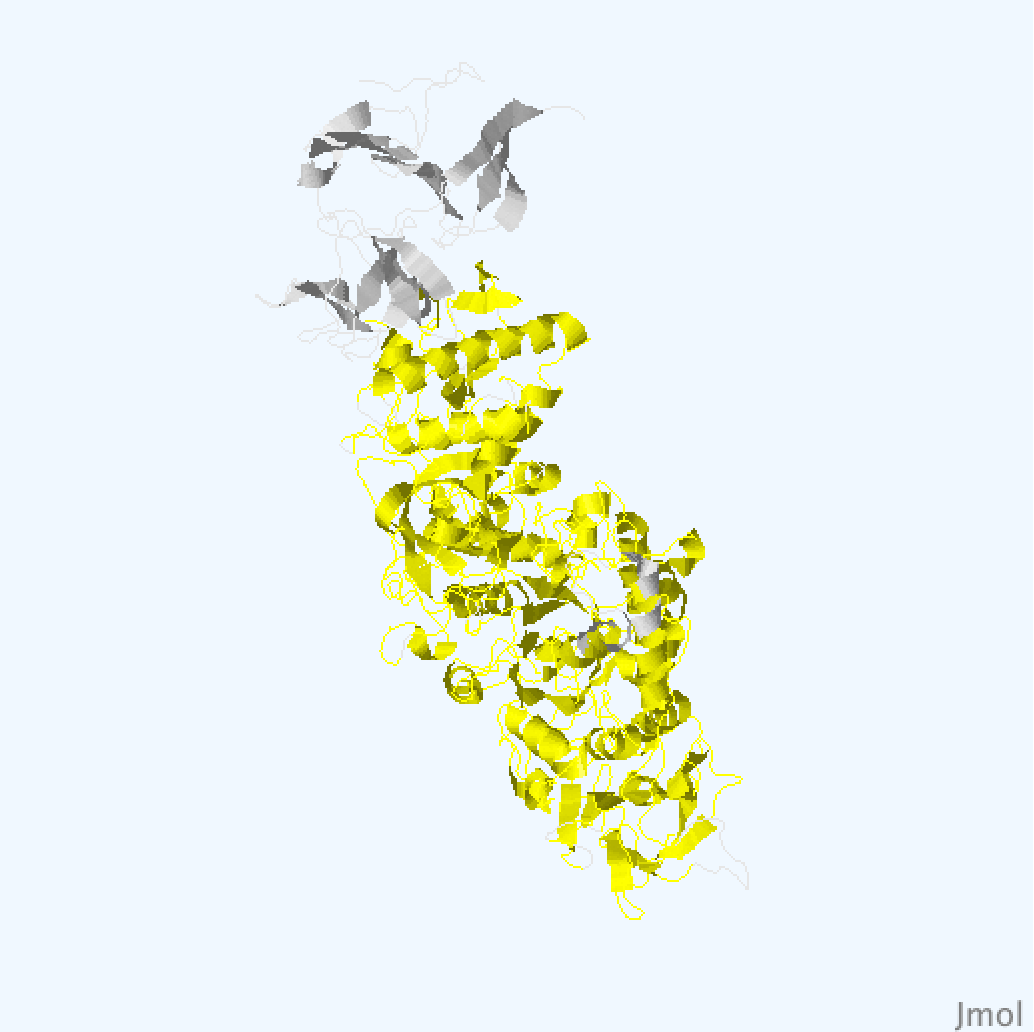
С помощью PDBeFold были найдены структурные гомологи для цепи А структуры 3AIC (Рис. 2): 3aib:A (Рис. 3), 3aie:A (Рис. 4), 3kll:A (Рис. 5), 4amc:A (Рис. 6). Выравнивание структур представлено на Рис.1.
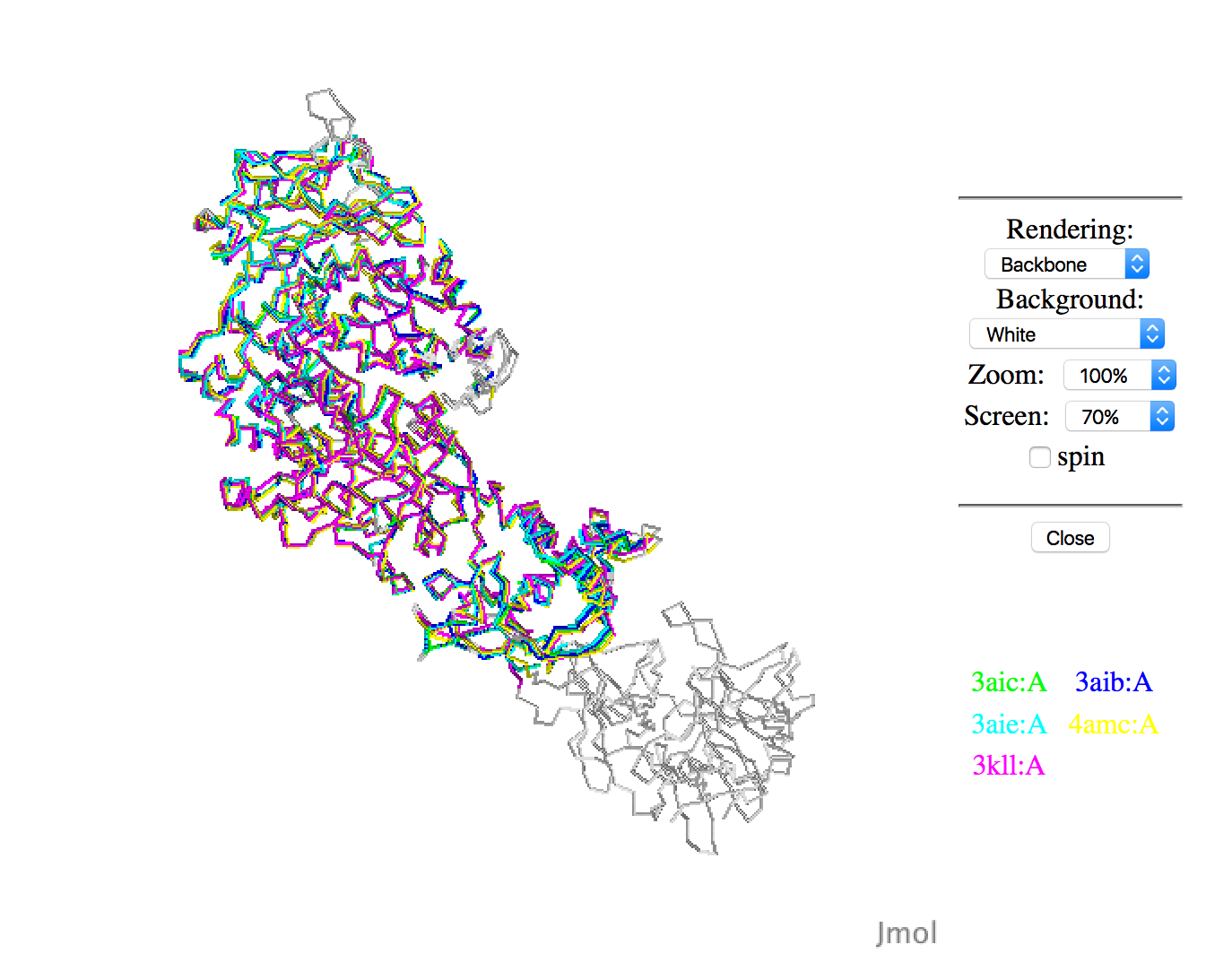
Рис.1. Пространственное совмещения цепи А структуры 3AIC и структурных гомологов (с помощью JMol на PDBeFold)
Структура 3aic - зеленая, 3aib - синяя, 3aie - голубая, 3kll - фуксия, 4amc - желтая.
Выравнивание по структуре (большие буквы в файле с выравниванием), полученное с сервиса PDBeFold можно сравнить с выравниванием последовательностей, выполненным в MUSCLE и визуализированном в программе JalView.
Найдем отличия в этих выраниваниях – пару остатков из разных белков, выровненных по структуре, но не выровненных программой выравнивания последовательностей.
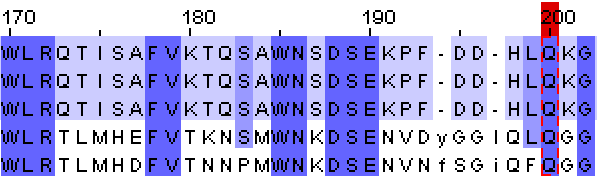
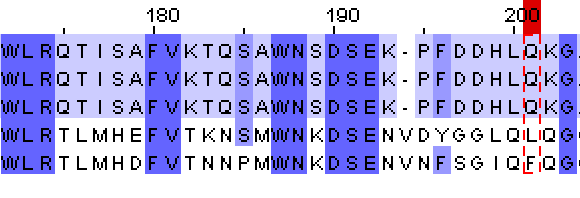
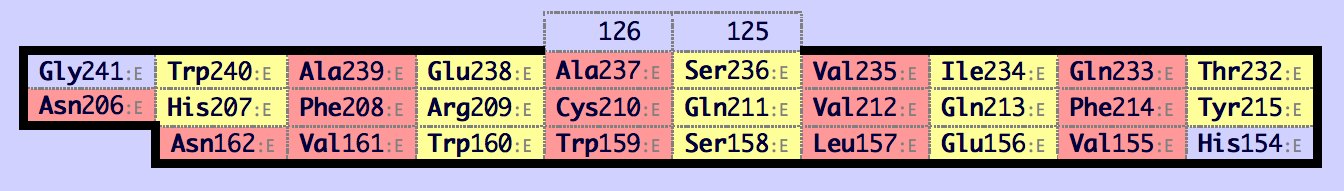
Рис.7. Снизу: выравнивание последовательностей , Сверху: выравнивание последовательностей по структуре Окраска по проценту идентичности. Последовательности структур на выранивниваниях сверху вниз: 3aic, 3aib, 3aie, 3kll, 4amc. Пара остатков (Q, выделенных красной рамкой) из структур 3kll, 4amc, которые выравнены в структурном выравнивание отсутствуют в выранивание последовательностей.
Из рис.7 видно, что в целом, выравнивания очень похожи. Почти все найденные отличия наблюдаются в неструктурированных участках или в граничных остатках вторичной структуры.
Поиск по сходству структур в PDBeFold
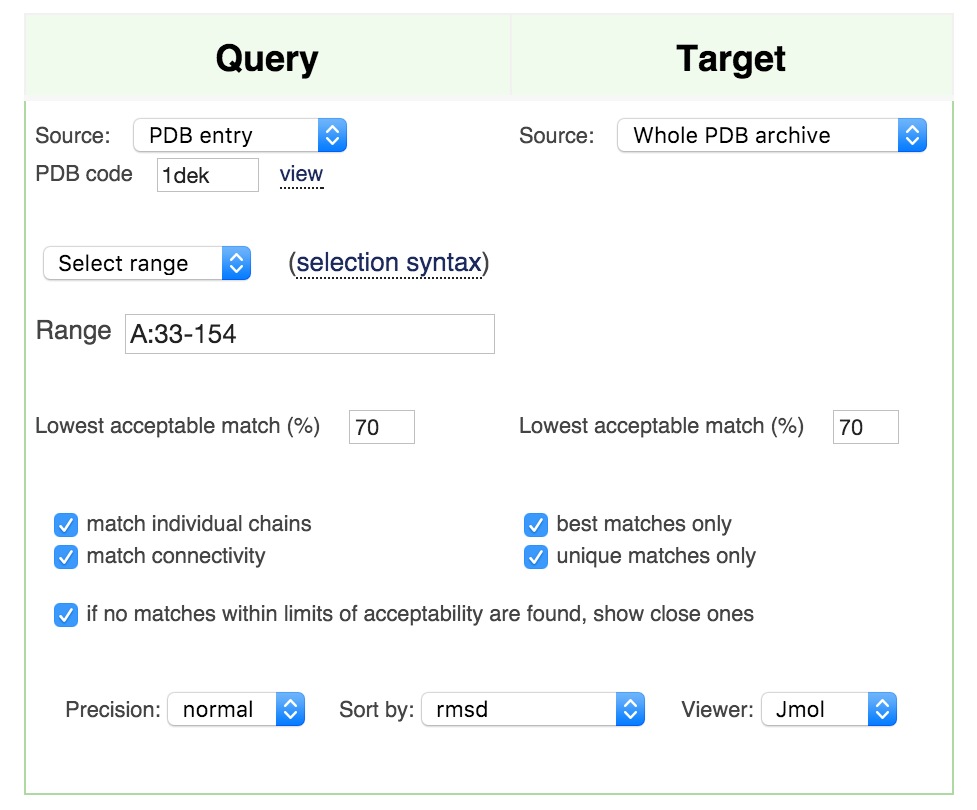
Осуществим поиск структурных гомологов для домена 1dek A: 33-154. Сначала запустим со стандартными параметрами (параметры PDBeFold при поиске гомологов преедставлены на рис.7). Однако, по такому запросу мы не найдем структур, вылезет следующее предупреждение:
Как видно, параметры по умолчанию требуют, чтобы процент совпадения структур был не меньше 70. То есть для мультидоменного белка, искомый домен занимает меньше 70% общей длины белка, сам белок находиться не будет. Но при таких параметрах ничего найдено не было. Нам советуют снизить порог до 60%, что мы и сделаем.
При таких параметрах находятся всего 3 структуры: 2l51:A, 2l51:B, 2l4b:A. Снизим порог до 50%, тогда найдется уже 1052 записи. При снижении порога, например, до 40%, белок 1dek находится в первых строках при сортировке по RMSD.
Совмещение по заданному выравниванию
Cтруктуры константного домена человеческого T-клеточного рецептора из цепи α (1oga, region d: 118-202) и из цепи β (1oga, region e: 119-245) сохраним в формате pdb: alpha.pdb, beta.pdb.
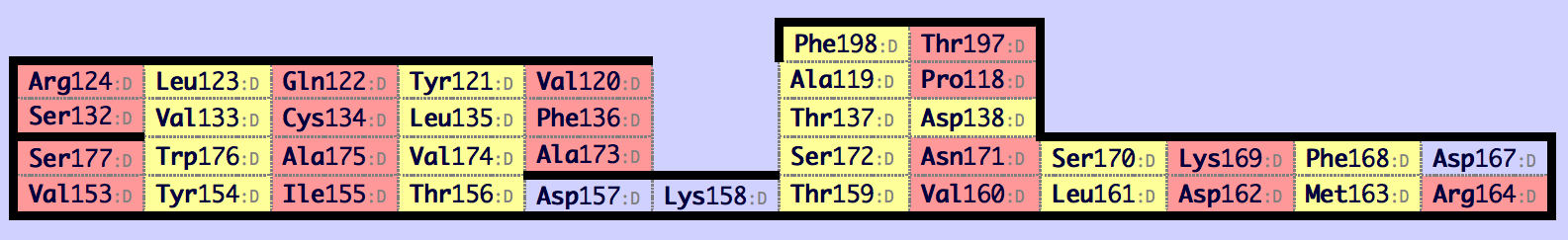
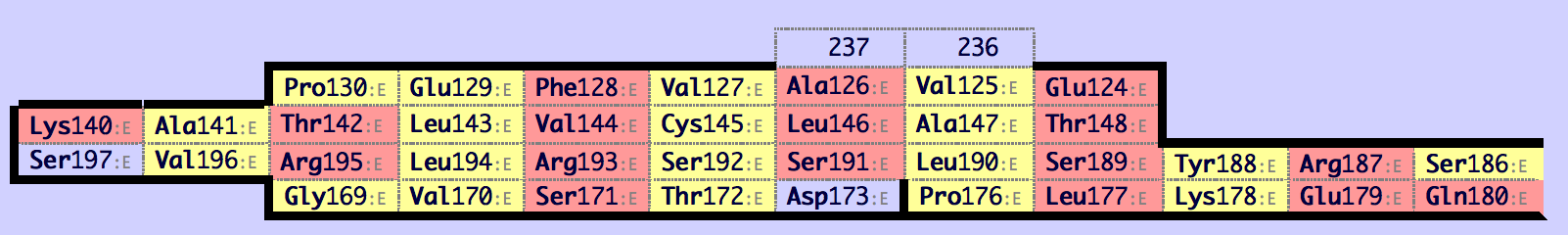
По видимому, лист с картой map_0 в цепи β соответствует листу в цепи α. Карты соответствующих друг другу листов представлены в одной ориентации. Теперь построим выравнивание этих бета-листов. Консервативные остатки цистеина (Cys134 и Cys145) образуют дисульфидную связь. Выровняем два листа, ориентируясь на них. Их выравнивание задает нам выравнивание всего центрального тяжа. Остатки, спаренные с консервативным цистеином, также считаем выровненными. Они задают выравнивание еще каких-то тяжей. Так можно построить выравнивание целых листов.
Полученную информацию о выровненных остатках можно использовать для совмещения структур в PyMol:
alpha and resi 134 and (name CA), beta and resi 145 and (name CA), \
alpha and resi 122 and (name CA), beta and resi 127 and (name CA), \
alpha and resi 175 and (name CA), beta and resi 192 and (name CA), \
alpha and resi 155 and (name CA), beta and resi 172 and (name CA), \
alpha and resi 133 and (name CA), beta and resi 144 and (name CA), \
alpha and resi 135 and (name CA), beta and resi 146 and (name CA)
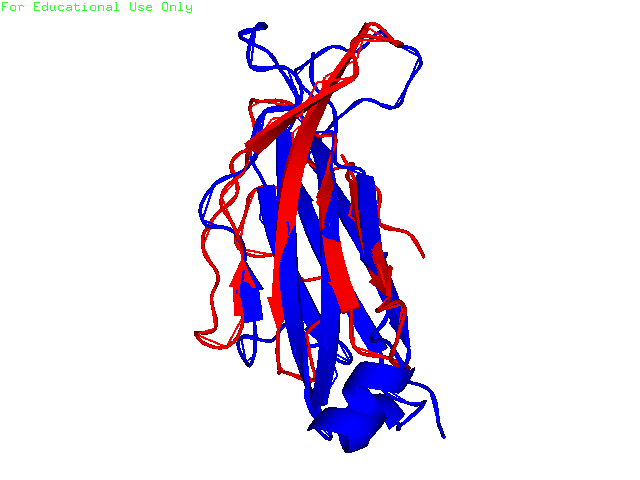
Рис.12. Cовмещение доменов T-клеточного рецептора Структура из цепи α представлена красным, а цепи β синим цветом.
Совмещение сохранено в файле all.pse. На изображении совмещения видно, что общий ход полипептидной цепи в пространстве совпадает, то есть топологии доменов хорошо совпадают.