Построение и визуализация электронной плотности
Описание белка
Структура мутантного фермента глюкансукразы Bacillus subtilis (PDB ID 3AIC, разрешение – 3.11 Å) была выбрана для дальнейшей работы. Было проверено, что структура удовлетворяет следующим требованиям:
- Сервис EDS (Electron Density Server) содержит информацию для выбранной структуры. Это означает, что в PDB, помимо модели структуры, помещены экспериментальные данные – файл структурных факторов (Structure factors).
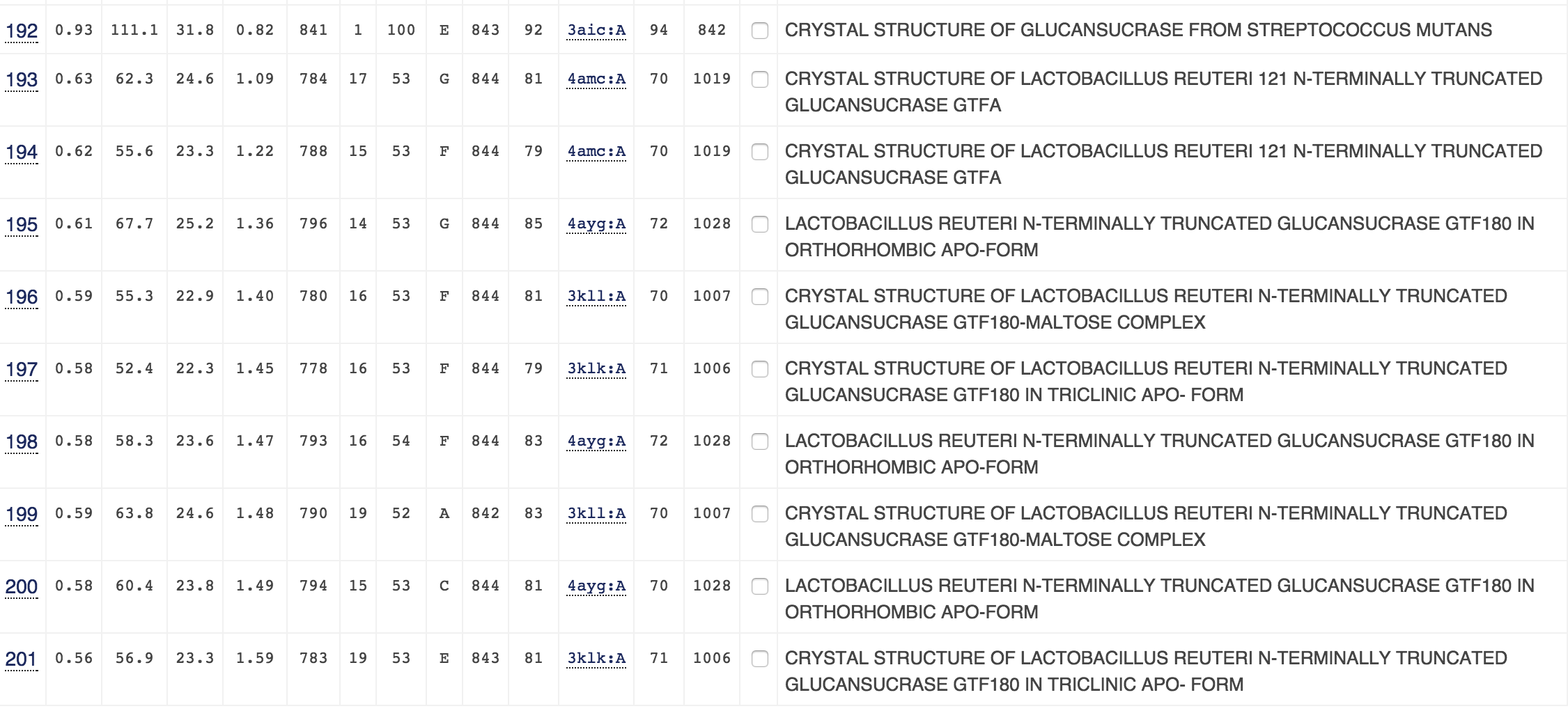
- Имеются подходящие структурные гомологи белка. Для проверки этого был использован сервис PDBeFold. Найдено более 5 разных белков с RMSD от 0.8 до 1.59 ангстрем и N_align от половины числа аминокислотных остатков входного белка до 90%. Полученные результаты представлены на рис.1.
В пятой колонке представлены значения RMSD, в шестой - Nalign (длина выравнивания, то есть число сопоставленных остатков).
Бактерии используют фермент глюкансукразу, чтобы построить длинные, липкие цепочки сахаров, называемых глюканы, используя сахар в нашем рационе. Эти глюканы приклеивают бактерии на поверхности наших зубов, образуя биопленку, которую трудно удалить. Исследователи в настоящее время с помощью структуры этого фермента пытаются найти ингибиторы, которые будут блокировать его действие, и тем самым устранить эту форму защиты для бактерий и в конечном счете защитить нас от кариеса.
Глюкансукразы выполняют две реакции. Во-первых, фермент берет сахарозу и ломает ее пополам, образуя глюкозу и фруктозу. Фруктоза высвобождается, а глюкоза затем добавляется к растущей цепи глюкана. Глюкансукразы - большие белки с большим количествов доменов. Эти реакции проводятся в центральной части фермента, которая включена в эту структуру. У фермента также есть дополнительные домены, которые прикрепляют фермент к существующим цепям глюкана.
Файл с картой электронной плотности для 3AIC был загружен с сайта EDS по ссылке "Download", "Maps". Для визуализации структуры и электронной плотности была использована программа PyMol.
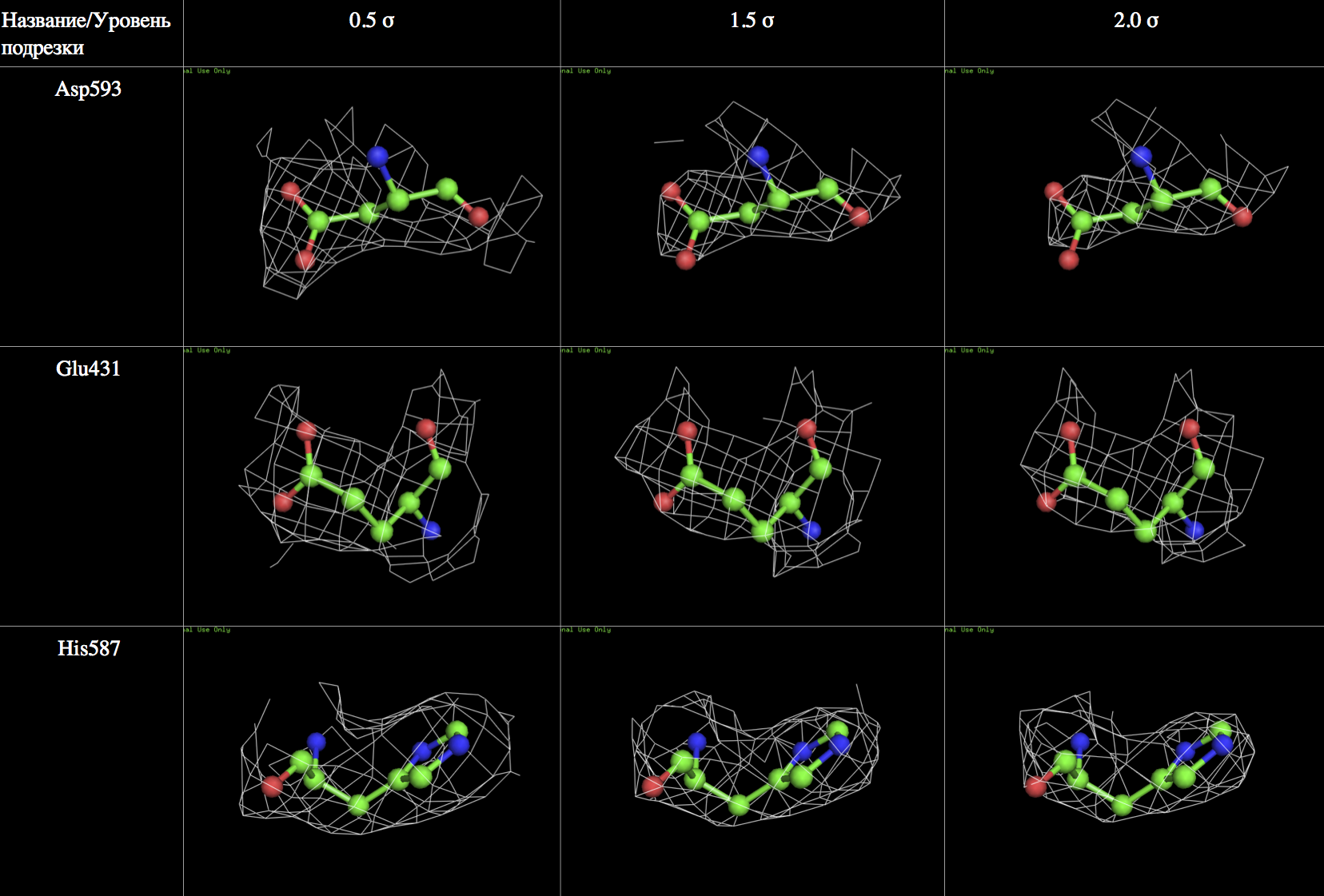
Изображение ЭП вокруг полипептидной цепи
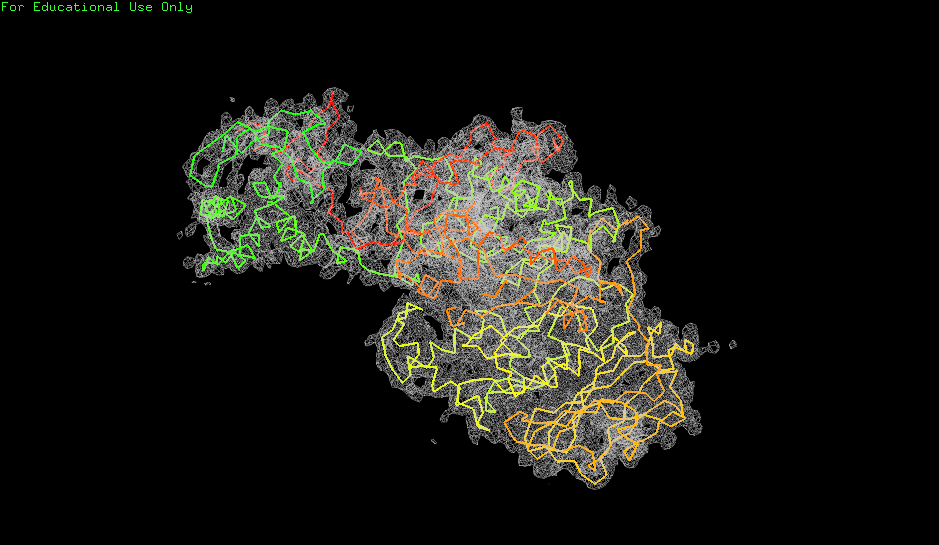
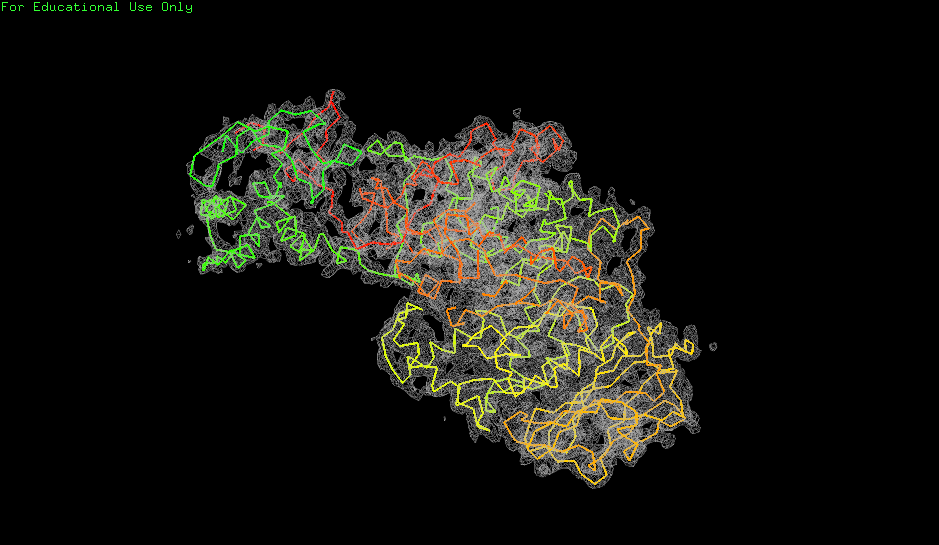
На рис.2 показаны изображения электронной плотности вокруг полипептидной цепи для уровней подрезки 1.0 σ, 1.5 σ и 2.0 σ и параметра carve. Этот параметр нужен для того, чтобы отсекать сигнал от соседних атомов. Он представляет собой расстояние от выбранного множества атомов, в пределах которого будет учитываться электронная плотность. Значение этого параметра в данной работе всегда будет составлять 2.0Å.
load 3aic.omap, 3aic_map
spectrum count, green_yellow_red, chain A, byres=1
isomesh backbone_map, 3aic_map, 1, chain A, carve=2
isomesh backbone_map, 3aic_map, 1.5, chain A, carve=2
isomesh backbone_map, 3aic_map, 2, chain A, carve=2
Изображена элеткронная плотность вокруг полипептидной цепи на уровне подрезки 1, 1.5 и 2 σ соотвественно. Параметр carve для всех изображений составляет 2.0Å. Полипептидная цепь покрашена в градиент от зеленого до красного. Электронная плотность обозначена белым цветом. Все рисунки кликабельны.
| level = 1.0 σ, carve = 2Å | level = 1.5 σ, carve = 2Å |
 |
 |
| level = 2.0 σ, carve = 2Å | |
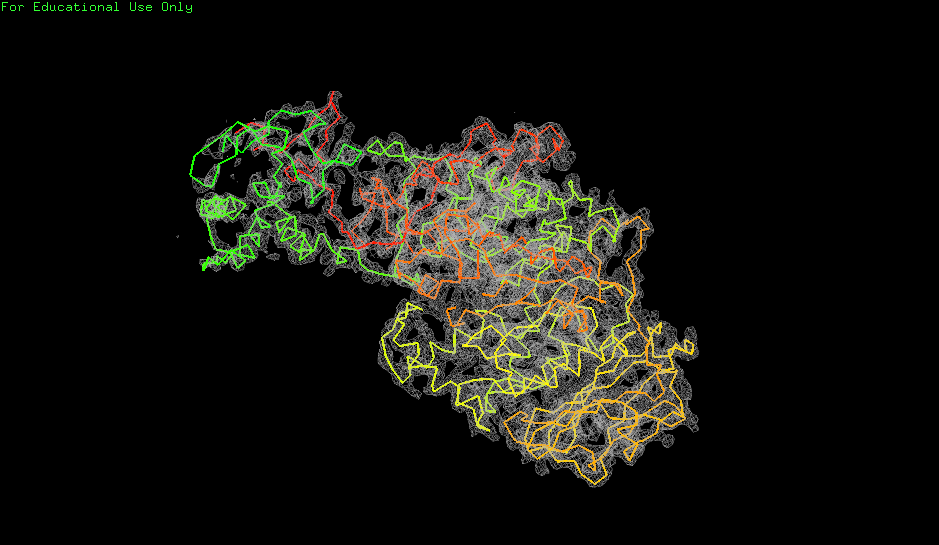 |
На рисунке видно, что электроннная плотность в целом хорошо соответствует модели.
Изображение ЭП вокруг аминокислотных остатков
Для дальнейшего анализа были выбраны аминокислотные остатки Asp593, Glu431, His587. Чтобы получить представленные ниже изображения, были использованы следующие команды (в примере расматриваются команды для одной подрезки для одной аминокислоты):
load 3aic.omap, 3aic_map
hide all
select asp, resi 593 and chain A
show sticks, asp
set stick_radius, 0.1
show spheres, asp
set sphere_scale, 0.2, asp
isomesh map, 3aic_map, 0.5, asp, carve=2
color white, map
set mesh_width, 0.5
center asp
zoom center, 4
Изображение ЭП аминокислотных остатков на разных уровнях подрезки представлены на рис.2.
Из рис.3 мы можем увидеть положение боковых групп аминокислот по сгущениям ЭП. Разрешения структуры в целом не очень достаточно для того, чтобы визуально наблюдать отдельные атомы боковых радикалов на изображении карты электронной плотности.