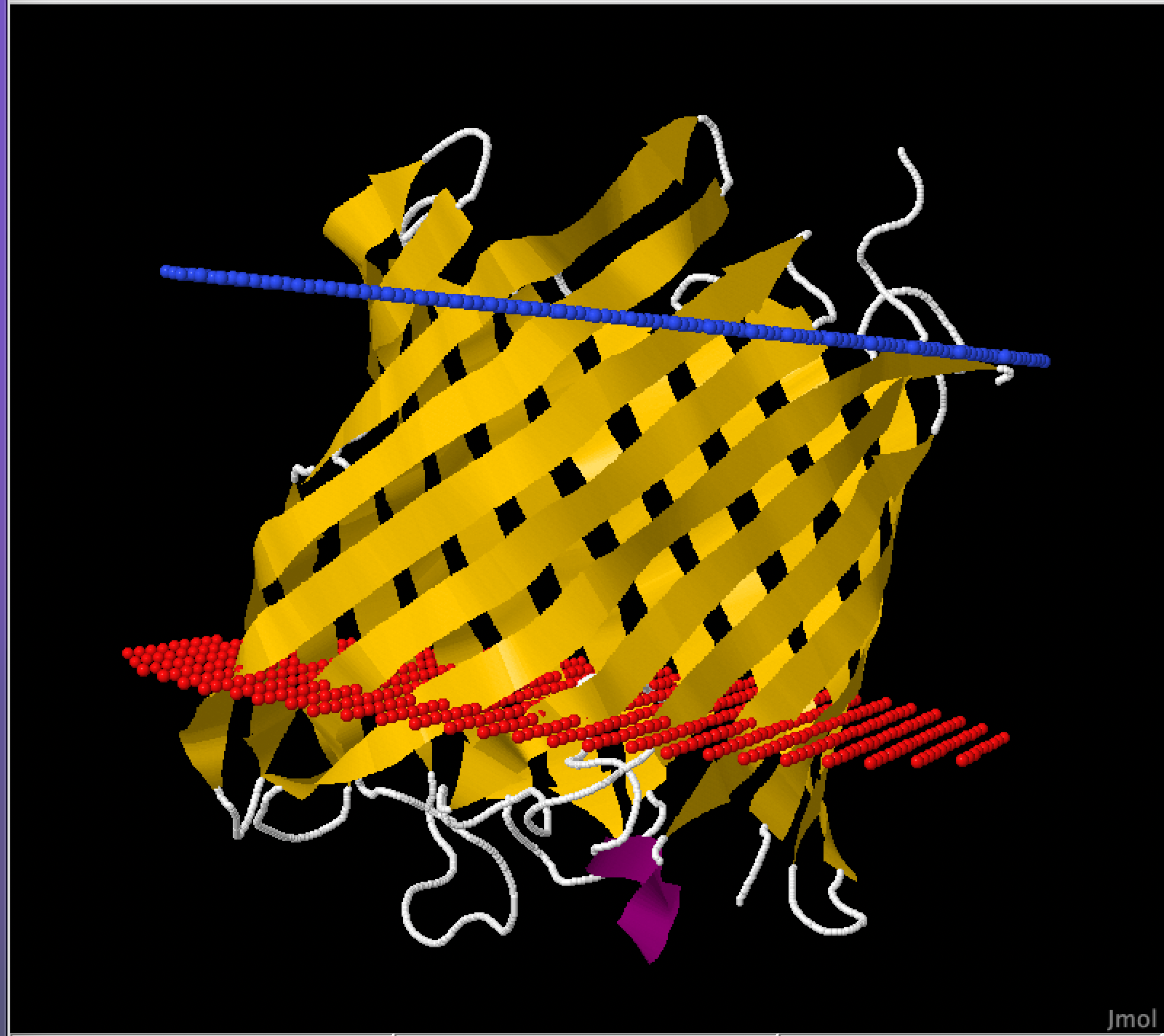
Для выполнения данного задания в базе данных OMP я выбрала экспортный белок Поли-бета-1,6-N-ацетил-D-глюкозамина (англ. Poly-beta-1,6-N-acetyl-D-glucosamine export protein). AC в UniProt: P69434; идентификатор PDB 4y25. По ссылке доступна структура белка в формате PDB.
| Характеристика | Данные |
| Тип белка | Трансмембранный белок, бета-бочонок |
| Суперсемейство | Poly acetyl glucosamine porin |
| Семейсво | Poly acetyl glucosamine porin |
| Организм | Escherichia coli (Кишечная палочка) |
| Локализация | Бактериальная внешняя грам-отрицательная мембрана |
| Uniprot ID | PGAA_ECOLI |
| Толщина гидрофобного слоя | 23.6 Å |
| Число трансмембранных структур | 16 |
| Средняя длина трансмембранных структур | 15 |

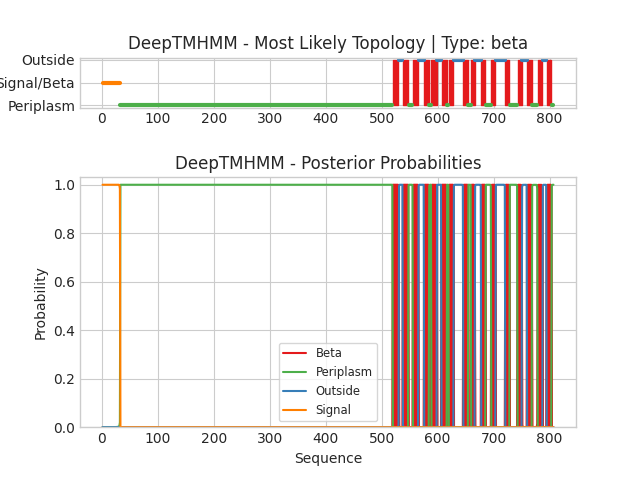
Использовав выделеннный мне белок с AC в UniProt Q52813 (англ. General L-amino acid transport system permease protein AapQ) и выбранный мной для первого задания белок с AC в UniProt P69434 при помощи сервиса DeepTMHMM я провела анализ каждого белка. Сервис позволяет предсказать трансмембранные участки белка по первичой структуре.
Каждый цвет на схеме соответствует определенному участку: Beta - бета-слой, periplasm - периплазматический слой, Outside - внеклеточный, Signal - сигнальный пептид. По горизонтали отмечены координаты остатков белка, по вертикали на верхней схеме рисунка - предсказание положения участков белка, нижняя схема показывает вероятность с которой остаток принадлежит той или иной топологии. Для этого белка предсказано 16 трансмембранных участков. N-конец оказался сигнальным, после него следует большой периплазматический участок. С-конец находится так же в периплазматическом слое. Выдача
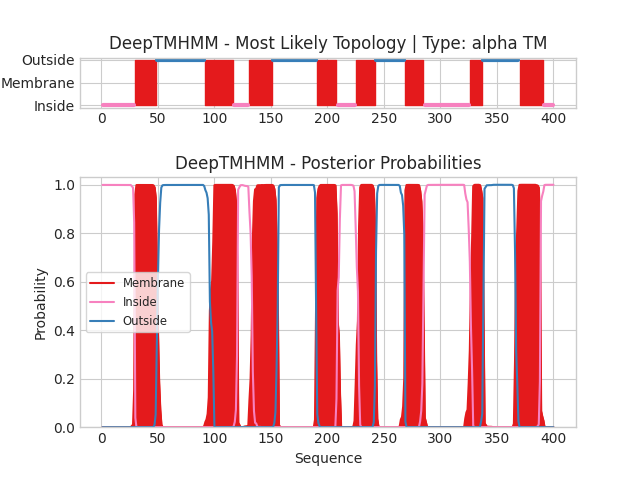
Обозначения на схеме: membrane - участки белка, находящиеся внутри мембраны, inside - участки расположенные с внутренней стороны мембраны, outside - с внешней стороны мембраны. По горизонтали отмечены координаты остатков белка, по вертикали на верхней схеме рисунка - предсказание положения участков белка, а на нижней схеме - вероятность с которой остаток принадлежит той или иной топологии. Программа предсказала 8 трансмембранных участков. N и C концы находятся с внутренней стороны мембраны. Выдача
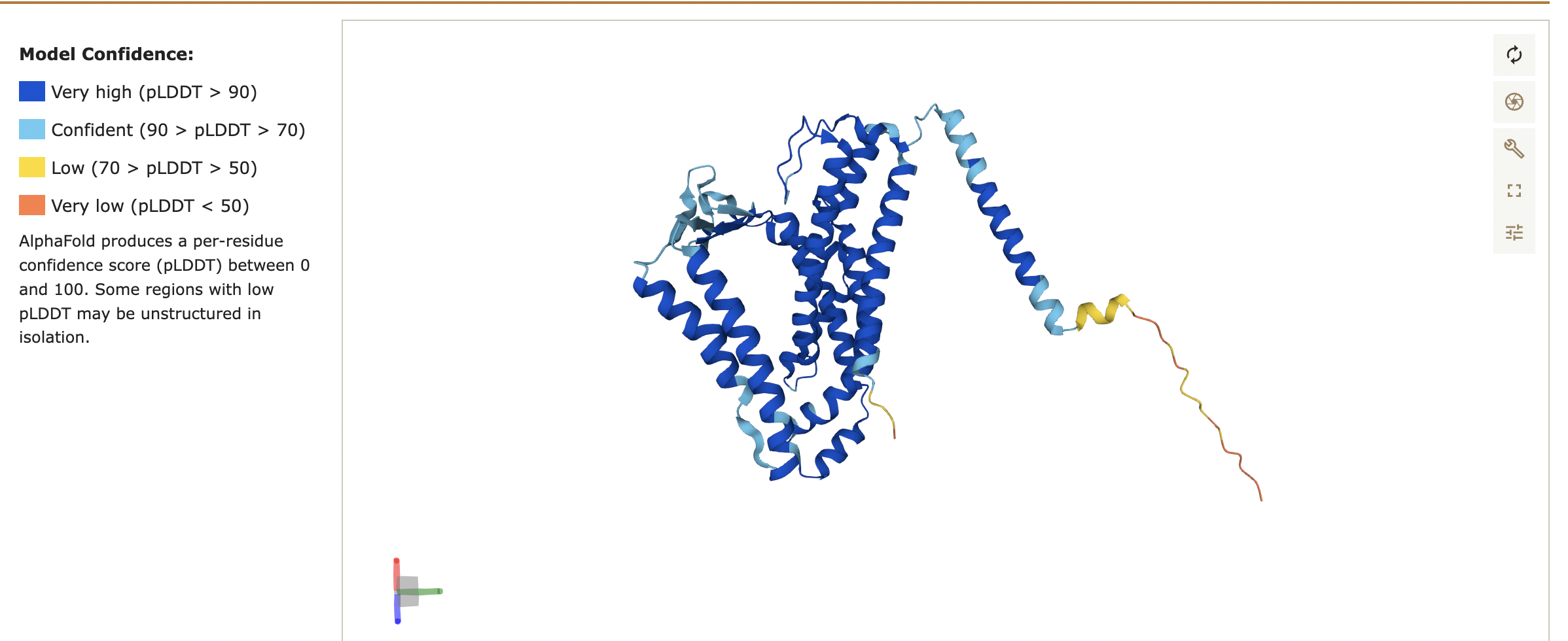
Для данного задания я использовала выданный мне белок с AC в UniProt Q52813. Так как мой белок принадлежит грамотрицательной бактерии Rhizobium, никаких дополнительных параметров я не указывала. В соответствии с данными, полученными в предыдущем задании параметр Topology (N-ter) был установлен in.
| Характеристика | Данные |
| Толщина гидрофобного слоя | 27.2 ± 1.7 Å |
| ΔGtransfer | -82.7 kcal/mol |
| Угол изгиба структур | 11 ± 0° |
| Число трансмембранных структур | 8 |
| Трансмембранные сегменты | 1( 29- 48), 2( 95- 117), 3( 134- 158), 4( 189- 209), 5( 224- 243), 6( 267- 285), 7( 327- 347), 8( 370- 394) |
| Средняя длина трансмембранного сегмента | 20 |
| PDB | PDB файл |

Предскаания в целом схожи. Отличаются в среднем на 1-4 остатка. Количество трансмембранных элементов одинаковое. Спирали находятся в одних и тех же местах.
DeepTMHMM: 1(30-48), 2(96-120), 3(134-156), 4(190-208), 5(228-242), 6(269-284), 7(329-338), 8(367-388)
PPM: 1(29-48), 2(95-117), 3(134-158), 4(189-209), 5(224-243), 6(267-285), 7(327-347), 8(370-394)
Исхдя из структуры белка в AlphaFold структура белка предсказана с очень хорошей точностью, не считая концов, но они не оказывают особого влияния на работу программы PPM.
PPM для беталистового белка: 1(517- 528), 2(541- 548), 3(555- 563), 4(577- 584), 5(591- 596), 6(607- 615), 7(622- 627), 8(646- 654), 9(661- 667),10(678- 685),11(695- 703),12(720- 729),13(740- 751),14(760- 770),15(778- 786),16(796- 806)
DeepTMHMM для беталистового белка: 1(520- 530), 2(538- 547), 3(556- 565), 4(576- 584), 5(589- 598), 6(607- 616), 7(620- 628), 8(646- 654), 9(660- 666),10(678- 685),11(696- 703),12(721- 728),13(743- 750),14(760- 768),15(779- 787),16(795- 803)
Предсказания двух алгоритмов схожи, положение отличается в среднем на 1-3 остатка. Количество трансмембранных элементов совпадает. Бета-листы находятся в одних и тех же местах.
В базе данных TCDB мною был найден выделенный мне белок с AC в UniProt Q52813 и выбранный мной для выполнения первого задания белок с AC в UniProt P69434. Для первого идентификатор в базе данных TCDB стал 3.A.1.3.8(Q52813), для второго 1.B.55.1.1 (P69434).
Значение кода 3.A.1.3.8: А - транспортный белок, 1 - Канал, 3 - семейство транспортеров аминокислотных органокатионов, 3.A.1 - АТФ-связывающая кассета, 3.8 - код белка.
Значение кода 1.B.55.1.1: 1 - Каналы, B - β-бочонковые, 55 - семейство полиацетилглюкозаминовых поринов, 1.1 - код белка.