EMBOSS
Программа getorf
Начнём с получения записи D89965 из банка EMBL. Используем следующую команду: entret embl:d89965, на выходе получаем следующий файл. Почитав запись, узнаём, что имеем дело с последовательностью мРНК из тканей желудка крысы.
Следующий этап - работа с программой getorf. Используя команду tfm getorf, узнаем, как менять параметры поиска открытых рамок считывания. Теперь можем получить набор трансляций всех открытых рамок данной последовательности, которые определены при использовании стандартного кода, имеют длину не менее 30 аминокислот, начинаются со старт-кодона и заканчиваются стоп-кодоном. Первые два из перечисленных параметров установлены по умолчанию, поэтому используем команду:
getorf -sequence D89965.txt -table 1
Полученные рамки считывания содержатся в файле по ссылке. Сравним полученные рамки, с рамкой, содержащейся в записи из EMBL. Ей соответствует рамка номер 5.
Теперь перейдём к записи в Swiss-Prot, на которую ссылается используемая нами запись из EMBL. В ней содержится информация о белке из кишечной палочки, и последовательности этого белка соответствуеют девятая из ранее нами найденных рамок считывания. Противоречие.
Почему же связанные записи в двух банках относятся к разным белкам и организмам? Вспомним, что EMBL является архивной базой данных, записи там проверяются только тем человеком, который их туда добавил, тогда как Swiss-prot - реферируемая база данных. Вероятно, во время эксперимента с клетками желудка крысы примешались клетки E.coli, которые могли обитать в ЖКТ или просто находиться в воздухе. Ошибочно полученная последовательность была принята за крысиную, в EMBL запись не отредактирована, а в Swiss-prot была проверена и исправлена.
Файлы-списки
Cкачаем в файл adh.fasta все доступные в банке Swiss-prot последовательности алкогольдегидрогиназ, используя команду
entret sw:adh*_*
Теперь получим список универсальных адресов последовательностей алкогольдегидрогеназ такой командой:
infoseq -only -usa -sequence adh.fasta > adh_usa.txt
Из полученного списка выберем последовательности, принадлежащие только заданным организмам, используя команду
grep -f zveri.txt adh_usa.txt > adh_zveri.txt
Список организмов можно увидеть, перейдя по этой ссылке, список полученных адресов - по этой. Теперь можем получить сами последовательности, соответствующие адресам из списка, используем команду
seqret @adh_zveri.txt > seq_zveri.fasta
Полученный в итоге файл с последовательностями можно увидеть здесь. Теперь мы знаем, как быстро и удобно с помощью программ пакета EMBOSS создать выборку последовательностей, заданных каким-либо списком.
EnsEMBL
EnsEMBL - портал, предназначенный для визуализации известной информации о геномах человека и животных. Чтобы рассмотреть различные его функции, возьмём ранее выбранный ген ODC1 в качестве примера.
Если провести поиск с главной страницы, введя в строку "Search" краткое название гена, можно получить ссылки на страницы о гене и о транскриптах. Всего их 4, видимо, по разным вариантам сплайсинга. Их же можно увидеть и на странице о гене (см. рис. 1), там есть так же информация о типе гена, методе предсказания, вторичной структуре и др. С этой же страницы можно перейти к "Region in detаil"
|
| Рис. 1 Таблица с расположением кодирующих участков последовательности четырёх вариантов транскриптов гена ODC1. |
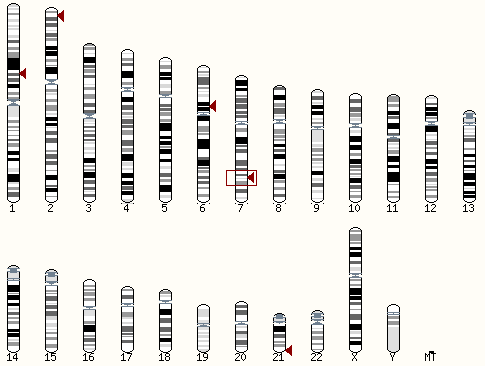
Теперь поищем ген ODC1 в геноме человека с помощью сервиса "BLAST/BLAT". На странице видим изображение кариотипа человека с отметками хороших выравниваний на хромосомах (см. рис. 2)
|
| Рис. 2 Кариотип человека. Красными треугольниками отмечено расположение удачных находок, в рамку обведён лучший хит. |
Ещё можно посмотреть локализацию в геноме последовательности-запроса и выровненной последовательности (см. рис. 3).
|
| Рис. 3 Расположение последовательностей запроса и выравнивания в геноме. |
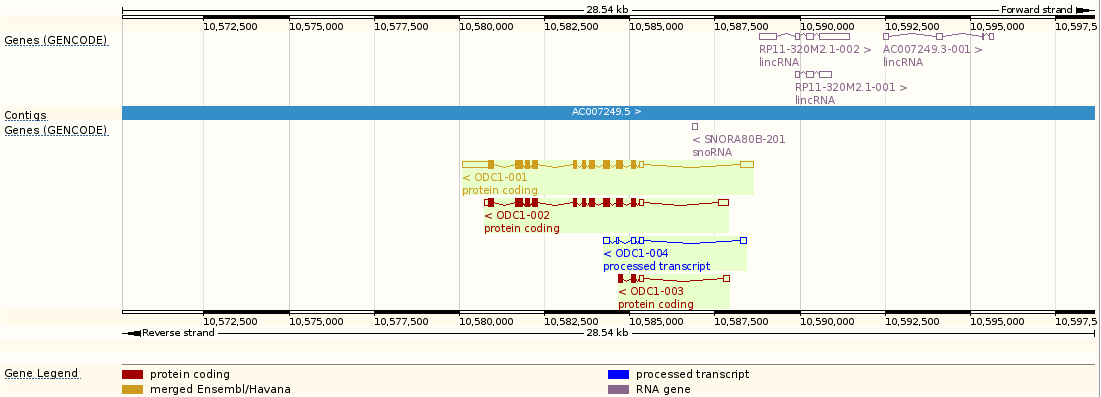
Далее идёт список лучших выравниваний с координатами начала и цонца, направлением цепи, названием хромосомы, E-value, процентом сходства и длиной. Кроме того, для каждой последовательности есть ссылки на выравнивание - [A], геномную последовательность -[G], и подробную карту участка - [C]. Перейдя по третьей ссылке попадаем на страницу "Region in detail", аналогичную которой мы уже видели, когда рассматривали поиск по названию гена. Одна из последовательностей, собственно, и есть та, которую мы нашли. На странице "Region in detail" можно увидеть расположение гена на хромосоме (рис. 4), окружающий участок с другими генами (рис.5), расположение кодирующих участков при различных вариантах сплайсинга (рис. 6).

|
| Рис. 4 Расположение гена на хромосоме (выделен красной рамкой). |

|
| Рис. 5 Отображение региона, в котором расположен ген. В сине-голубой строке размечены контиги, чёрно-белая полоса сверху отмечает координаты. |

|
| Рис. 6 Различные варианты сплайсинга гена ODC1. Обозначения те же, что и на рис. 5. Внизу также показан процент GC-пар (красная линия - 50%). |