Алгоритмы реконструкции деревьев. Укоренение и бутстреп.
Укоренение в среднюю точку
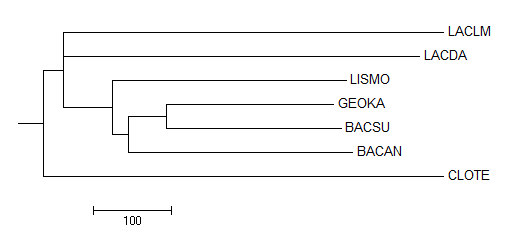
Для того, чтобы сделать правильно укоренённым дерево, полученное в предыдущем практикуме методом neighbor-joining, проведём укоренение в среднюю точку. Для этого воспользуемся программой retree пакета PHILIP, где имеется команда M - "Midpoint root the tree". Полученное дерево визуализируем с помошьюпрограммы MEGA (см. рис. 1).
|
| Рис. 1. Дерево, полученное алгоритмом neighbour-joining и укоренённое в среднюю точку. |
Полученное дерево не совпадает с правильным, но укоренено верно - корень отделяет Clostridia от Bacilli
Использование внешней группы
В некоторых случаях удобно использовать укоренение с помощью внешней группы. Для этого нужно добавить лист, о котором заранее известно, что он отделился от общего предка раньше разделения интересующих нас групп.
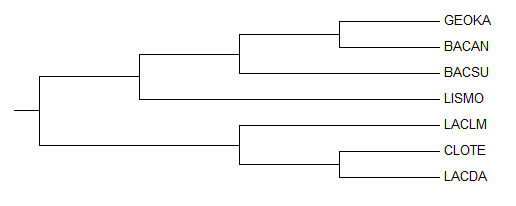
В случае построения дерева по белкам бактерий, выбранным в предыдущем практикуме, возьмём в качестве внешней групы шаперонин E. coli. Добавим его в fasta-файл с остальными последовательностями (полученный файл - здесь), выравняем с помощью программы Muscle в JalView (файл с выравниванием - здесь), в программе MEGA посторим дерево методом Maximum Parsimony, укореним в ветвь, отделяющую ECOLI и затем отобразим только дерево без ветви ECOLI (см. рис. 3).
|
| Рис. 2. Дерево, полученное укоренением с использованием внешней группы. |
Дерево не изменилось и всё так же отличается от правильного. Это объясняется тем, что эволюция последовательности одного белкане отражает эволюцию видов.
Бутстреп
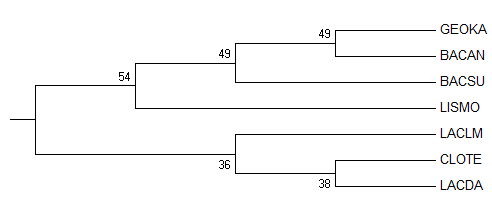
Проведём бутстрэеп-анализ интересующих нас белков, используя "Bootstrap method" программы MEGA с числом реплик равным 100. Деревья, построенные методом Maximum paesimony и укоренённые по внешней группе (ECOLI, как и в предыдущем случае) приведены на рисунках 3 и 4.
|

|
| Рис. 3. Original tree. | Рис. 4. Bootstrap consensus tree. |
Полученные деревья не отличаются друг от друга, но отличаются от правильного дерева, и ветви их меют небольшую поддержку, что говорит о недостоверности.