Исследование DNA primase small subunit P.furiosus
На данной странице вы можете ознакомиться с отчетом по блоку "UniProt". Далее будут представлены описание выбранного белка и обучающие задачи по работе в UniProt.
Введение
Информация о белке
DNA primase small subunit PriS (p41): субъединица ДНК-праймазы, участвующая в процессе репликации ДНК. Данный белок относится к семейству малых субъединиц праймаз эукариотического типа и встречается у Archaea и Eukaryota [1]. p41 является каталитическим компонентом в гетеродимере и напрямую осуществляет функции синтеза РНК-праймера и его удлинения. Что интересно, эта субъединица способна самостоятельно синтезировать длинные фрагменты ДНК без матрицы, однако гетеродимер (с большой субъединицей p46) имеет повышенную аффинность к матрице и синтезирует более короткие фрагменты ДНК. [2]. В качестве кофакторов p41 выступают Mg2+ и Mn2+, которые необходимы для связывания NTPs и удлинения цепи соответственно [3]
Информация об организме
Pyrococcus furiosus, дословно переводящийся как "бешенный огненный шар", относится к домену Archaea, филуму Euryarchaeota - сестринскому к группе Crenarchaeota, включающей в свой состав Eukaryota.[4] P.furiosus относится к классу Thermococci, представители которого отичаются крайней экстремофильностью и имеют оптимум температуры роста 75-100°C и pH 6-7.[5] Сам P.furiosus же имеет крайне высокие температуры оптимума роста (до 100°C) и является гетеротрофным анаэробом.[6] Морфологически это Грам-отрицательный кокк, несущий до 50 жгутиков на одном полюсе клетки. При культивировании на дрожжевом и бактериальном экстракте, пептоне, триптоне с добавлением олигосахаридов было выявлено образование H2, CO2 и H2S (при добавлении S0) как метаболических продуктов.[6]
Основная информация о белке.
| UniProt ID(AC) | PRIS_PYRFU (Q9P9H1) | ||
|---|---|---|---|
| Evidence | 1: Evidence at protein level | ||
| RecName |
DNA primase small subunit PriS (EC 2.7.7.-) AltName: DNA primase 41 kDa subunit (p41) |
||
| EMBL IDs | AB037376; BAB03293.1; -; Genomic_DNA. AE009950; AAL80234.1; -; Genomic_DNA. |
||
| PDB ID | 1G71 | ||
| Length | 347 AA | ||
| M (Da) | 40772 | ||
Вторичная структура
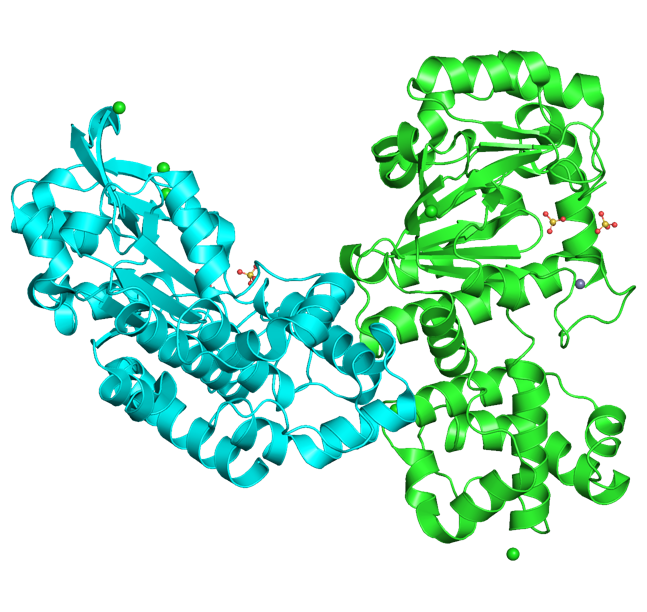
Вторичная структура белка в PDB была получена с помощью рентгеноструктурного анализа[7], дополнительно были определены важные аминокислотные сайты, участвующие в связывании лигандов. По результатам выдачи PDBe-KB было установлено, что структура белка определена на 99%, причём "непокрытыми" остаются 345-347 аминокислотные остатки. Белок представляет собой гомодимер, каждая из субъединиц которого связывает ионы Zn2+, Cl- и SO42-/. В информации о белке в UniProt указано наличие 21 спирали, 11 β-слоёв и 3 поворотов (turns).[1] Стоит отметить наличие в белке Zn-knuckle мотива, осуществляющего связывание ионов цинка (в полях FT UniProt). Предполагается, что цинк в данном белке участвует в координации и правильном расположении первого нуклеотида будущего праймера [8]. На рисунках слева вы можете видеть формат отображения структуры белка в PDB_Structure, где отмечены важные аминокислотные сайты и домены. Для более подробного ознакомления перейдите по ссылке.
Сеансы поиска в UniProt
1. Встречаемость ДНК-праймазы эукариотического типа в трёх доменах жизни.
Запрос: goa:("dna primase activity") family:"eukaryotic-type primase small subunit family" NOT taxonomy:"Archaea [2157]" NOT taxonomy:"Eukaryota [2759]"
По упомянутой в введении принадлежности данной праймазы к Eukaryota и Archaea было решено проверить, встречается ли PriS в иных таксонах жизни. По результатам выдачи запроса было найдено лишь одно совпадение (PRIM_OSHVF) - предполагемая (Inferred from homology) праймаза герпесвируса устриц 1 (OsHV-1, сем. Malacoherpesviridae). Вероятно ген праймазы случайно встроился в геном вируса при сборке вирусных частиц, но данный вопрос требует дальнейшего изучения.
Запрос: goa:("DNA primase activity [3896]") family:primase NOT family:"eukaryotic-type primase small subunit family" NOT family:"eukaryotic-type primase large subunit family"
Данный запрос был направлен на выявление праймаз других семейств. Было выявлено 3 семейства:
- DnaG primase family: широко представленная среди бактерий ДНК-праймаза. Представляет собой мономер и синтезирует короткие РНК-праймеры.[9]
- Archaeal DnaG primase family: ДНК-праймаза бактериального типа, встречающаяся у Archaea. Действительно, в геноме некоторых архей закодированы и экспрессируются оба типа ДНК-праймаз(бактериального и эукариотического типа).[10] По дополнительному запросу было установлено, что в геноме P.furiosus также присутствует ДНК-праймаза бактериального типа.(Inferred from homology)
- Herpesviridae DNA primase family: ДНК-праймаза герпесвирусов, встречающаяся в том числе у многих известных представителей данной группы (HHV, CMV, лимфотропный герпесвирус B и др.) . Дополнительно проведенный запрос показал, что у упомянутого выше OsHV-1 (герпесвируса устриц) отсутствует праймаза данного типа, что связано с присутствием в его геноме праймазы эукариотического типа.
2. Цинк-связывающий мотив в структуре ДНК-связывающих белков.
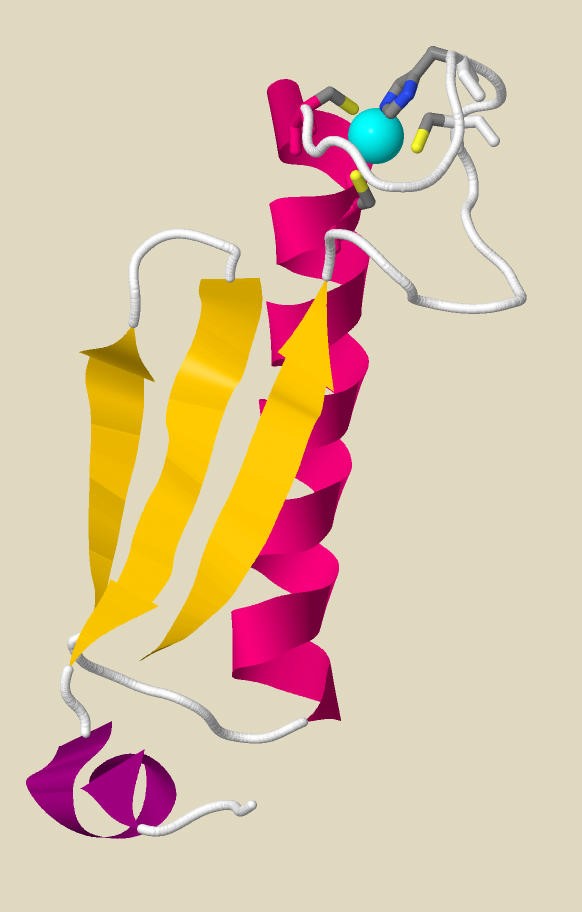
Первый Zn-связывающий мотив был открыт в составе транскрипционного фактора X.laevis (TFIIIA) и назван цинковыми пальцами. В настоящий момент Zn-fingers domain одним из самых известных мотивов (второй по представленности). Он участвует в белок-белковых взаимодействиях, а также взаимодействиях белков с ДНК и РНК.[11]
В структуре PriS (поле FT "MOTIF") указан "Zinc knuckle motif" - CNHEPGTVCPIC (-C-X1-H-X5-C-X2-C-), напоминающий класс zn ribbon domain.[11] Дальнейшие запросы направлены на выявление белков, участвующих в репликации ДНК и содержащих в своей структуре похожие Zn-связывающие мотивы.
Запрос: annotation:(type:motif "zinc knuckle motif")
Выдача по данному запросу состоит только из малых субъединиц ДНК-праймазы эукариотического типа. Дальнейший поиск направлен на расширение круга белков.
Запрос: NOT goa:("DNA primase activity [3896]") annotation:(type:metal zinc) annotation:(type:function "dna synthesis") keyword:"dna replication"
По данному запросу были получены записи о ДНК-полимеразах Eukaryota, АТФазах WRNIP1 и 2 позиции Unreviewed (фактор сплайсинга Aphanomyces invadans и ДНК-полимераза Geobacillus kaustophilus (strain HTA426)). Внимания заслуживает тот факт, что все белки реплисомы, осуществляющие синтез ДНК, содержат ион цинка в координированном виде. Это действительно подтверждает гипотезу о его роли в данном процессе.
Дополнительно
Интерес для исследования представляет присутствие Zn-связывающего мотива в других организмах/таксонах. Для ответа на этот вопрос был использован портал EMBL-EBI, а именно предоставляемый ими BLASTP. Последовательность PriS P.furiosus была "забластована" на entry UniProtKB/Swiss-Prot. Неудивительно, что в выдаче присутствовали лишь ДНК-праймазы родственных видов. После этого был выбран участок с 91 по 120 аминокислоту, содержащий Zn-связывающий мотив. В результате были получены все те же ДНК-праймазы родственных видов и конотоксины с значительно низким identity. Все вышесказанное может свидетельствовать об уникальности данного мотива для рода Pyrococcus либо о неумении автора пользоваться BLAST'ом.
Анализ гомологичных последовательностей с помощью UniRef
UniRef50
ID: UniRef50_Q9V292
В данном кластере находятся 83 записи малых субъединиц ДНК-праймаз, принадлежащих к представителям семейства Thermococcaceae (Thermococcus, Pyrococcus, Palaecoccus). "Representative" записью является PRIS_PYRAB - праймаза Pyrococcus abyssi, что говорит о большей изученности этого вида. Представленность схожих белков невелика и ограничена лишь семейством Thermococcaceae.
UniRef90
ID: UniRef90_Q9P9H1
В данном кластере присутствуют 3 записи о PriS в различных штаммах P.furiosus. "Representative & Seed" является именно изучаемая запись, она же единственная была проверена вручную и перенесена в Swiss-Prot. Из полученного результата можно сделать вывод, что вариант праймазы, характерный для P.furiosus отличается от родственных организмов и является уникальным в пределах вида.
UniRef100
ID: UniRef100_Q9P9H1
Данный кластер полностью повторяет UniRef90 и дополнительной информации не несет.
Работа с протеомами
Таксон Archaea на данный момент является крайне малоизученным доменом жизни во многом из-за сложности культивирования и получения образцов. Это объясняется уникальной экстремофильностью представителей: археи обитают в термальных морских источниках(черных курильщиках), особенно солёных водоемах(соляные озера, моря), источниках с высоким или низким pH[12]. В нижеприведенной части для сравнения с гипертермофильным P.furiosus был выбран крайне галофильный Halobacterium salinarum с целью выявить сходства и различия приспособлений к обитанию при высокой температуре и солёности соответственно.
Pyrococcus furiosus
| Proteome ID | UP000001013 |
|---|---|
| Taxonomy | 186497 - Pyrococcus furiosus (strain ATCC 43587 / DSM 3638 / JCM 8422 / Vc1) |
| Status | Reviewed |
| Proteins | 2,045 |
| Reviewed entries | 502 (24,54%) |
Halobacterium salinarum
| Proteome ID | UP000000554 |
|---|---|
| Taxonomy | 64091 - Halobacterium salinarum (strain ATCC 700922 / JCM 11081 / NRC-1) |
| Status | Reviewed |
| Proteins | 2,426 |
| Reviewed entries | 491 (20,24%) |
Анализ представленности белков разных функциональных групп
Белки с ферментативной активностью
В данном разделе приведено сравнение представленности белков различных классов у выбранных видов архей. Для поиска использовался UniProtKB, ссылки на запросы приведены в таблице(под количеством белков в каждом КФ). Дополнительно были рассчитаны доли белков в протеоме (приведены в скобках)
| Организм | КФ1 | КФ2 | КФ3 | КФ4 | КФ5 | КФ6 | КФ7 | Общее количество |
|---|---|---|---|---|---|---|---|---|
| P.furiosus | 65 (3,18%) | 141 (6,89%) | 92 (4,50%) | 48 (2,35%) | 23 (1,12%) | 57 (2,79%) | 5 (0,24%) | 431 (21,08%) |
| H.salinarum | 97 (4,00%) | 210 (8,66%) | 116 (4,78%) | 63 (2,60%) | 34 (1,40%) | 67 (2,76%) | 11 (0,45%) | 598 (24,65%) |
Основная часть классов ферментов представлена одинаково у двух видов, однако относительно значимые различия наблюдаются в полях КФ2(трансферазы) и КФ7(транслоказы). Различия в представленности трансфераз может быть объяснена гиперсалинными условиями обитания H.salinarum, так как одним из способов приспособления является присутствие в клетке большого количества мембранных переносчиков, выкачивающих излишек анионов. Для H.salinarum дополнительно описаны уникальные K+-переносчики: KdpFABC P-type ATPase и др.[13] Связь представленности трансфераз и среды обитания не так очевидна.
Трансмембраные белки
По поисковым запросам UniProtKB(указаны в названии археи) для H.salinarum и P.furiosus был произведен анализ представленности трансмембранных белков в протеоме. Результаты выдачи приведены в таблице.
| Организм | Кол-во записей | Доля в протеоме | Присутствие бактериородопсина |
|---|---|---|---|
| P.furiosus | 117 | 5,72% | - |
| H.salinarum | 464 | 19,13% | + |
Из полученных выводов видно, что в протеоме H.salinarum присутствует большее количество трансмембранных белков, чем у P.furiosus. Данный факт кореллирует с приспособлением организмов к гиперсалинным местообитаниям: осмолярность цитозоля таких видов гораздо выше нормы и соответствует окружающей среде, и, дополнительно, в клетке экспрессируется большое число трансмембранных ионных каналов, помп и насосов, которые активным транспортом выкачивают избыток ионов.[13] Дополнительно, для H.salinarum описан уникальный вид аноксигенного фотосинтеза - светозависимая ретиноид-содержащая протонная помпа бактериородопсин , которая осуществляет создание протонного градиента для синтеза АТФ.[14] Фотосинтетический аппарат включает в состав большее количество трансмембранных белков, и поэтому их доля в протеоме выше.
Биохимические характеристики
Для исследования особенностей выбранных архей было решено проанализировать температурные особенности ферментов. Благодаря предварительному сеансу поиска в UniProtKB было установлено, что для белков H.salinarum отсутствует информация об их температурной устойчивости (раздел Temperature dependence поля CC). Для P.furiosus были скачаны все entry ферментов, содержащих информацию о Temperature dependence (с помощью поиска). Для дальнейшего получения информации использовался скрипт Python. По результатам были получены значения в 70-100°C, что говорит о приспособленности организма к гипертермофильным условиям на биохимическом уровне.
Дополнительная информация
История изменений записи (entry) в UniProt
Запись о PriS P.furiosus была внесена в UniProtKB/TrEMBL 1 октября 2000 года на основе экспериментальных данных (1: Evidence at protein level). 1 мая 2001 года она была обновлена , а 27 апреля 2001 года - внесена в UniProt/Swiss-Prot (то есть получила статус Reviewed). Запись суммарно модифицировалась 116 раз (последний: 7 апреля 2021). В целом, запись редактировалась параллельно с развитием баз данных: вносились новые Cross-references, ссылки, связанные публикации. Однако следует обратить внимание на динамику редактирования информации о Zn-связывающем мотиве. До 61ой версии записи участок связывания цинка значился в поле FT как ZN_FING, однако после модификации это "звание" было изменено на MOTIF. Вероятно, в ходе исследования было установлено, что данный участок не относится к позициям в существующей классификации Zn-fingers, поэтому ему было присвоено более размытое название - мотив. (сплошная головная боль с этим мотивом, чесслово)
REFERENCES
- Информация о белке в UniProt
- Liu L. et al. The Archaeal DNA Primase: biochemical characterization of the p41-p46 complex from Pyrococcus furiosus. // Journal of Biological Chemistry. – 2001. – Т. 276. – №. 48. – С. 45484-45490.
- Keen B. A. et al. Molecular dissection of the domain architecture and catalytic activities of human PrimPol // Nucleic acids research. – 2014. – Т. 42. – №. 9. – С. 5830-5845.
- Cavalier-Smith, T., Chao, E.EY. Multidomain ribosomal protein trees and the planctobacterial origin of neomura (eukaryotes, archaebacteria)// Protoplasma 257, 621–753 (2020).
- Schut G. J. et al. The order thermococcales and the family thermococcaceae // The Prokaryotes: Other Major Lineages of Bacteria and the Archaea. – 2014. – С. 363-383.
- Fiala G., Stetter K. O. Pyrococcus furiosus sp. nov. represents a novel genus of marine heterotrophic archaebacteria growing optimally at 100 C // Archives of Microbiology. – 1986. – Т. 145. – №. 1. – С. 56-61.
- Augustin M. A., Huber R., Kaiser J. T. Crystal structure of a DNA-dependent RNA polymerase (DNA primase) // Nature structural biology. – 2001. – Т. 8. – №. 1. – С. 57-61.
- Martínez-Jiménez M. I. et al. The Zn-finger domain of human PrimPol is required to stabilize the initiating nucleotide during DNA priming //Nucleic acids research. – 2018. – Т. 46. – №. 8. – С. 4138-4151.
- Bocquier A. A. et al. Archaeal primase: bridging the gap between RNA and DNA polymerases //Current Biology. – 2001. – Т. 11. – №. 6. – С. 452-456.
- Liu B. et al. A primase subunit essential for efficient primer synthesis by an archaeal eukaryotic-type primase //Nature communications. – 2015. – Т. 6. – №. 1. – С. 1-11.
- Krishna S. S., Majumdar I., Grishin N. V. Structural classification of zinc fingers: survey and summary //Nucleic acids research. – 2003. – Т. 31. – №. 2. – С. 532-550.
- Baker B. J. et al. Diversity, ecology and evolution of Archaea //Nature microbiology. – 2020. – Т. 5. – №. 7. – С. 887-900.
- Gunde-Cimerman N., Plemenitaš A., Oren A. Strategies of adaptation of microorganisms of the three domains of life to high salt concentrations //FEMS microbiology reviews. – 2018. – Т. 42. – №. 3. – С. 353-375.
- Gonzalez O. et al. Systems analysis of bioenergetics and growth of the extreme halophile Halobacterium salinarum //PLoS Comput Biol. – 2009. – Т. 5. – №. 4. – С. e1000332.