Трансмембранные белки
Последующие задания выполнялись с белком TCR_STRRM - детерминанта устойчивости к тетрациклину (tetB). Этот белок, по данным с Uniprot, является эффлюксной помпой, выкачивающей комплекс тетрациклина с металлом из клетки в обмен на H+ (т.е. это антипортер).
Для работы была скачана последовательность белка и предсказание его трехмерной структуры.
1. База данных OPM
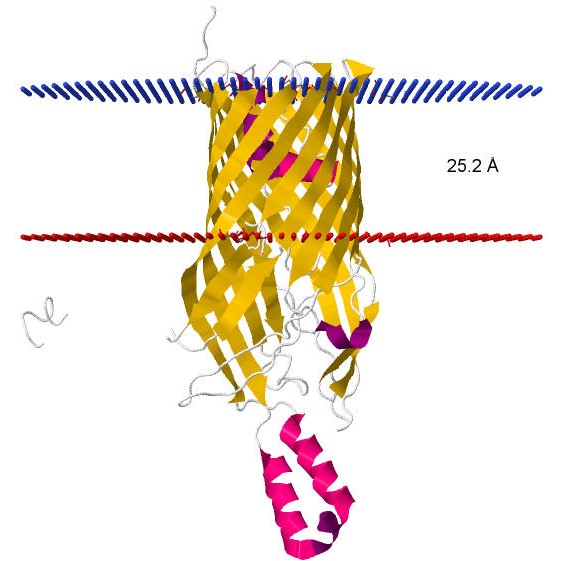
В качестве трансмембранного белка был выбран FadL (FADL_ECOLI, 3pgu) - транспортер длинных жирных кислот внешней мембраны E.coli. Что интересно, данный белок также служит рецептором, с которым связывается фаг T2 для проникновения в клетку. [1]
Трансмембранная часть этого белка представлена 14-ми β-тяжами из 8-ми аминокислотных остатков, формирующими так называемый β-бочонок.
По данным OPM гидрофобная часть этого белка имеет толщину 25.2 Å. По измерениям 3D-структуры в Jmol такая величина была получена как расстояние между сторонами мембраны. Между атомами трансмембранной части белка мною было найдено максимальное расстояние в 24.26 Å.
В базе данных указаны координаты трансмембранных сегментов: 1( 43- 52), 2( 78- 87), 3( 94- 101), 4( 126- 135), 5( 142- 149), 6( 207- 216), 7( 223- 229), 8( 274- 281), 9( 289- 296),10( 325- 333),11( 341- 348),12( 367- 376),13( 383- 390),14( 411- 421)
| Тип | Трансмембранный |
|---|---|
| Класс | Трансмембранный β-бочонок |
| Суперсемейство | FadL белок наружной мембраны |
| Семейство | Транспортеры жирных кислот FadL |
| Организм | Escherichia coli |
| Локализация | Внешняя мембрана Грам-отрицательных бактерий |
| Толщина гидрофобной части (Глубина) | 25.2 Å |
| Угол изгиба | 1° |
| Топология | с/ед А (N-конец внеклеточный) |
2. DeepTMHMM
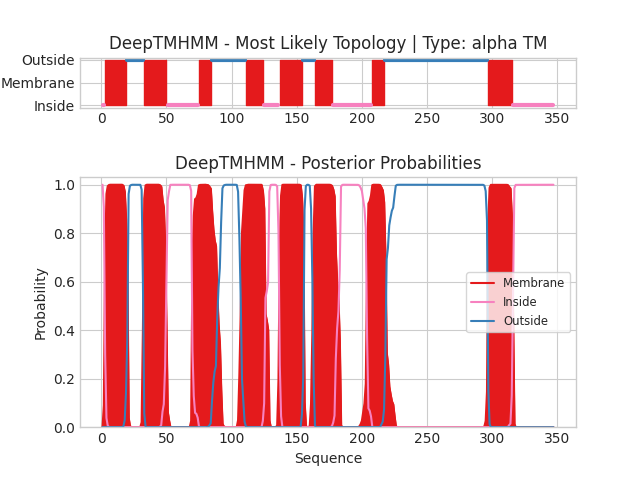
Белок(α): TCR_STRRM
Fasta-файл: seq
Выдача: файл
Обсуждение:
Сервис DeepTMHMM позволяет предсказывать трансмембранные участки по первичной структуре белка. В графической выдаче по горизонтали отложены координаты остатков белка, на вертикальной оси показаны предсказание положение определенных участков (верхний рисунок) и вероятность принадлежности конкретного остатка к той или иной топологии (нижний рисунок). Каждый цвет означает определенную локализацию участка: Membrane - трансмембранный, Inside - внутриклеточный, Outside - внеклеточный.
Для выданного белка было предсказано 8 трансмембранных участков (α-спиралей). N-конец белка согласно предсказанию является внутриклеточным, а С-конец - внеклеточным.
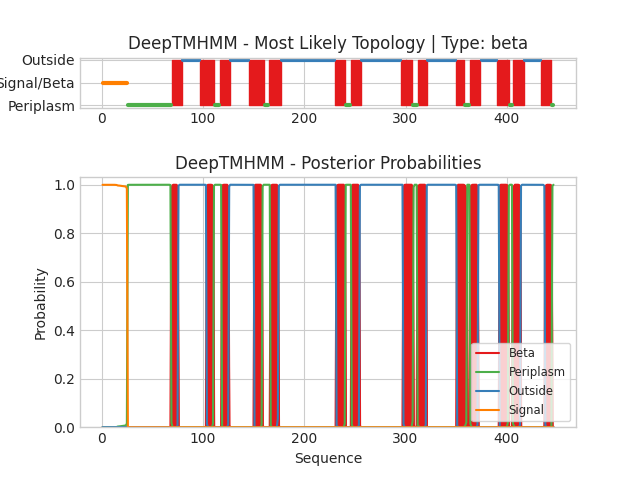
Белок(β): FADL_ECOLI
Fasta-файл: seq
Выдача: файл
Обсуждение:
Смысл полученных изображений и значение осей аналогичны выдаче для α-спирального белка. Каждый цвет означает определенную локализацию участка: Beta - бета-слой (мембранный), Periplasm - периплазматический (аналог внутриклеточной части), Outside - внеклеточный, Signal - сигнальный пептид для периплазматической локализации.
Для выданного белка было предсказано 14 трансмембранных участков (β-слоев). N-конец белка является сигнальным пептидом, за ним согласно предсказанию следует периплазматический участок, а С-конец - внеклеточным. Стоит сказать, что предсказания, на первый взгляд, не совпадают с OPM, однако в данной выдаче учитывается сигнальный пептид. Поэтому координаты всех предсказанных β-слоев как бы "смещаются" на его длину.
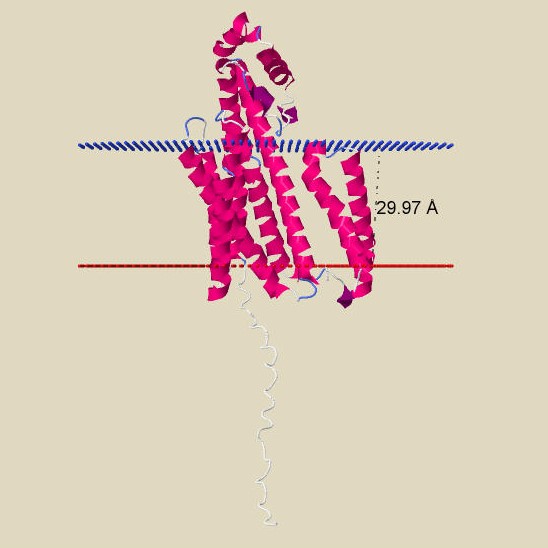
3. PPM
В данном разделе проведено предсказание топологии для выданного альфа-спирального белка TetB Streptomyces rimosus с помощью сервиса PPM 3.0 на сайте OPM.
Для запуска сервиса необходимо определиться с большим количеством параметров:
- Number of Membranes: 1
- Type of membrane: Gram-positive bacteria inner membrane (по данным Wikipedia Streptomycota окрашиваются по Граму)
- Allow curvature: no (чтобы не усложнять анализ выдачи)
- Topology (N-ter): out (по предсказанию DeepTMHMM)
Программе передавался файл с предсказанной структуры в формате pdb. Гетероатомы в рассмотрении не учитывались, так как этот белок их не содержит (по данным UniProt).
С полученной моделью можно ознакомиться по ссылке.
| Толщина гидрофобной части (Глубина) | 29.9 ± 1.1 Å |
|---|---|
| ΔGtransfer | -51.5 kcal/mol |
| Угол изгиба | 15 ± 0° |
| Трансмембранные сегменты | 1( 3- 27), 2( 31- 50), 3( 70- 92), 4( 105- 125), 5( 135- 155), 6( 161- 183), 7( 196- 218), 8( 297- 315) |
| Средняя длина трансмембранного сегмента | 22 остатка |
4. Сравнение алгоритмов предсказания
DeepTMHMM
Предсказанные трансмембранные участки:
- 3 - 20
- 33 - 50
- 70 - 88
- 108 - 128
- 137 - 155
- 163 - 183
- 204 - 218
- 297 - 315
PPM
Предсказанные трансмембранные участки:
- 3 - 27
- 31 - 50
- 70 - 92
- 105 - 125
- 135 - 155
- 161 - 183
- 196 - 218
- 297 - 315
Обсуждение
Можно с уверенностью сказать, что предсказания разных программ значительно похожи. Чаще всего, DeepTMHMM "пропускает" 2-3 остатка на N-концевой стороне участка, однако число и общее положение трансмембранных спиралей совпадают.
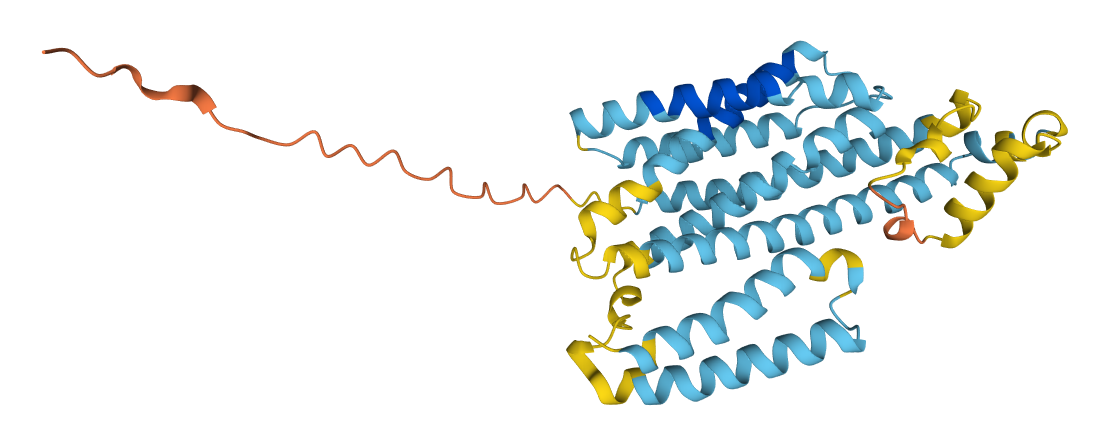
Стоит отметить, что положение каждого остатка в предсказанной с помощью AlphaFold структуры характеризуется своим доверительным score'ом (насколько точно верно предсказание). Что интересно, участки трансмембранных α-спиралей с относительно низким весом (70 > pLDDT > 50 - желтый цвет) как раз отличаются в вышеполученных предсказаниях.
В целом, можно считать, что структура предсказана успешно, так как в состав предсказанных α-спиралей (хоть и с низким весом) входят остатки, чье положение PPM считает трансмембранным, как и остатки, которые достоверно предсказаны хорошо.
5. База данных TCDB
TetB: в базе данных отстутствует. В UniProt я нашел его AC и ввел в поле поиска TCDB его.
FadL: запись имеется (link).
Данная запись содержит довольно подробную информацию о функциях, свойствах (массу, локализацию в клетке, принадлежность к организму и др.) белка, а также кросс-ссылки на другие базы данных. В данной записи присутствует ошибка, которая сразу бросается в глаза - указан только 1 трансмембранный участок (Number of TMSs).
Для трансмембранных белков-транспортеров существует своя классификация TC, подобная EC. Код для FadL - 1.B.9.1.1, где
- 1. - Каналы/поры
- .B. - β-бочонковые
- .9. - Семейство белка наружной мембраны FadL
- .1.1 - конкретный код данного белка
Стоит отметить, что суперсемейство белков 1.B.9.- называют суперсемейством пора-образующего белка I наружней мембраны (OMPPI).
References
- Black P. N. (1988). The fadL gene product of Escherichia coli is an outer membrane protein required for uptake of long-chain fatty acids and involved in sensitivity to bacteriophage T2. Journal of bacteriology, 170(6), 2850–2854. https://doi.org/10.1128/jb.170.6.2850-2854.1988