Практикум №9. Гомологичное моделирование комплекса белка с лигандом¶
1-2. Построение и модификация выравнивания¶
В программе Muscle было построено парное выравнивание аминокислотных последовательностей следующих белков: лизоцим форели (кристаллографическая структура известна) и лизоцим устрицы (LYS_OSTED, кристаллографическая структура неизвестна). Построенное выравнивание было модифицировано в соответствии с требованиями программы MODELLER. Был получен файл, в котором число точек после аминокислотной последовательности равняется числу лигандов белка.
3. Модификация файла с кристаллографической структурой лизоцима форели¶
В файле с кристаллографической структурой лизоцима форели была удалена вода и были модифицированы названия атомов лиганда. Был получен файл.
4. Создание и запуск управляющего скрипта¶
В практикуме №1 в структуре 1lmp было найдено семь водородных связей, образуемых между белком и лигандом:
- ND2:46:А - O1L:132:А
- N:59:A - O7:131:A
- NE1:63:A - O7:131:A
- NE1:63:A - O3:131:A
- ND2:103:А - O7:130:A
- O:107:A - N2:131:A
- N:109:A - O6:132:A
В соответствии с построенным выравниванием было предположено пять водородных связей между атомами лизоцима устрицы и лигандом:
- N:57:A - O7B:138:B
- NE1:61:A - O3B:138:B
- OG:96:A - O7A:138:B
- O:100:A - N2B:138:B
- N:102:A - O6C:138:B
Был создан скрипт для моделирования связывания лиганда лизоцимом устрицы. Созданный скрипт был запущен командой:
mod9v7 lys_osted.py &
Были получены пять моделей связывания лиганда лизоцимом устрицы:
6. Визуальный анализ моделей¶
Пять полученных моделей связывания лиганда лизоцимом устрицы были совмещены друг с другом и с известной кристаллографической структурой лизоцима форели (на Рисунке 1 показана зеленым). Все пять полученных моделей очень похожи между собой и немного отличаются от известной модели.
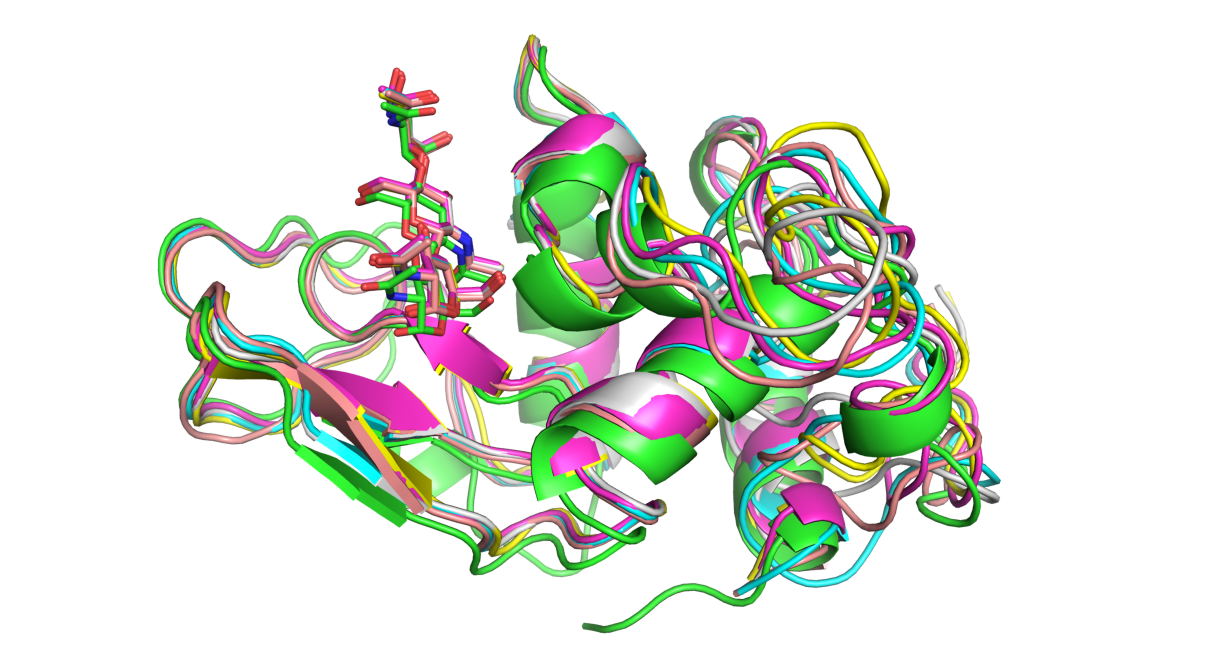
Рисунок 1. Совмещение пяти полученных моделей связывания лиганда лизоцимом устрицы друг с другом и с известной кристаллографической структурой лизоцима форели (показана зеленым).
7. Выбор наилучшей модели¶
С помощью веб сервиса WHAT IF была проведена проверка каждой из пяти полученных моделей связывания лиганда лизоцимом устрицы, в частности были получены такие результаты:
# Для первой модели
RMS Z-scores, should be close to 1.0:
Bond lengths : 0.992
Bond angles : 1.581
Omega angle restraints : 1.149
Side chain planarity : 0.293 (tight)
Improper dihedral distribution : 1.166
# Для второй модели
RMS Z-scores, should be close to 1.0:
Bond lengths : 0.991
Bond angles : 1.442
Omega angle restraints : 1.017
Side chain planarity : 0.230 (tight)
Improper dihedral distribution : 0.958
# Для третьей модели
RMS Z-scores, should be close to 1.0:
Bond lengths : 1.012
Bond angles : 1.553
Omega angle restraints : 1.139
Side chain planarity : 0.375 (tight)
Improper dihedral distribution : 1.140
# Для четвертой модели
RMS Z-scores, should be close to 1.0:
Bond lengths : 1.002
Bond angles : 1.538
Omega angle restraints : 1.134
Side chain planarity : 0.305 (tight)
Improper dihedral distribution : 1.100
# Для пятой модели
RMS Z-scores, should be close to 1.0:
Bond lengths : 1.011
Bond angles : 1.713
Omega angle restraints : 1.199
Side chain planarity : 0.314 (tight)
Improper dihedral distribution : 1.285
Возможно, наилучшей является вторая модель (на Рисунке 1 показана малиновым).