Комплексы ДНК-белок
При запуске в программе Jmol скрипт sets_selection последовательно выделяет шариками множества атомов: атомы кислорода 2'-дезоксирибозы, атомы кислорода фосфатов, атомы азота нуклеотидов.
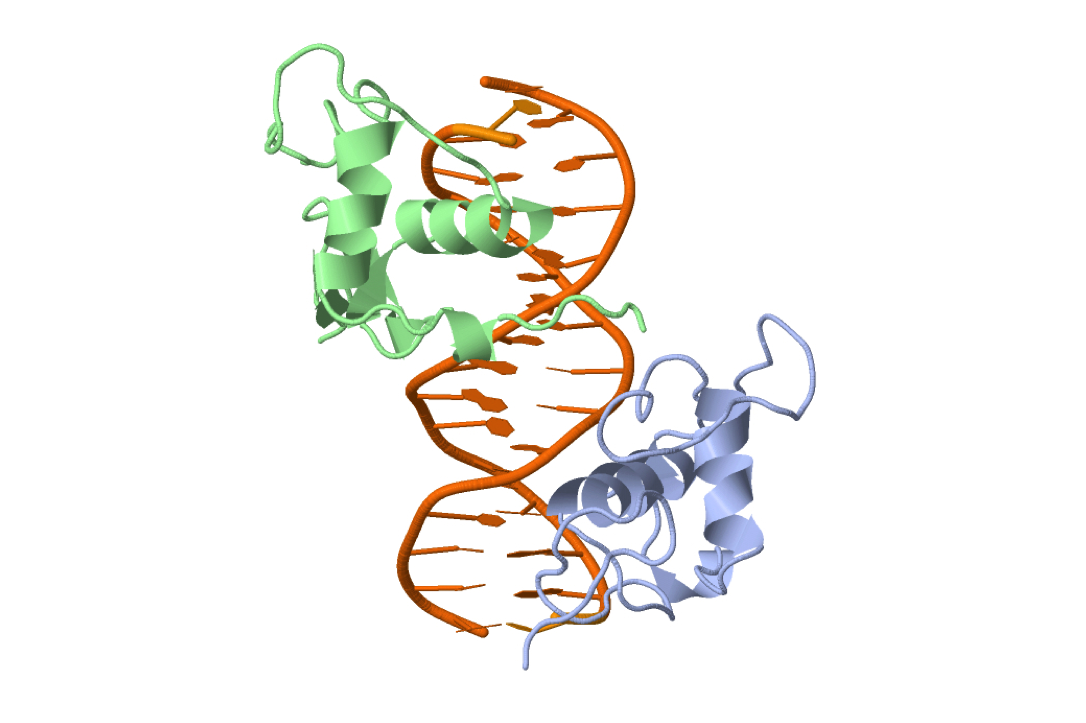
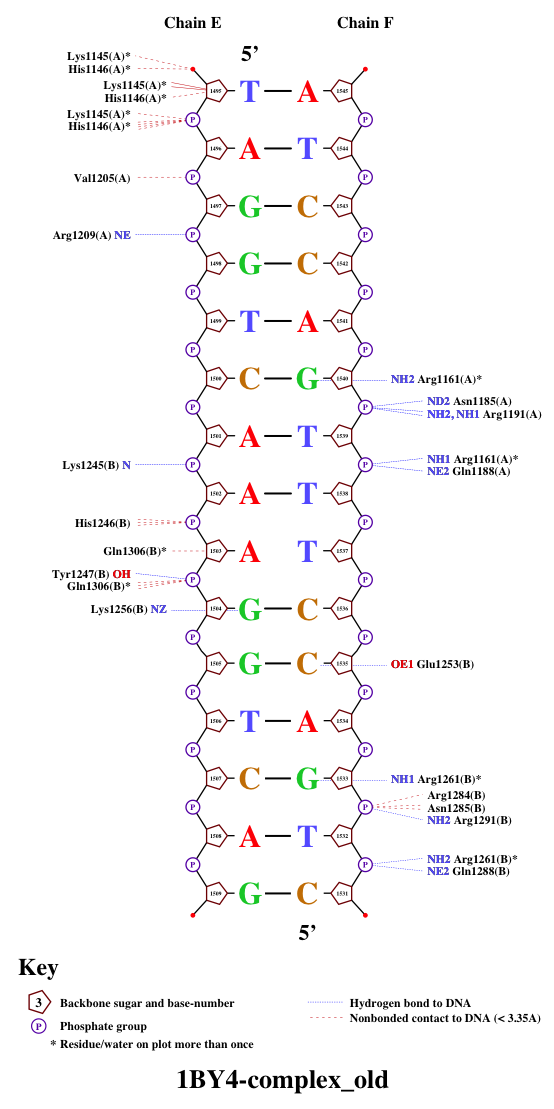
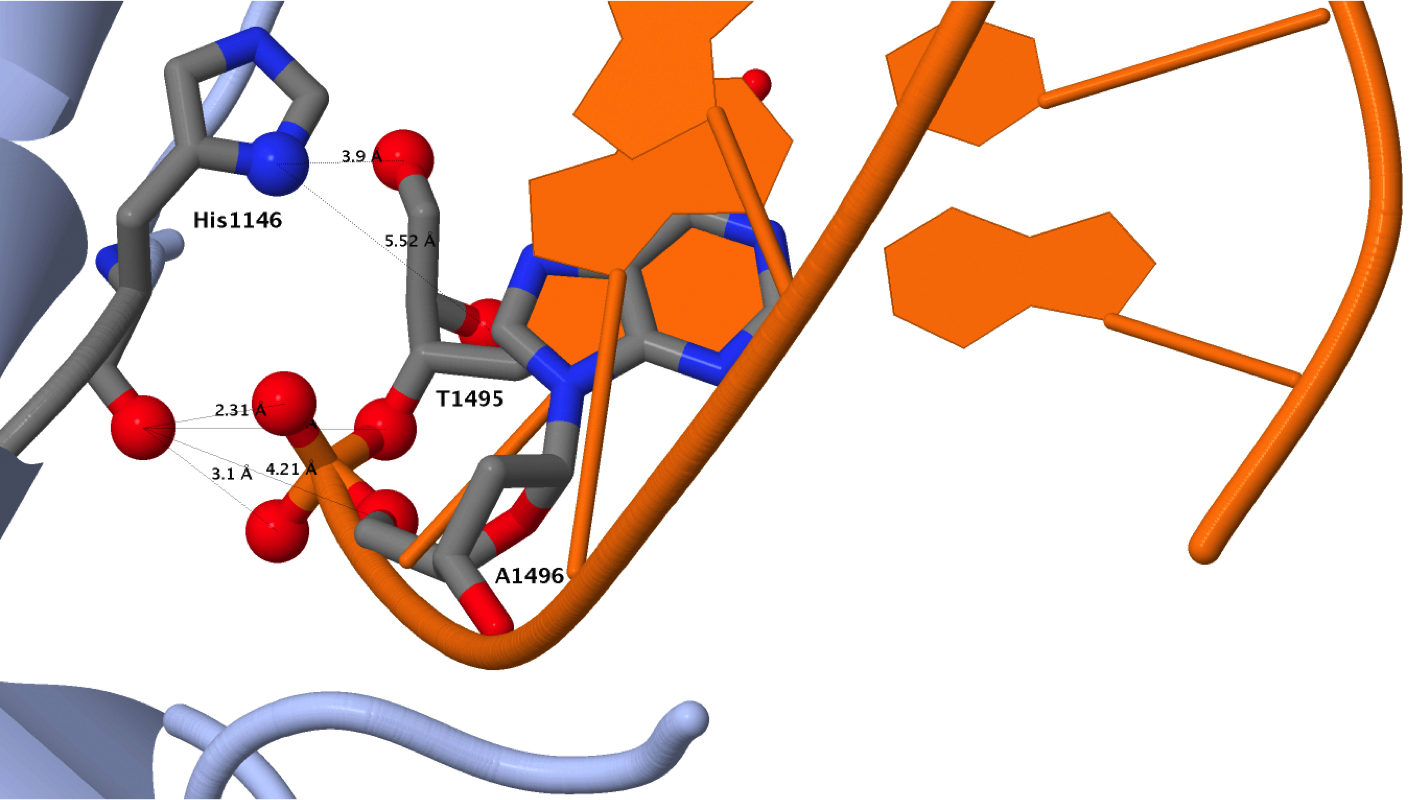
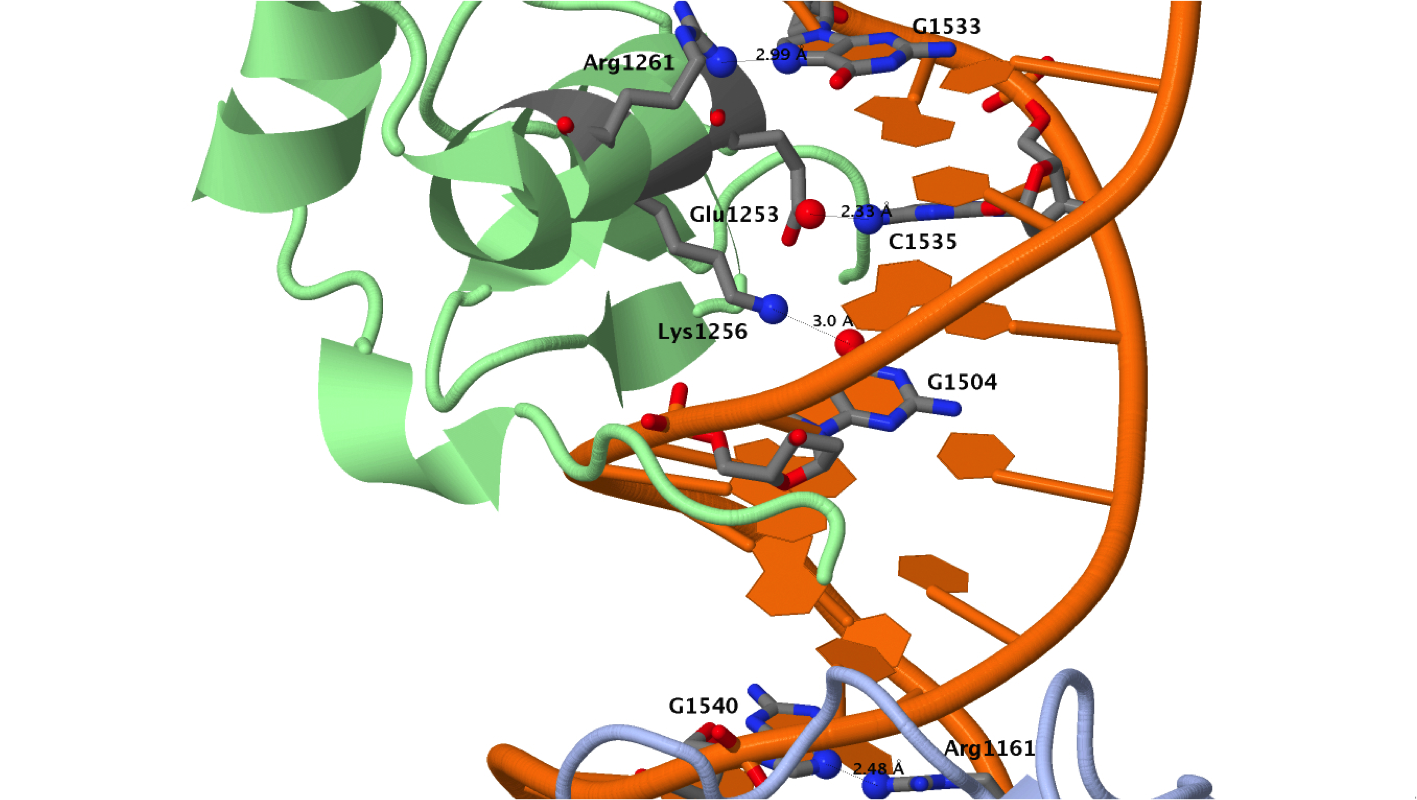
Контакты белка-рецептора ретиноевой кислоты RXR-alpha с В-ДНК:
| Полярные | Неполярные | Всего | |
| С атомами фосфорной кислоты | 24 | 18 | 42 |
| С атомами дезоксирибозы | 5 | 17 | 22 |
| С атомами азотистых оснований, обращенными к большой бороздке | 4 | 11 | 15 |
| С атомами азотистых оснований, обращенными к малой бороздке | 0 | 0 | 0 |
Можно заметить, что больше всего ДНК контактирует с белком атомами фосфорной кислоты, причём полярных контактов образуется немного больше, чем неполярных контактов. В два раза меньше ДНК контактирует с белком атомами дезоксирибозы, причём полярных контактов образуется примерно в три раза меньше, чем неполярных контактов. Число контактов с белком, образуемых атомами азотистых оснований ДНК, обращёнными к большой бороздке, невелико, причём полярных контактов образуется так же примерно в три раза меньше, чем неполярных контактов. Атомы азотистых оснований ДНК, обращённые к малой бороздке, вовсе не контактируют с белком.
Скачать файл 1BY4.pdb
Скачать файл 1BY4-complex.pdb
Скачать файл 1BY4-complex_old.pdb
(получен в программе remediator командой remediator --old 1BY4-complex.pdb > 1BY4-complex_old.pdb)
Скачать файл nucplot.ps
(получен в программе nucplot командой nucplot 1BY4-complex_old.pdb)
Предсказание вторичной структуры тРНК
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Просмотреть файл 2DXI-trna.fasta.txt
Просмотреть файл sequence.inv
(получен в программе einverted командой einverted 2DXI-trna.fasta.txt при запуске с параметрами:
Gap penalty: 5, Minimum score threshold: 10, Match score: 3, Mismatch score: -5)
Предсказание вторичной структуры тРНК по алгоритму Зукера
Просмотреть файл 2DXI-trna.ig.out
(получен в программе mfold командой mfold SEQ=2DXI-trna.fasta.txt P=0)
| Реальная структура тРНК | Структура тРНК, предсказанная с помощью программы einverted | Структура тРНК, предсказанная с по алгоритму Зукера | |
| Акцепторный стебель | 5'-501-572-3' 5'-502-571-3' 5'-503-570-3' 5'-504-569-3' 5'-505-568-3' 5'-506-567-3' 5'-507-566-3' (всего 7 пар) |
Предсказано 0 пар | Предсказано 7 пар |
| D-стебель | 5'-510-525-3' 5'-511-524-3' 5'-512-523-3' 5'-513-522-3' (всего 4 пары) |
Предсказано 2 пар | Предсказано 4 пар |
| Антикодоновый стебель | 5'-526-544-3' 5'-527-543-3' 5'-528-542-3' 5'-529-541-3' 5'-530-540-3' 5'-531-539-3' 5'-532-538-3' (всего 7 пар) |
Предсказано 5 пар | Предсказано 5 пар |
| T-стебель | 5'-549-565-3' 5'-550-564-3' 5'-551-563-3' 5'-552-562-3' 5'-553-561-3' (всего 5 пар) |
Предсказано 0 пар | Предсказано 5 пар |
| Число канонических пар нуклеотидов |
всего 19 канонических пар нуклеотидов | Предсказано 7 канонических пар нуклеотидов | Предсказано 18 канонических пар нуклеотидов |
Главная -> Семестры -> III семестр -> Комплексы ДНК-белок. Предсказание вторичной структуры тРНК.