Реконструкция филогенетического дерева по нуклеотидным последовательностям
Отобранные бактерии:
| Вид организма | Мнемоника | EMBL AC | Координаты 16S rRNA | Цепь |
| Bacillus subtilis | BACSU | D26185 | 73411..74963 | + |
| Clostridium tetani | CLOTE | AE015927 | complement(8715..10223) | - |
| Enterococcus faecalis | ENTFA | AE016830 | 248466..249987 | + |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255..60826 | + |
| Lactococcus lactis | LACLM | AM406671 | 511423..512971 | + |
| Listeria monocytogenes | LISMO | AL591974 | 37466..39020 | + |
| Staphylococcus epidermidis | STAES | AE015929 | complement(1598006..1599559) | - |
| Streptococcus pneumoniae | STRPN | AE005672 | 1353039..1354581 | + |
./blastn -task blastn -subject ae005672.entret -query am406671.fasta -out
ae005672.blast -evalue 0.001
Множественное выравнивание отобранных последовательностей 16S rRNA было построено в программе Muscle:
./muscle -in 16S_rRNA.fasta -out 16S_rRNA.aln
Сравнение правильного дерева с деревьями, реконструированными по нуклеотидным последовательностям:
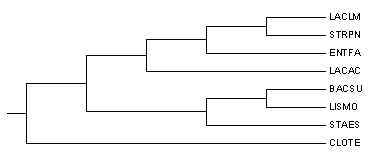
| Правильное дерево | |
 |
|
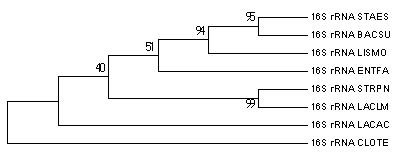
| Neighbour Joining, Bootstrap consensus tree, 500 Replications |
Maximum Likelihood, Bootstrap consensus tree, 500 Replications |
 |
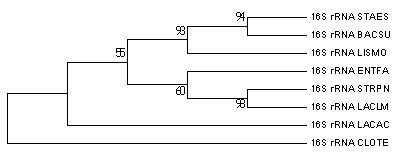 |
Деревья, реконструированные по нуклеотидным последовательностям, были укоренены так, чтобы Clostridium tetani (CLOTE) находился против клады, образованной таксоном Bacilli. Полученные укоренения не соответствуют правильному дереву.
Неправильные ветви дерева, реконструированного алгоритмом Neighbour Joining:
• Bacillus subtilis (BACSU) ближе к Staphylococcus epidermidis (STAES), чем к Listeria monocytogenes (LISMO)
(ветвь неправильная, но имеет хорошую поддержку, т.к. отличается от правильного дерева незначительно)
• Enterococcus faecalis (ENTFA) принадлежит таксону Bacillales, а не таксону Lactobacillales
(ветвь спорная, т.к. образуется лишь в 51% случаев)
• Lactobacillus acidophilus (LACAC) образует отдельную ветвь, а не принадлежит таксону Lactobacillales
(ветвь спорная, т.к. образуется лишь в 40% случаев)
Неправильные ветви дерева, реконструированного алгоритмом Maximum Likelihood:
• Bacillus subtilis (BACSU) ближе к Staphylococcus epidermidis (STAES), чем к Listeria monocytogenes (LISMO)
(ветвь неправильная, но имеет хорошую поддержку, т.к. отличается от правильного дерева незначительно)
• Lactobacillus acidophilus (LACAC) образует отдельную ветвь, а не принадлежит таксону Lactobacillales
(ветвь спорная, т.к. образуется лишь в 55% случаев)
Сравнение правильного дерева с деревьями, реконструированными по аминокислотным последовательностям и по нуклеотидным последовательностям:
Дерево, реконструированное по аминокислотным последовательностям, лучше соответствует правильному дереву, чем дерево, реконструированное по нуклеотидным последовательностям. Например, алгоритмом Neighbour Joining с использованием Bootstrap удалось реконструировать правильное дерево отобранных бактерий по аминокислотным последовательностям пептидил-тРНК гидролаз. По нуктеотидным последовательностям 16S rRNA не удалось реконструировать правильное дерево отобранных бактерий ни алгоритмом Neighbour Joining, ни алгоритмом Maximum Likelihood.
Реконструкция филогенетического дерева ортологов и паралогов
Отобранные бактерии:
| Вид организма | Мнемоника |
| Bacillus subtilis | BACSU |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Lactobacillus acidophilus | LACAC |
| Lactococcus lactis | LACLM |
| Listeria monocytogenes | LISMO |
| Staphylococcus epidermidis | STAES |
| Streptococcus pneumoniae | STRPN |
Множественное выравнивание гомологов CLPX_BACSU, найденных с e-value = 0.00001 среди белков отобранных бактерий, было построено в программе Muscle:
./muscle -in clpX.fasta -out clpX.aln
Дерево ортологов и паралогов, реконструированное по аминокислотным последовательностям:
| Neighbour Joining, Bootstrap consensus tree, 200 Replications |
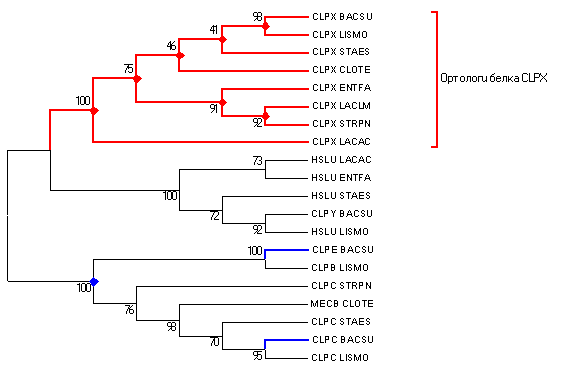 |
На реконструированном дереве красным цветом отмечены узлы, в которых произошло видообразование, синим цветом отмечен узел, в котором произошла дупликация гена и синим цветом отмечены ветви, соответствующие паралогам бактерии BACSU.