Поиск по сходству (нуклеотидный blast).
Задание 1. Определить таксономию и функцию прочтённой в практикуме 6 нуклеотидной последовательности.
Для начала была получена консенсусная последовательность из выравнивания (ae1.fasta) в практикуме 6
c помощью программы consambig; результат - consensus.fasta. Затем с помощью blastn были получены следующие результаты:
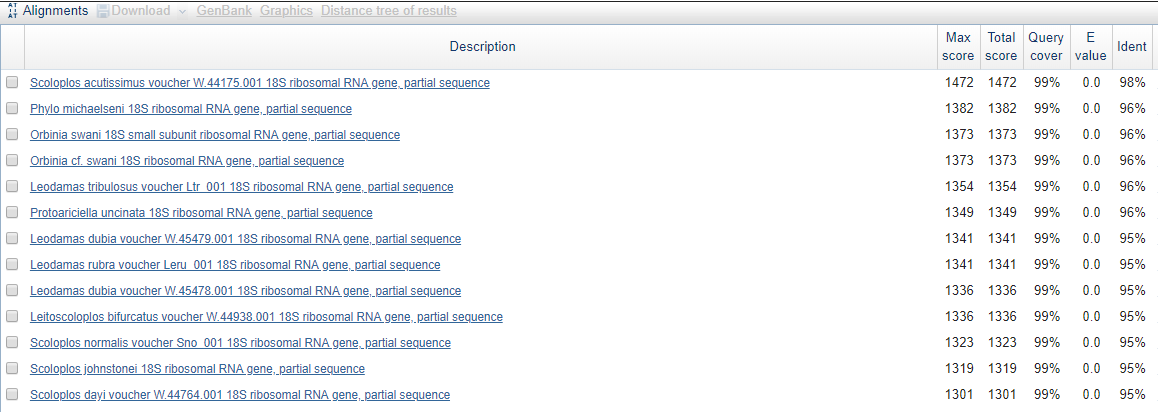
Отсюда видно, что наша последовательность - это часть последовательности гена 18S рибосомальной
РНК. Исходя из трех лучших находок можно предположить, что это ген из организма семейства
Orbiniidae:



Задание 2. Сравнение списков находок нуклеотидных последовательностей тремя разными
вариантами blast.
Для консенсусной последовательности из задания 1 были получены находки с помощью megablast,
blastn с параметрами по умолчанию и blastn с максимально чувствительными параметрами.
Для blastn с параметрами по умолчанию результат был получен ранее:

Для megablast результаты отличаются только чуть большим Max score для каждой соответствующей
находки:
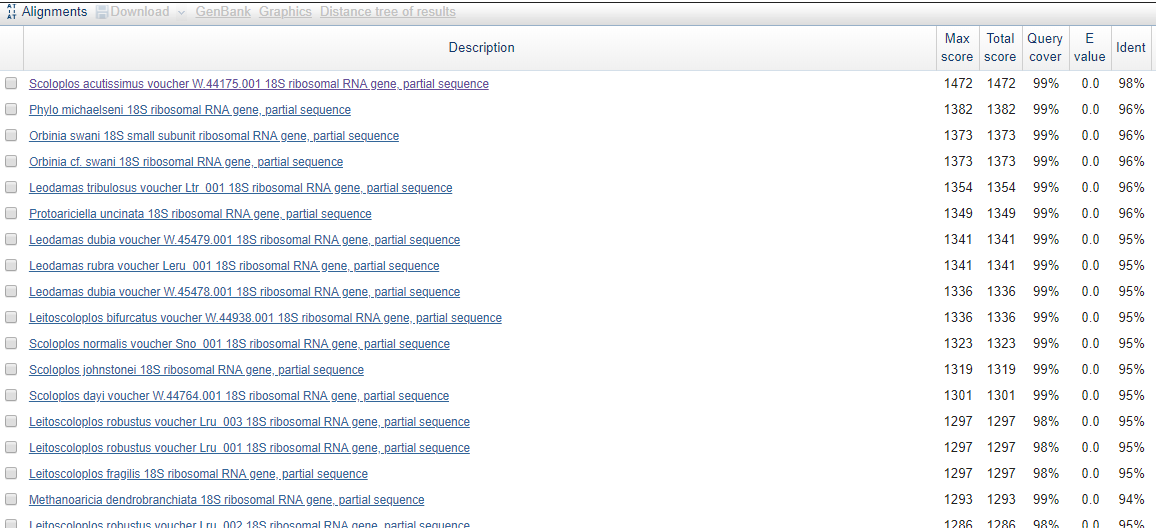
При изменении параметров blastn: word size с 11 на 7, Match/Mismatch Scores с 2,-3 на 1,-1 и
при ограничении поиска таксоном Orbiniidae, исключая Scoloplos, количество находок уменьшилось
с максимального 100 до 36, хотя кроме этого рода остальные полученные ранее остались, ранее
количесвто находок было максимальным:

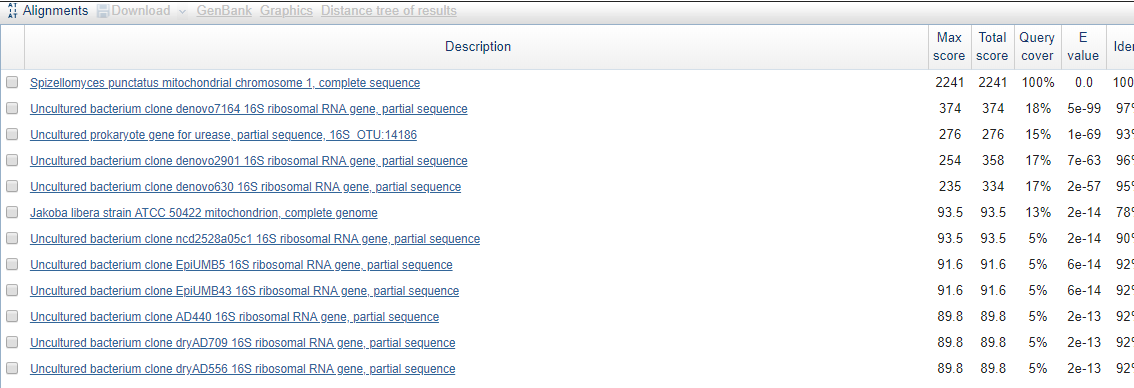
Далее была взята последовательность рРНК из митохондриального генома из предыдуущего
практикума seq.txt. По результатам megablast выявилось 100 находок, причем
на первом месте - из нашего митохондриального генома, оставшиеся были с намного меньшими Max
score и покрытием:
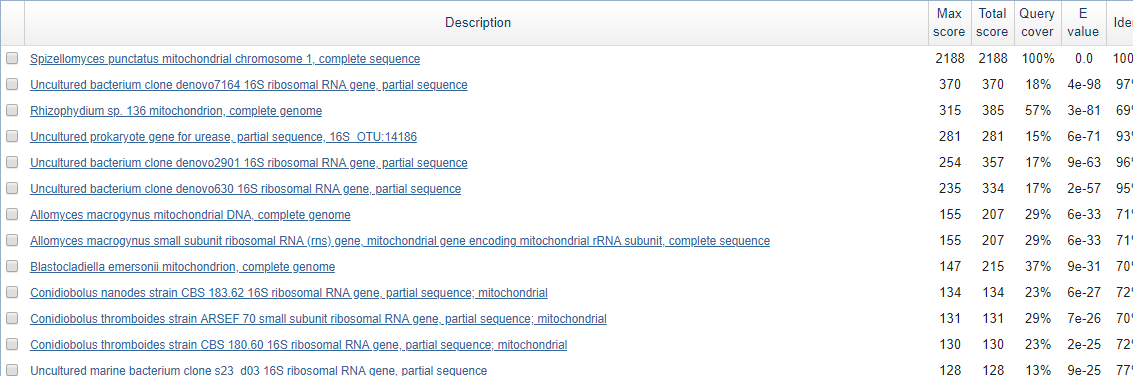
Для стандартного blastn количество находок такое же, причем лучшая
находка, что неудивительно, осталась прежней (только с чуть меньшим Max score), в остальном
результаты довольно схожи, но здесь вклинилась 3-я находка с много меньшим identity:
При изменении параметров blastn: Word size с 11 на 7, лучшая находка не изменилась, как и
их количество:
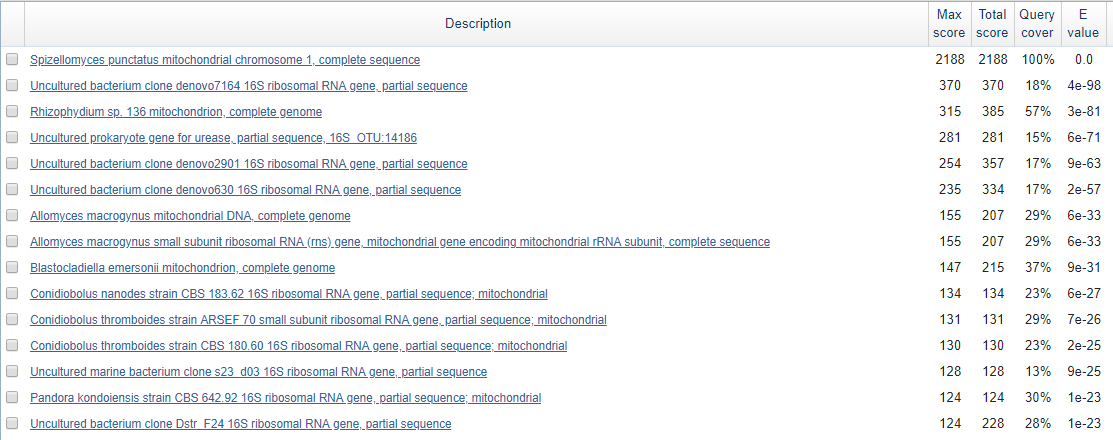
Хотя некоторые находки увеличились в покрытии:
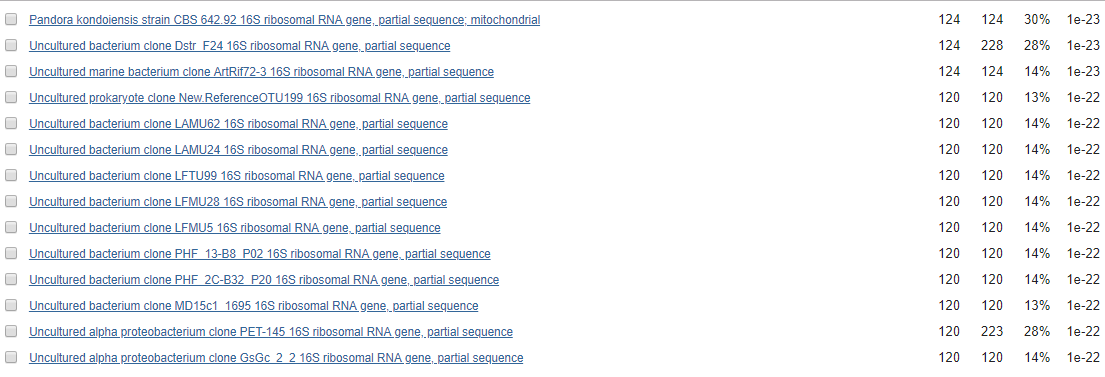
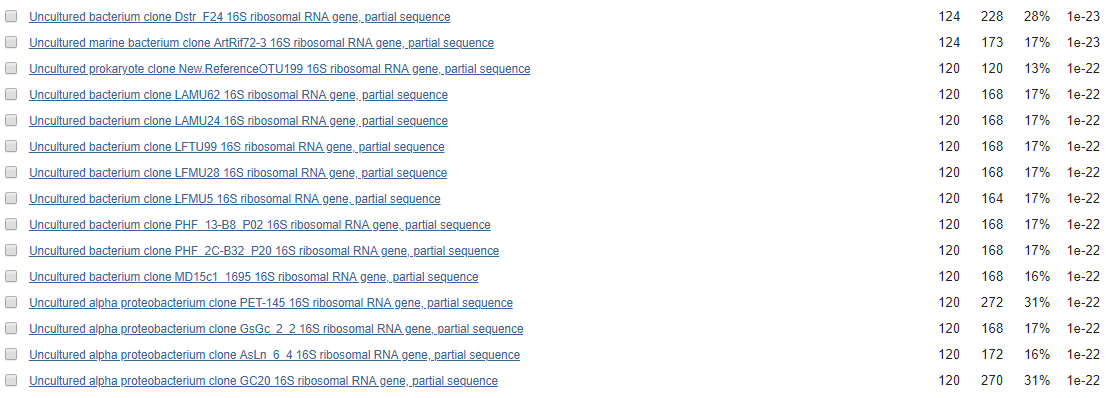
Из всех этих данных, особенно для второй последовательности, видно, что megablast выбирает только
максимально схожие последовательности и сделать blastn чувствительным довольно сложно.
Задание 3. Проверка наличия гомологов трех белков в неаннотированном геноме.
Для этого задания использовался неаннотированный геном организма Amoeboaphelidium protococcarum (
файл X5.fasta, на основе которого была создана локаальная база данных с
помощью команды:
makeblastdb -in X5.fasta -dbtype nucl
Поиск гомологов проводился для трех белков: TERT_SCHPO, TBB_NEUCR и EIF3G_SCHPO.
Для поиска гомологов применялся tblastn. Шаблон команды:
tblastn -query name.fasta -db X5.fasta > name.out
1. TERT_SCHPO
Это теломераза,восстанавливающая длину хромосомы при репликации; имеется у большинства
(но не всех) эукариот, и для нее лучшая находка оказалась scaffold-17 со Score(bits): 108 и
E-value: 1e-23. В общем в полученном выравнивании содержались лишь участки с низким
процентом идентичности (25%), из этого можно сделать вывод, что гомологии нет.
2. TBB_NEUCR
Это тубулин, белок, участвующий в образовании микротрубочек. Лучшая находка в геноме: scaffold-26
со Score(bits):693 и E-value:0.0. В полученном участке выравнивания процент идентичности очень
высок (87%), почти нет гэпов и показатель E-value равен нулю, отсюда можно сделать вывод, что
наблюдается гомология.
3. EIF3G_SCHPO
Это фактор инициации трансляции eIF3g, содержит также РНК связывающий домен. Здесь лучшая находка
scaffold-20 со Score(bits): 95.5 и E-value: 2e-21. В общем показатель Score был довольно низкий -
меньше 95.5, да и Identity меньше 38%, отсюда можно предположить, что гомология не наблюдается.
Задание 4. Поиск какого-нибудь гена белка в одном из контигов.
Был выбран контиг unplaced-307, последовательность которого была получена по формуле:
seqret <имя файла>:<имя последовательности> -out <имя выходного файла>
Так как надо было найти гомологичные последовательности белка по нуклеотидной последовательности
одного из контигов организма Amoeboaphelidium protococcarum,то использовался blastx.
Поиск осуществлялся в базе данных Reference proteins на сайте NCBI. Ставилось ограничение на
таксон Fungi, поскольку известно, что Amoeboaphelidium protococcarum - примитивный родственник
грибов. В итоге:
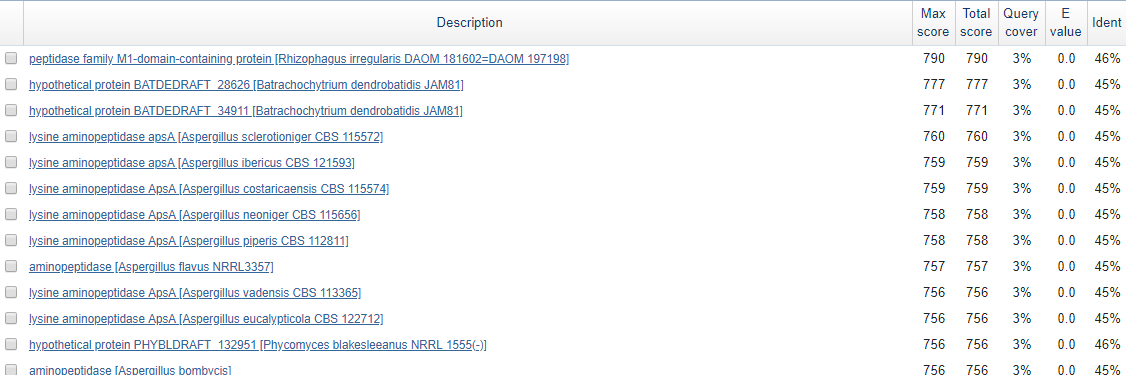
Здесь E-value находок равно 0.0, Max score довольно высокий и находки - это в основном белки
пептидазы, что позволяет сделать ввод, что в этом контиге есть ген наиболее часто возникшей
хорошей находки - лизина аминопептидазы. Аминопептидазы катализируют отщепление концевой
аминокислоты пептида, лизин аминопептидаза, соответственно, более специфична к реакции
отщепления концевых лизинов.
Задание 5. Карта локального сходства геномов двух бактерий.
Для этого задания были выбраны геномы двух бактерий, а точнее их 2-е хромосомы: Brucella ovis
(NC_009504.1) и Brucella abortus (NC_006933.1), и для них получена карта локального сходства
с помощью выравнивания megablast:
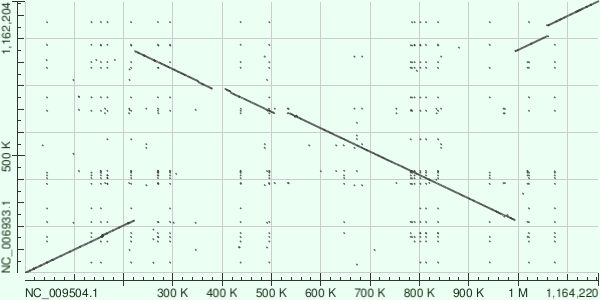
Здесь можно увидеть делецию в правом верхнем углу и инверсию посередине.
©Makarikova Olga 2018
















