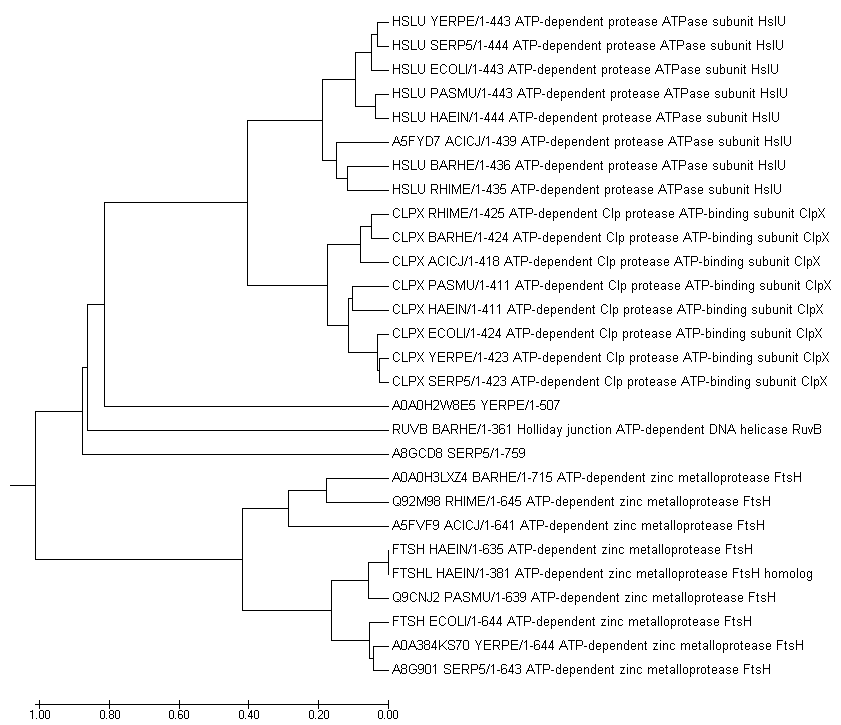
Паралоги
1) FTSH_HAEIN и FTSHL_HAEIN
2) CLPX_ECOLI и HSLU_ECOLI
3) CLPX_RHIME и HSLU_RHIME
4) CLPX_YERPE и HSLU_YERPE
Ортологи
1) HSLU_SERP5, HSLU_YERPE, HSLU_ECOLI, HSLU_HAEIN, HSLU_PASMU, HSLU_RHIME, HSLU_BARHE
2) CLPX_SERP5, CLPX_YERPE, CLPX_ECOLI, CLPX_HAEIN, CLPX_PASMU, CLPX_RHIME, CLPX_BARHE, CLPX_ACICJ
3) FTSH_HAEIN, FTSH_ECOLI
Дупликация гена
1) При образовании паралогов FTSH_HAEIN и FTSHL_HAEIN

2) При образовании паралогов CLPX_RHIME и HSLU_RHIME

Разделение путей эволюции белков в результате видообразования
1) При образовании ортологов HSLU_YERPE и HSLU_SERP5

2) При образовании ортологов HSLU_PASMU и HSLU_HAEIN

3) При образовании ортологов HSLU_BARHE и HSLU_RHIME

Комментарий: на картинках красным выделены ветви, разделение которых описывает события выше.
©Makarikova Olga 2019