Трансмембранные белки
Четвертый семестр
Трансмембранные белки
- а)
Структуры α-спиральных трансмембранных белков

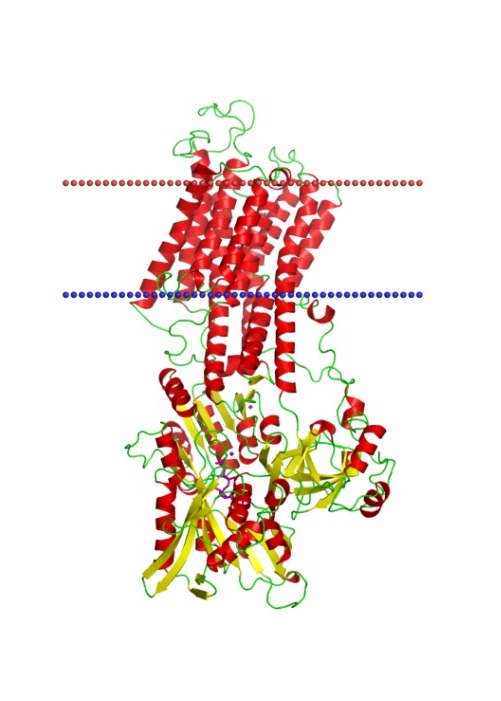
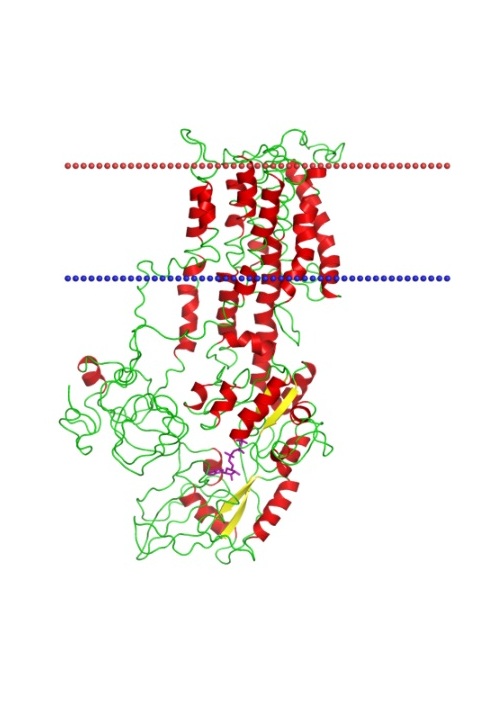
Dopamine D3 receptor,
Eukaryotic plasma membrane,
Homo sapiensCalcium ATPase, E1-ATP state,
Endoplasmic reticulum,
Oryctolagus cuniculusSodium-potassium pump, E2-Pi state,
Eukaryotic plasma membrane ,
Arabidopsis thalianaMitochondrial ADP/ATP carrier,
Mitochondrial inner membrane,
Bos taurusMethionine importer MetNI,
Bacterial gram-negative inner membrane ,
Escherichia coliСтруктуры трансмембранных β-баррелей

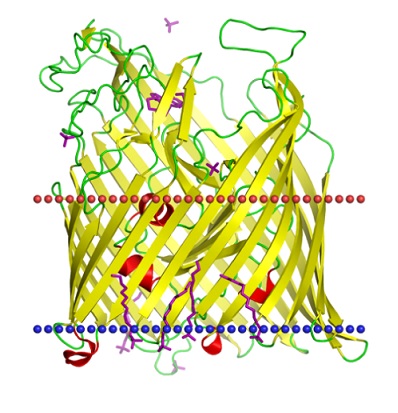
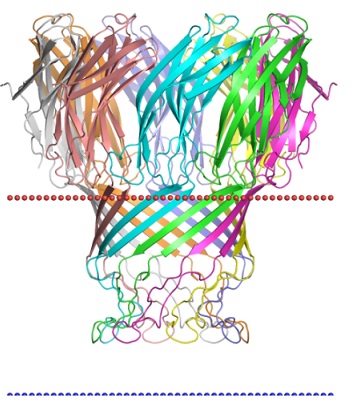
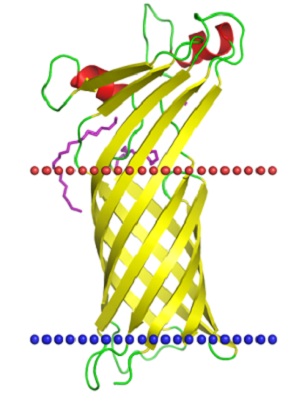
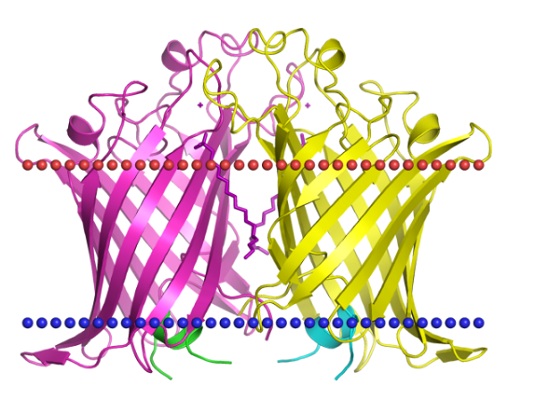
P pilus usher PapC translocation domain,
Bacterial gram-negative outer membrane,
Escherichia coliFe(III)-pyochelin receptor FptA,
Bacterial gram-negative outer membrane,
Pseudomonas aeruginosaPorin MspA,
Bacterial gram-positive acid-fast cell wall,
Mycobacterium smegmatisMajor outer membrane protein from Thermus thermophilus,
Bacterial gram-negative outer membrane,
Thermus thermophilusOuter membrane phospholipase A,
Bacterial gram-negative outer membrane,
Escherichia coli
б)Сравние топологии трансмембранных частей двух белков, с α-спиральными трансмембранными участками и с трансмембранными β-баррелями.
Выбор белков - произвольный из БД PDBTM .
PDB код Число цепей Тип
(спираль, баррель)Число трансмембранных участков в цепи Число остатков в одном трансмембранном участке
(типичное, минимальное, максимальное)(*) Толщина мембраны в ангстремах
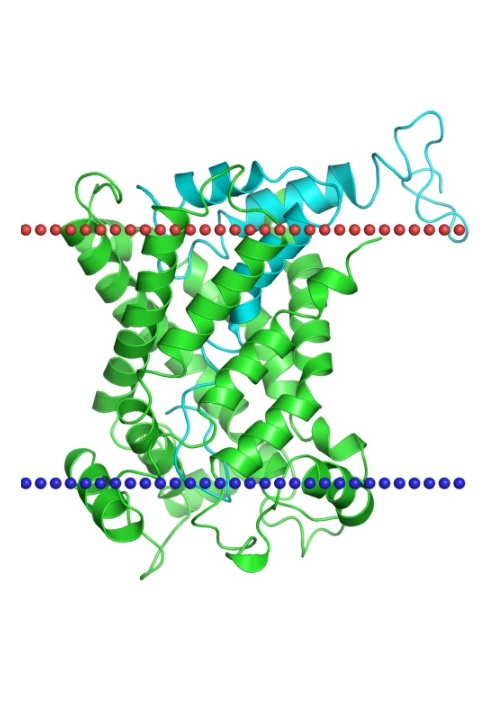
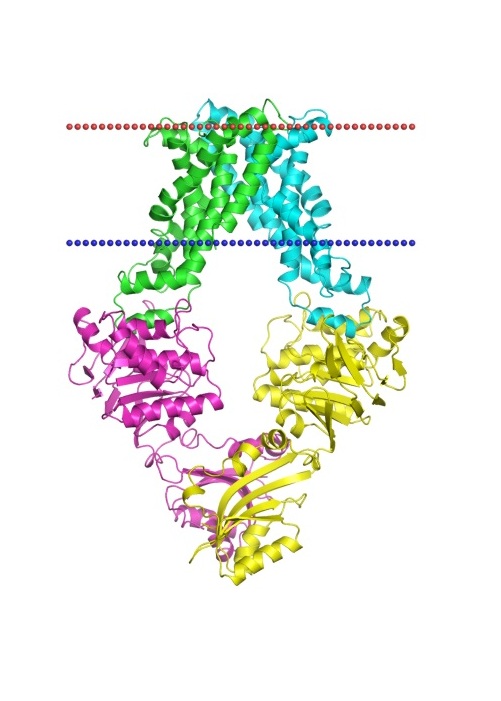
(расстояние между атомами на границах трансмембранных участковперпендикулярно мембране 2ahy 4 α-спиральный 3 22, 15 29.5 ±0.7Å 2jk4 1 &beta-баррель 19 7, 6, 10 23.4 ± 2.3 Å

2ahy, NaK potassium channel 2jk4, Voltage-dependent anion channel (VDAC) - Дан белок AC Uniprot Q5E9Z5
PDB ID гомолога - 2ZW3
UniprotID гомолога - CXB2_HUMAN
UniProt
По аннотациям записи Uniprot это белок-коннексон из Bos Taurus. По данным UniProt трансмембранные участки 23-45, 76 - 98, 132 - 154, 193 - 215.
Его гомолог CXB2_HUMAN - коннексон человека.TMHMM
Программа TMHMM предсказывает трансмембранные участки 23 - 45, 76 - 98, 132 - 154, 193 - 215 (что совпадает с данными в UniProt).
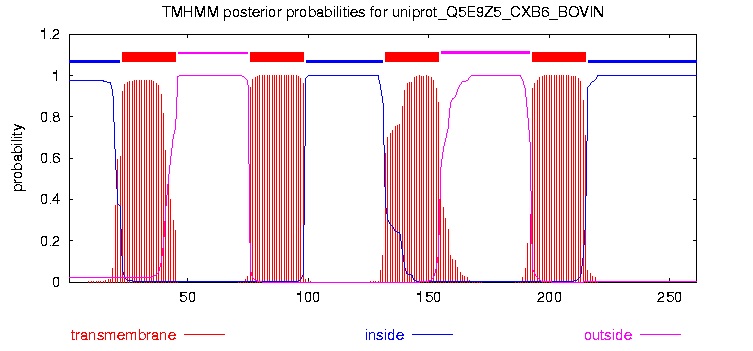
# CXB6_UniProt Length: 261 # CXB6_UniProt Number of predicted TMHs: 4 # CXB6_UniProt Exp number of AAs in TMHs: 89.60724 # CXB6_UniProt Exp number, first 60 AAs: 20.98675 # CXB6_UniProt Total prob of N-in: 0.97697 # CXB6_UniProt POSSIBLE N-term signal sequence CXB6_UniProt TMHMM2.0 inside 1 22 CXB6_UniProt TMHMM2.0 TMhelix 23 45 CXB6_UniProt TMHMM2.0 outside 46 75 CXB6_UniProt TMHMM2.0 TMhelix 76 98 CXB6_UniProt TMHMM2.0 inside 99 131 CXB6_UniProt TMHMM2.0 TMhelix 132 154 CXB6_UniProt TMHMM2.0 outside 155 192 CXB6_UniProt TMHMM2.0 TMhelix 193 215 CXB6_UniProt TMHMM2.0 inside 216 261
Сравнение с гомологом с известной пространственной структурой
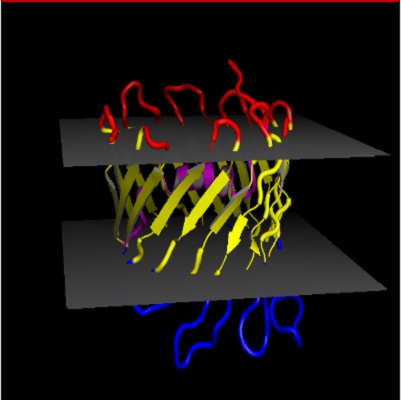
Cтруктура CXB2_HUMAN (2zw3) из PDBTM :
красным отмечены наружные участки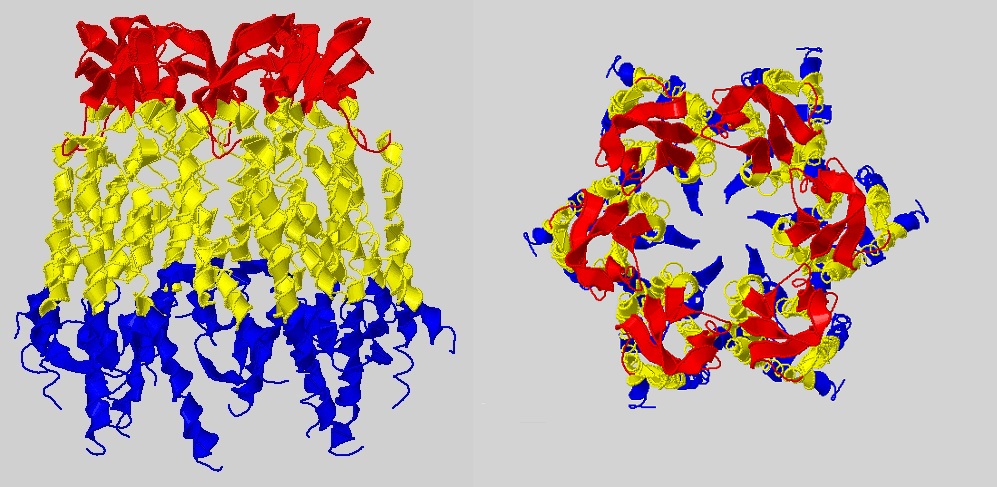
желтым отмечены трансмембранные участки
синим отмечены внутренние участки
Сравнением с несколькими гомологами по последовательности

membr.msf
Сравние предсказания TMHMM с предсказанием по структуре гомолога.
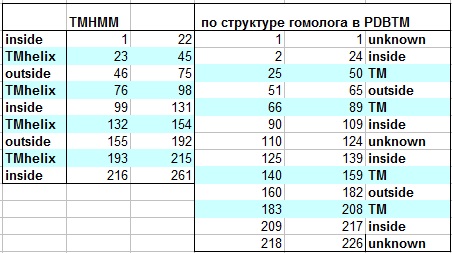
Считаем только до 226 позиции, т.к. длина гомолога - 226.
Число а.к. остатков Всего а.к. остатков в последовательности 261 (рассматриваем первые 226) Остатки, предсказанные TMHMM как локализованные в мембране (всего) 92 Правильно предсказали - совпадают с 3D предсказанием (true positives, TP) 66 Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным 3D таковыми не являются, false positives, FP) 26 Правильно не предсказали ( не предсказаны, и по данным 3D не находятся в мембране, true negatives, TN) 106 Не предсказали то, что нужно (остатки по данным 3D находятся в мембране, false negatives, FN) 28 Чувствительность (sensivity) = TP / (TP+FN) 70,2% Специфичность (specificity) = TN / (TN+FP) 79,1% Точность(precision) = TP /(TP+FP) 71,7% Сверхпредсказание = FP/ (FP+TP) 28,2% Недопредсказание = FN / (TN+FN) 20,9%
Проверка правила "positive inside" для трансмембранных белков.
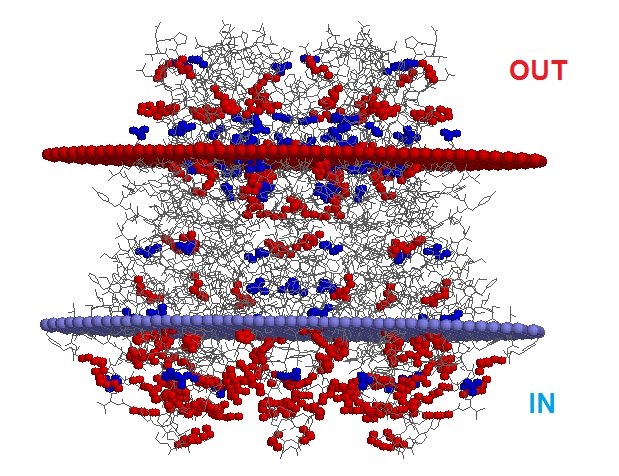
Правило "positive inside" выполняется для большинства положительно заряженных атомов (отмечены красным). Атомы, заряженные отрицательно, выделенны синим.
- (*) Найдите "код транспортера" данного вам белка в соответствии с БД TCDB
- Дан белок AC Uniprot Q5E9Z5