филогенетические деревья
Занятие 1.
На главную
Назад
Отобранные бактерии :
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Enterococcus faecalis | ENTFA |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus acidophilus | LACAC |
| Listeria monocytogenes | LISMO |
| Staphylococcus epidermidis | STAES |
| Thermoanaerobacter tengcongensis | THETN |
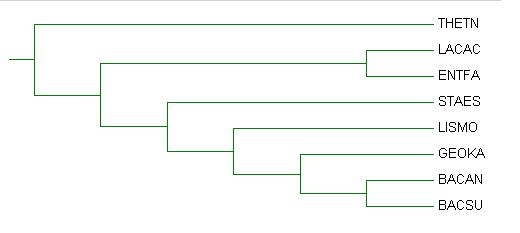
Скобочная формула дерева
(THETN,((LACAC,ENTFA),(STAES,(LISMO,(GEOKA,(BACAN,BACSU))))));
Изображение дерева
Ветви дерева
Дерево содержит 5 нетривиальныx ветвей:1). {LACAC, ENTFA} против {THETN, STAES, LISMO, GEOKA, BACAN, BACSU}
2). {STAES, LISMO, GEOKA, BACAN, BACSU} против {LACAC, ENTFA, THETN}
3). {LISMO, GEOKA, BACAN, BACSU} против {STAES, LACAC, ENTFA, THETN}
4). {GEOKA, BACAN, BACSU} против {STAES, LISMO, LACAC, ENTFA, THETN}
5). {BACAN, BACSU} против {GEOKA, STAES, LISMO, LACAC, ENTFA, THETN}
Занятие 2.
1). Таксономия выбраных бактерий:
| BACAN | Bacillus anthracis | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| BACSU | Bacillus subtilis | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| ENTFA | Enterococcus faecalis | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| GEOKA | Geobacillus kaustophilus | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| LACAC | Lactobacillus acidophilus | cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| LISMO | Listeria monocytogenes | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| STAES | Staphylococcus epidermidis | cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| THETN | Thermoanaerobacter tengcongensis | cellular organisms; Bacteria; Firmicutes; Clostridia; Thermoanaerobacterales; Thermoanaerobacteraceae; Caldanaerobacter; Caldanaerobacter subterraneus |
Clostridia и Bacilli разделяет {BACAN, BACSU, GEOKA, STAES, LISMO, LACAC, ENTFA}vs{THETN}
Lactobacillales выделяет {LACAC, ENTFA} vs{THETN, STAES, LISMO, GEOKA, BACAN, BACSU}
Bacillales выделяет {STAES, LISMO, GEOKA, BACAN, BACSU} vs {LACAC, ENTFA, THETN}
Bacillus cereus group выделяет {BACAN, BACSU} vs {GEOKA, STAES, LISMO, LACAC, ENTFA, THETN}
2).Для реконструкции филогенетического дерева был выбран рибосомный белок S12.
3).Выравнивание в формате FASTA :
aligned.fasta
4).Тоже самое, но с раскрашенными по таксонам консервативными позициями.

 - Clostridia;
- Clostridia;
 - Lactobacillales;
- Lactobacillales;
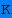 - Bacillales;
- Bacillales; - Bacillaceae;
- Bacillaceae;
 - Bacillus
- BacillusПример диагностических позиций :
L в 73 позиции встречается только у Lactobacillales
у всех Bacilli в позициях 21 и 25 S и N, а у Clostridia - A и Q.
5).Реконструкция дерева программой fprotpars.
Программа выдала одно дерево, которое не совпадает с правильным :
fprotpars - tree
+-----------------STAES
!
+--2 +--------------LISMO
! ! !
! ! ! +-----BACSU
! +--7 +-----4
! ! ! ! +--GEOKA
! ! ! +--5
1 +--3 +--BACAN
! !
! ! +--ENTFA
! +--------6
! +--LACAC
!
+--------------------THETN
((STAES,(LISMO,((BACSU,(GEOKA,BACAN)),(ENTFA,LACAC)))),THETN);
Неправильно построенные ветви :
ветвь {GEOKA,BACAN} vs {BACSU,STAES,LISMO,THETN,ENTFA,LACAC}ветвь {BACSU,GEOKA,BACAN,ENTFA,LACAC} vs {STAES,LISMO,THETN}
ветвь {LISMO, THETN, STAES} vs {LACAC, ENTFA, GEOKA, BACAN, BACSU}
Правильно построенные ветви :
ветвь {BACSU,GEOKA,BACAN} vs {BACSU,STAES,LISMO,THETN,ENTFA,LACAC}ветвь {LACAC, ENTFA} против {THETN, STAES, LISMO, GEOKA, BACAN, BACSU}
Отсутствующие правильные ветви :
{STAES, LISMO, GEOKA, BACAN, BACSU} vs {LACAC, ENTFA, THETN}{LISMO, GEOKA, BACAN, BACSU} vs {STAES, LACAC, ENTFA, THETN}
{BACAN, BACSU} vs {GEOKA, STAES, LISMO, LACAC, ENTFA, THETN}
6).Оценка эволюционных расстояния программой fprotdist.
матрица расстоянийУльтраметричность : из трёх расстояний между тремя объектами два всегда равны между собой и не меньше третьего d(A,B) >= d(B,C) то d(A,C) = d(A,B).
Наблюдается отклонение расстояний от ультаметричности : d(THETN,BACAN) = 0.287349, d(BACAN,GEOKA) = 0.121361, d(THETN,GEOKA) = 0.229253;Расстояния отклоняются от ультаметричности на 14,4% от среднего расстояния.
Аддитивность : если есть четыре последовательности A,B,C,D, то из трёх сумм d(A,B)+d(C,D) , d(A,C)+d(B,D) , d(A,D)+d(B,C) две равны между собой и больше третьей.
d(THETN,BACSU) = 0.280362 , d(THETN,BACAN) = 0.287349, d(THETN,GEOKA) = 0.229253;d(BACAN,GEOKA) = 0.121361, d(BACSU,GEOKA) = 0.090655, d(BACSU,BACAN) = 0.131405;
d(THETN,BACAN) + (BACSU,GEOKA) = 0.378004;
d(THETN,BACSU) + (BACAN,GEOKA) = 0.401723;
d(THETN,GEOKA) + (BACAN,BACSU) = 0.360658;
Расстояния отклоняются от аддитивности на 6,2% от среднего расстояния.
7).Реконструкция дерева программой fneighbor.
Алгоритм UPGMA выдаёт укоренённое дерево с длинами ветвей. Его можно применять, если справедлива гипотеза молекулярных часов (матрица расстояний не слишком далека от ультраметрической), а в данном случае это не так.(THETN, (STAES, (LACAC, (ENTFA, LISMO), (BACSU, GEOKA), BACAN)));
Алгоритм Neighbor-Joining выдаёт неукоренённое дерево с длинами ветвей; не предполагает молекулярных часов.
(GEOKA, ((LACAC, BACAN), (((STAES, ENTFA), LISMO), BACSU)), THETN);
Выводы :
Наиболее правильная реконструкция дерева получается с помощью программы fprotpars.Во всех реконструированных деревьях встречается ветвь {BACSU, GEOKA}vs{BACAN, ...} , хотя правильно
{BACSU, BACAN}vs{GEOKA, ...}. Вероятно, это связано с тем, что последовательности белка RS12 BACSU и GEOKA действительно более похожи, чем BACSU и BACAN. Данный пример демонстрирует, что для построения наиболее правильного филогенетического дерева нужно сравнивать как можно больше белков.

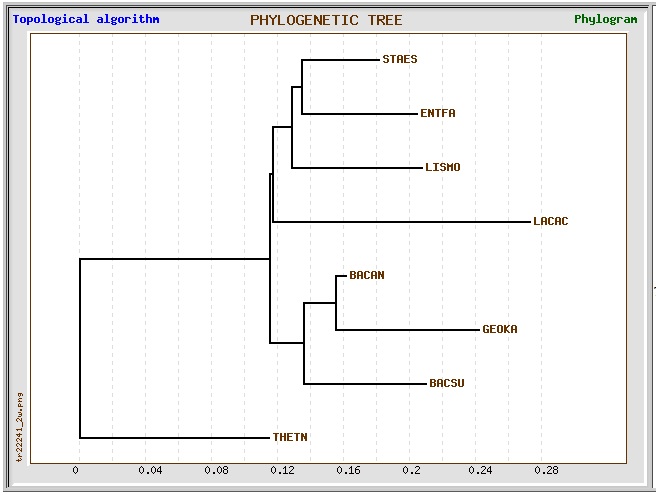
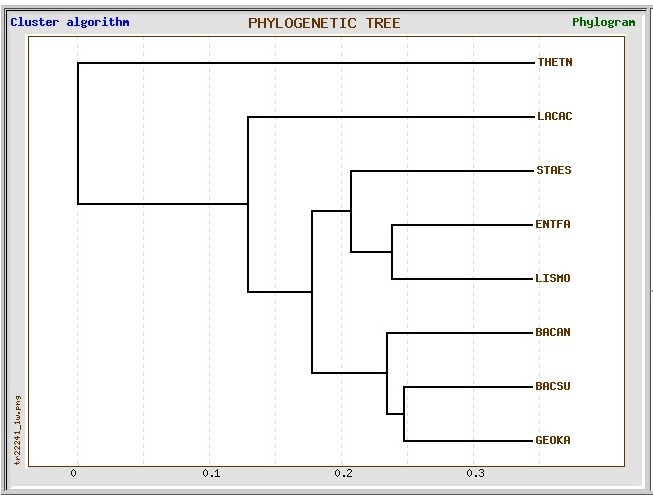
8).*TreeTop.
В результате были получены 2 дерева, построенные по кластерному и топологическому алгоритму.