Реконструкция филогении по нуклеотидным последовательнсотям. Анализ деревьев, содержащих паралоги.
Назад
Построение дерева по нуклеотидным последовательностям
Отобранные бактерии :
| Название | Мнемоника | AC | Координаты FT |
| Bacillus anthracis | BACAN | CP001598 | 9233..10741 |
| Bacillus subtilis | BACSU | AL009126 | 9810..11364 |
| Enterococcus faecalis | ENTFA | CP002621 | 213429..214977 |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421..11973 |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255..60826 |
| Listeria monocytogenes | LISMO | FM242711 | 234080..235590 |
| Staphylococcus epidermidis | STAES | CP000029 | 105734..107287 |
| Thermoanaerobacter tengcongensis | THETN | AE008691 | 53858..55384 |
1. Создание файла с последовательностью рРНК : seqret fasta::embl:CP001598[9233:10741]
2. Выравнивание с помощью muscle : muscle -in 16S.fasta -out 16S_al.fasta
3. По полученному выравниванию была построена матрица расстояний fdnadist :
4.С помощью fneighbor было построено дерево.(16s.fneighbor), (16s.tre)
+-GEOKA ! ! +-----LACAC ! ! 1-2 +--LISMO ! ! +-3 ! ! ! +--ENTFA ! +-4 ! ! +--STAES ! ! +-5 ! +-6 +BACAN ! ! ! +-BACSU ! +------THETN
Данное дерево отличается как от правильного, так и от построенного по белкам :
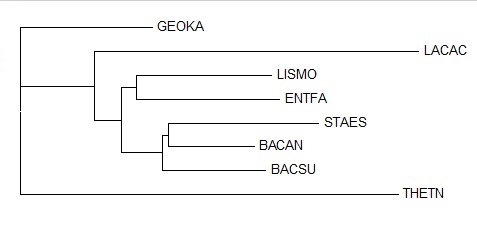 |
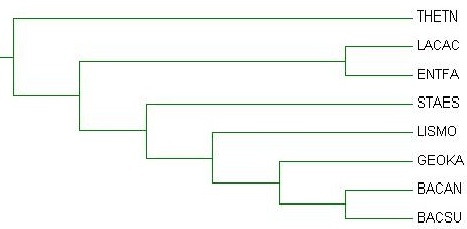 |
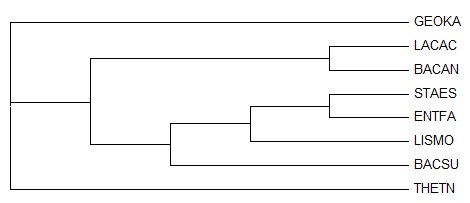 |
| по 16S рРНК | Правильное | по 12RS белкам |
| Правильное | по 12RS белкам |
Оба дерева сильно отличаются от правильного (нет совпадений по нетривиальным ветвям) и мало похожи между собой (совпадает только ветвь {THETN,GEOKA} против всех).
Построение и анализ дерева, содержащего паралоги
Исходное выравнивание, полученное с помощью muscle : clpx.fastaДалее с помощью fprotpars было построено дерево :
+--------------------------------------------NP_622291_THETN
!
! +-----CLPX_BACSU
! +--9
! ! ! +--NP_846917_BACAN
! +-----8 +-10
! ! ! +--YP_148505_GEOKA
! ! !
+--6 +--------------------------7 +--------NP_464793_LISMO
! ! ! !
! ! ! ! +--EFV89209_STAES
! ! ! +-----------5
! ! ! +--NP_764904_STAES
! ! !
! ! ! +--YP_147067_GEOKA
! ! ! +-17
! ! ! +-16 +--YP_0042076_BACSU
! +--4 ! !
! ! +----15 +-----ZP_0039411_BACAN
! ! ! !
+--2 ! +-14 +--------EFR84790_LISMO
! ! ! ! !
! ! ! ! ! +--EGG72872_STAES
! ! ! +----12 +----------13
! ! ! ! ! +--ZP_0661345_STAES
! ! ! ! !
! ! +----------------11 +-----------------NP_623065_THETN
1 ! !
! ! ! +--NP_815599_ENTFA
! ! +--------------------3
! ! +--ZP_0556575_ENTFA
! !
! +-----------------------------------------------YP_193739_LACAC
!
+--------------------------------------------------YP_0042919_LACAC
см. clpx.fprotparsПаралоги - гомологичные белки из одного организма :
NP_815599_ENTFA и ZP_0556575_ENTFA
YP_193739_LACAC и YP_0042919_LACAС
EGG72872_STAES и ZP_0661345_STAES
Ортологи - гомологичные белки из разных организмов :
CLPX_BACSU и NP_846917_BACAN
YP_147067_GEOKA и YP_0042076_BACSU