Практикум 14. Сборка de novo.
В практикуме была создана и проанализирована сборка чтений по проекту ILLUMINA-секвенирования SRR4240359 бактерии Buchnera aphidicola.
Подготовка чтений
Был создан файл с адаптерами секвенирования ILLUMINA на основе доступных адаптеров на сервере. Команда:
$ cat /P/y18/term3/block3/adapters/* > adapters.fa
На странице проекта секвенирования ILLUMINA был скачан fastq-файл по протоколу FTP. Затем программой trimmomatic были удалены возможные остатки адаптеров:
$ java -jar trimmomatic-0.30.jar SE -phred33 SRR4240359.fastq SRR4240359_trimmed.fastq ILLUMINACLIP:adapters.fa:2:7:7 TrimmomaticSE: Started with arguments: -phred33 SRR4240359.fastq SRR4240359_trimmed.fastq ILLUMINACLIP:adapters.fa:2:7:7 Using PrefixPair: 'AGATGTGTATAAGAGACAG' and 'AGATGTGTATAAGAGACAG' Using PrefixPair: 'TACACTCTTTCCCTACACGACGCTCTTCCGATCT' and 'GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT' Using Long Clipping Sequence: 'GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAG' Using Long Clipping Sequence: 'TTTTTTTTTTAATGATACGGCGACCACCGAGATCTACAC' Using Long Clipping Sequence: 'TCGTCGGCAGCGTCAGATGTGTATAAGAGACAG' Using Long Clipping Sequence: 'TTTTTTTTTTCAAGCAGAAGACGGCATACGA' Using Long Clipping Sequence: 'CTGTCTCTTATACACATCTGACGCTGCCGACGA' Using Long Clipping Sequence: 'AGATCGGAAGAGCTCGTATGCCGTCTTCTGCTTG' Using Long Clipping Sequence: 'AGATCGGAAGAGCACACGTCTGAACTCCAGTCAC' Using Long Clipping Sequence: 'TACACTCTTTCCCTACACGACGCTCTTCCGATCT' Using Long Clipping Sequence: 'GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT' Using Long Clipping Sequence: 'AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGTA' Using Long Clipping Sequence: 'AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGTAGATCTCGGTGGTCGCCGTATCATT' Using Long Clipping Sequence: 'AGATCGGAAGAGCGGTTCAGCAGGAATGCCGAG' Skipping duplicate Clipping Sequence: 'AGATCGGAAGAGCACACGTCTGAACTCCAGTCAC' Using Long Clipping Sequence: 'AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT' Using Long Clipping Sequence: 'AGATCGGAAGAGCGGTTCAGCAGGAATGCCGAGACCGATCTCGTATGCCGTCTTCTGCTTG' Using Long Clipping Sequence: 'CAAGCAGAAGACGGCATACGAGATCGGTCTCGGCATTCCTGCTGAACCGCTCTTCCGATCT' Skipping duplicate Clipping Sequence: 'AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGTA' Using Long Clipping Sequence: 'CTGTCTCTTATACACATCTCCGAGCCCACGAGAC' Using Long Clipping Sequence: 'AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT' ILLUMINACLIP: Using 2 prefix pairs, 17 forward/reverse sequences, 0 forward only sequences, 0 reverse only sequences Input Reads: 13557938 Surviving: 13502066 (99,59%) Dropped: 55872 (0,41%) TrimmomaticSE: Completed successfully
0.41% от числа всех ридов являлись предположительными адаптерами. Затем были удалены плохие буквы с концов чтений и оставлены чтения длиной не менее 32.
$ java -jar trimmomatic-0.30.jar SE -phred33 SRR4240359_trimmed.fastq SRR4240359_trimmed.fastq TRAILING:20 MINLEN:32 TrimmomaticSE: Started with arguments: -phred33 SRR4240359_trimmed.fastq SRR4240359_trimmed_2.fastq TRAILING:20 MINLEN:32 Input Reads: 13502066 Surviving: 12184080 (90,24%) Dropped: 1317986 (9,76%) TrimmomaticSE: Completed successfully
До триммирования размер fastq-файла с чтениями составлял 1375 Mb, а после 1223 Mb . Число чтений изменилось с 13557938 до 12184080, то есть осталось 89.87% от числа всех чтений.
Формирование сборки
Запуск программы velveth:
$ velveth Velveth_out 31 -fastq SRR4240359_trimmed_2.fastq -short [0.000000] Reading FastQ file SRR4240359_trimmed_2.fastq [28.622518] 12184080 reads found. [28.622555] Done [28.650216] Reading read set file Velveth_out/Sequences; [31.591929] 12184080 sequences found [45.959234] Done [45.959301] 12184080 sequences in total. [45.959457] Writing into roadmap file Velveth_out/Roadmaps... [51.465125] Inputting sequences... [51.467065] Inputting sequence 0 / 12184080 [54.279750] Inputting sequence 8000000 / 12184080 [54.522769] Inputting sequence 5000000 / 12184080 [54.819581] Inputting sequence 11000000 / 12184080 [55.147434] Inputting sequence 2000000 / 12184080 [59.244417] Inputting sequence 10000000 / 12184080 [59.476300] Inputting sequence 1000000 / 12184080 [60.090676] Inputting sequence 7000000 / 12184080 [60.292300] Inputting sequence 4000000 / 12184080 [64.781069] Inputting sequence 9000000 / 12184080 [64.807550] Inputting sequence 12000000 / 12184080 [65.345487] Inputting sequence 3000000 / 12184080 [65.879250] Inputting sequence 6000000 / 12184080 [66.802754] === Sequences loaded in 16.074920 s [66.804462] Done inputting sequences [66.804497] Destroying splay table [66.966899] Splay table destroyed
Запуск программы velvetg:
$ velvetg Velveth_out/ . . . . . Final graph has 694 nodes and n50 of 70607, max 125674, total 682206, using 0/12184080 reads
N50 оказалось равным 70607. Также был получен файл с основной информацией о контигах. Данные о 3 самых длинных контигах приведены в таблице 1.
ID контига |
Длина |
Покрытие |
Fasta-файл |
12 |
125674 |
44.550949 |
|
1 |
108447 |
42.009184 |
|
14 |
71403 |
39.411551 |
Анализ сборки
3 самых длинных контига были выровнены megablast с хромосомой бактерии Buchnera aphidicola (AC - CP009253). Число гэпов и однонуклеотидных различий есть в результатах выравниваний контигов с хромосомой: контиг 12, контиг 1, контиг 14. На рисунках 1, 2, 3 приведены карты локального сходства.
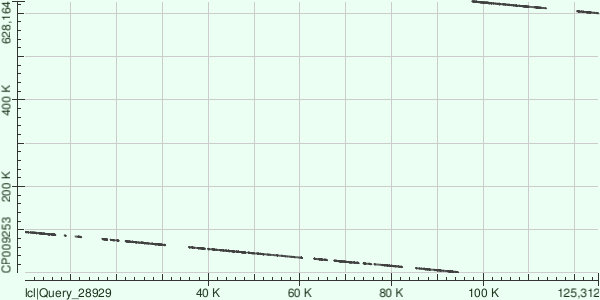
Рисунок 1. Контиг 12.
Обратное прочтение. 25 выравниваний. Координаты в хромосоме: 2004-94696 и 611229-627104.
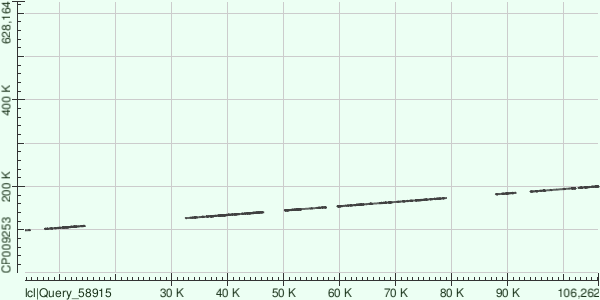
Рисунок 2. Контиг 1.
Прямое прочтение. 15 выравниваний. Координаты в хромосоме: 98408-200246.
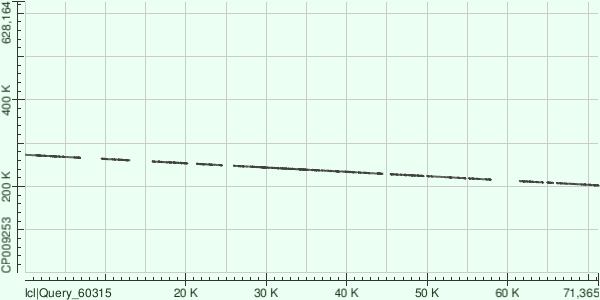
Рисунок 3. Контиг 14.
Обратное прочтение. 14 выравниваний. Координаты в хромосоме: 202390-273028.
Контиги ложились более-менее равномерно, визуальный разрыв 12 контига на карте локального сходства объясняется тем, что хромосома кольцевая. В целом можно сказать, что штамм из проекта секвенирования имел множественные индели, так как на рисунках 1-3 карты локального сходства не показывают непрерывного выравнивания контигов с хромосомой, насколько об этом позволяет утверждать информация о трёх самых длинных контигах. Для более точной информации следует анализировать остальные контиги.