Практикум 12. Трансмембранные белки
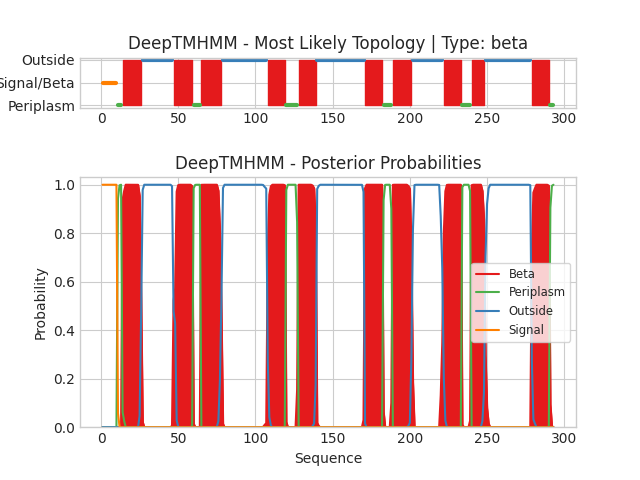
Для работы был выдан альфа-спиральный белок YVBV_BACSU.
Последовательность выданного белка в формате FASTA
Предсказание трехмерной структуры
База данных OPM
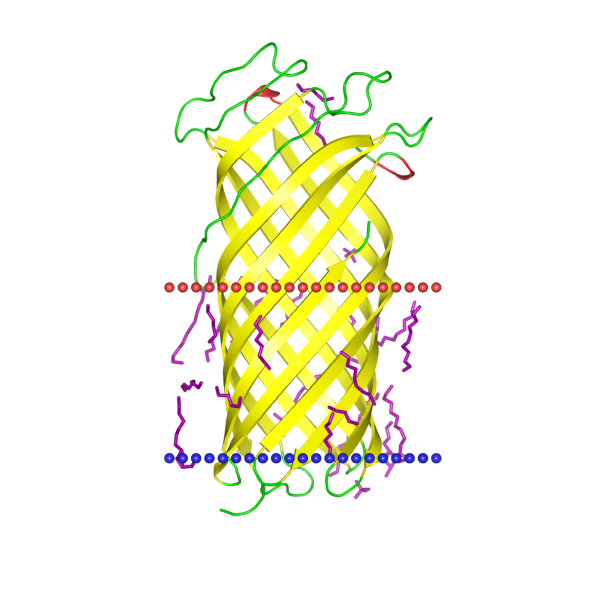
В базе данных OPM я выбрала трансмембранный белок, содержащий бета-бочонок из 10 трансмембранных бета-листов. Это активатор плазминогена PLA грамотрицательной бактерии Yersinia pestis. Интересно, что он является протеазой наружной мембраны (омптин), которая важна для вирулентности чумы. Его каталитическая функция заключается в том, что он превращает человеческий Glu-плазминоген в плазмин путем расщепления пептидной связи 560-Arg-|-Val-561, а также расщепляет аргинильные связи в других белках.
Идентификатор PDB: 2X55
Идентификатор Uniprot: COLY_YERPE
| Толщина гидрофобной части белка в мембране | 25.6 Å |
|---|---|
| Координаты трансмембранных участков | 1(13-22),2(51-60),3(65-73),4(111-121),5(126-136),6(174-183),7(188-197),8(224-235),9(239-248),10(282-292) |
| Среднее количество остатков в одном β-тяже белка | 9.4 |
| Мембрана, в которую встроен белок | Внешняя мембрана грамотрицательных бактерий |
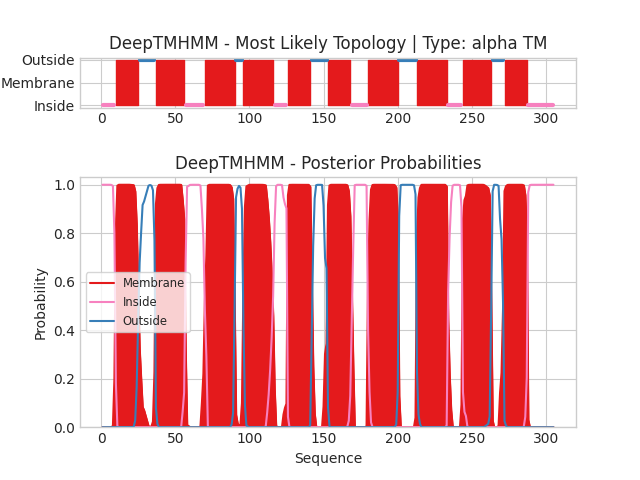
2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
С помощью сервиса DeepTMHMM были предсказаны положение альфа-спирального YVBV_BACSU и бета-листовогого COLY_YERPE.
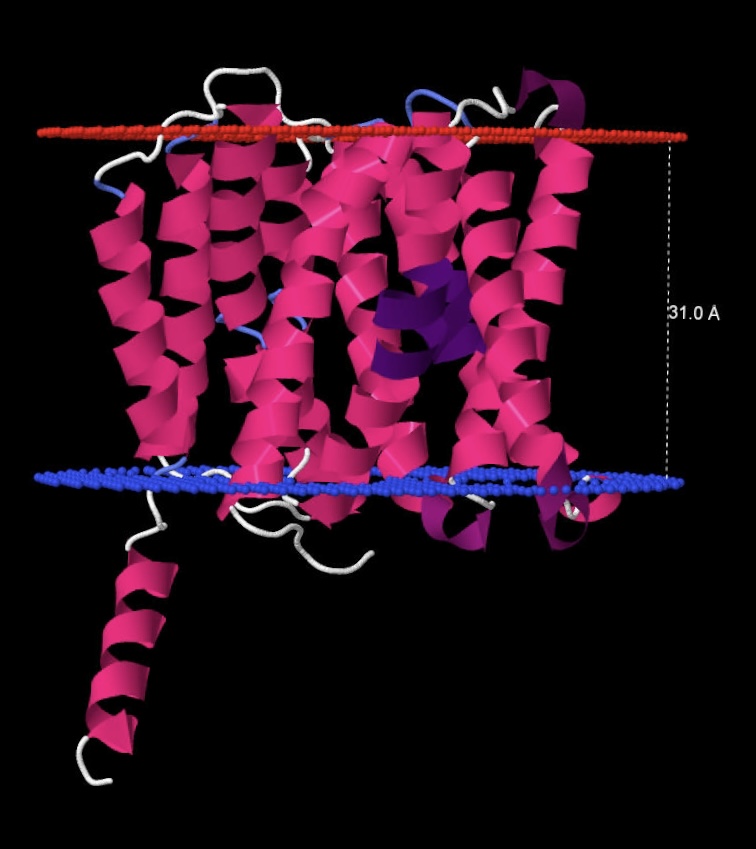
3. PPM: Предсказание положения выданного белка в мембране
Алгоритм PMM былы запущен для альфа-спирального белка YVBV_BACSU со следующими параметрами:
- Number of Membranes: 1
- Type of membrane: Gram-positive bacteria inner membrane
- Allow curvature: no
- Topology (N-ter): in (согласно результату DeepTMHMM N-конец внутри)
| Толщина гидрофобной части белка в мембране | 31.0 ± 2.0 Å |
|---|---|
| Координаты трансмембранных участков | 1( 7- 31), 2( 36- 57), 3( 67- 90), 4( 95- 116), 5( 123- 143), 6( 150- 171), 7( 179- 202), 8( 211- 237), 9( 241- 262),10( 269- 286) |
| Среднее количество остатков в одном β-тяже белка | 21.7 |
| Мембрана, в которую встроен белок | Внутренняя мембрана грамотрицательных бактерий |