Практикум 4
Составление списка гомологичных белков, включающих паралоги
Поиск гомолога белка CLPX_ECOLI осуществлялся среди отобранных бактерий:
| Название | Мнемоника |
|---|---|
| Bifidobacterium longum | BIFLO |
| Corynebacterium diphtheriae | CORDI |
| Corynebacterium efficiens | COREF |
| Leifsonia xyli | LEIXX |
| Mycobacterium leprae | MYCLE |
| Rubrobacter xylanophilus | RUBXD |
| Streptomyces avermitilis | STRAW |
Протеомы были объединены в файл all.fasta командой
cat ./proteomes/*.fasta > all.fasta
Поиск гомологов с помощью blastp был осуществлен командами:
makeblastdb -dbtype prot -in all.fasta
blastp -query CLPX_ECOLI.fasta -evalue 0.001 -outfmt 7 -db all.fasta 1>> out.txt
В результате были получены следующие 24 находки:
| CLPX_STRAW | ATP-dependent Clp protease ATP-binding subunit ClpX |
|---|---|
| Q1AVT0_RUBXD | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_MYCLE | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_COREF | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_CORDI | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_LEIXX | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLPX_BIFLO | ATP-dependent Clp protease ATP-binding subunit ClpX |
| Q1AU05_RUBXD | ATPase AAA-2 |
| Q8G871_BIFLO | Protease |
| Q8FMH5_COREF | Putative endopeptidase Clp ATP-binding chain C |
| Q6NFB1_CORDI | ATP-dependent Clp protease ATP-binding subunit |
| Q1AY82_RUBXD | ATPase AAA-2 |
| RUVB_BIFLO | Holliday junction branch migration complex subunit RuvB |
| Q6ACQ0_LEIXX | ATP-dependent zinc metalloprotease FtsH |
| Q8G3S2_BIFLO | ATP-dependent zinc metalloprotease FtsH |
| Q82QV8_STRAW | Putative AAA family ATPase |
| Q82EE9_STRAW | ATP-dependent zinc metalloprotease FtsH |
| Q82EB8_STRAW | Putative ATP-dependent Clp protease |
| FTSH_RUBXD | ATP-dependent zinc metalloprotease FtsH |
| Q6NF92_CORDI | ATP-dependent zinc metalloprotease FtsH |
| FTSH_MYCLE | ATP-dependent zinc metalloprotease FtsH |
| CLPC_MYCLE | Probable ATP-dependent Clp protease ATP-binding subunit |
| Q6NGK1_CORDI | AAA+ ATPase domain-containing protein |
| Q8FMG2_COREF | ATP-dependent zinc metalloprotease FtsH |
2. Реконструкция и визуализация
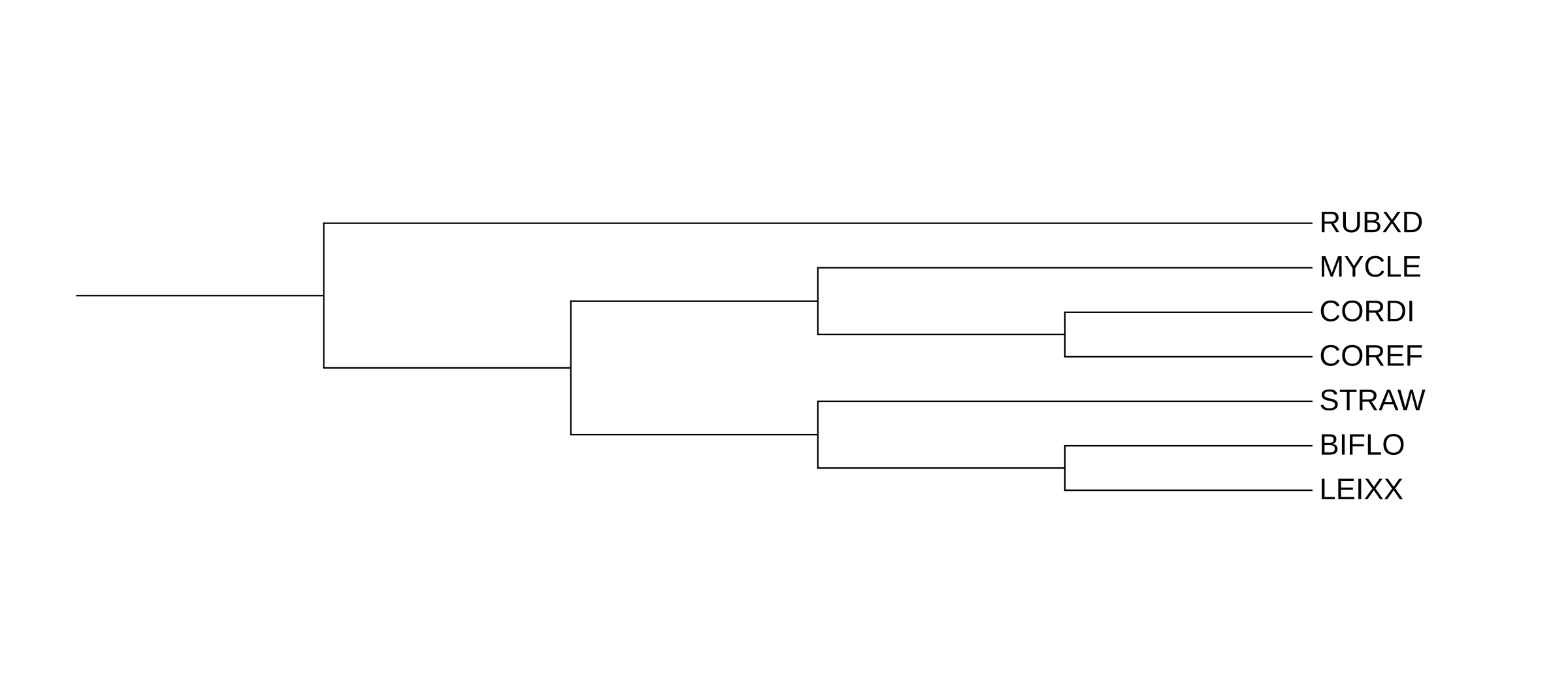
Все последовательности находок были объединены в fasta-файл и по нему было реконструировано дерево найденных гомологов программой FastME c параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик.
Полученное дерево в Newick формате.
Полученное дерево было переукренено в среднюю точку. Также были добавлены значения бутстреп‑поддержки.
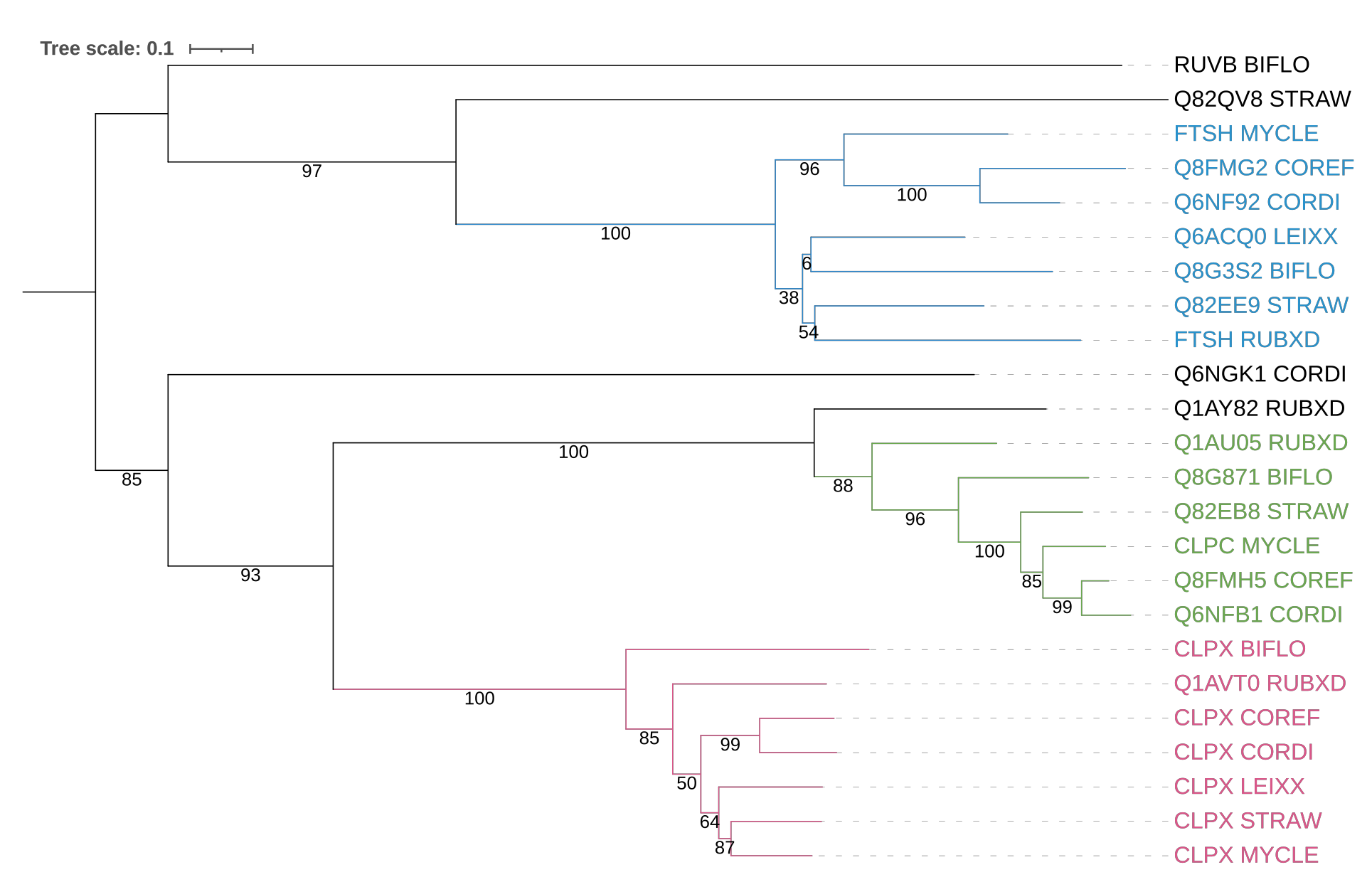
Ортологи в полученном дереве:
- CLPX_STRAW и CLPX_MYCLE
- Q6ACQ0_LEIXX и Q8G3S2_BIFLO
- Q8FMG2_COREF и Q6NF92_CORDI
Паралоги в полученном дереве:
- Q8G3S2_BIFLO и CLPX_BIFLO
- Q1AVT0_RUBXD и FTSH_RUBXD
- CLPX_COREF и Q8FMG2_COREF
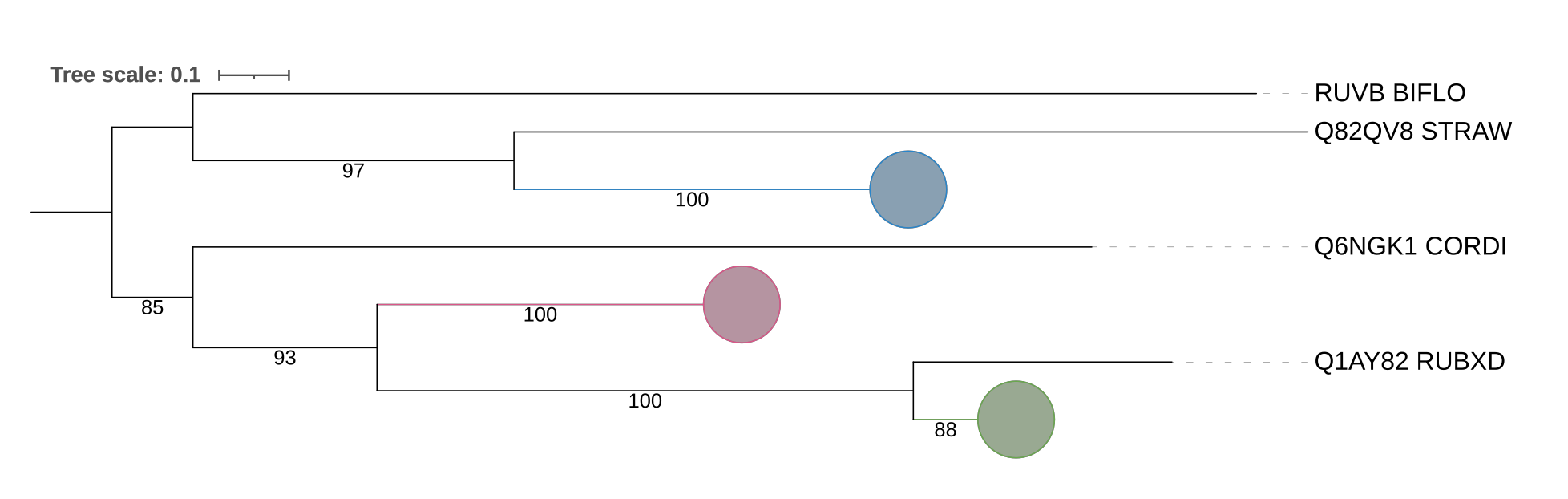
Вся голубая клада представлена цинковыми металлопротеиназами FtsH. По топологии все ветви соответсвуют эталонному дереву видов, хотя поддержка ветвей не самая высокая из всех клад.
В зеленой кладе часть белков являются протеинкиназами и один, содержащие АТФазный домен ААА-2. Если переукоренить, то совпадение с эталоном тоже будет полным, что совпадает с высокой поддержкой ветвей клады.
Розовая клада вся представлена АТФ-связывающими субъединицами ClpX протеиназ семейства Clp. Тут с эталоном вообще не совпадает, хотя поддержка ветвей достаточно высокая.