Практикум 8. Сигналы и мотивы - 3
1. Консервативный мотив в выравнивании последовательностей гомологичных белков
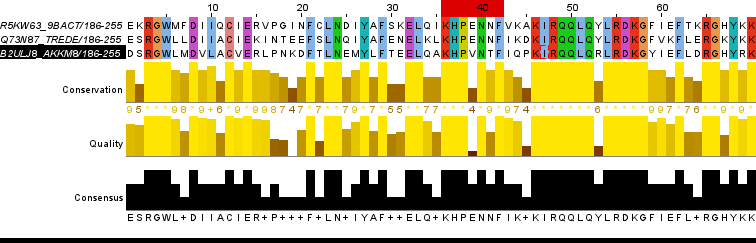
Было взято семейтсво эндонуклеаз DpnI_C (PF17726) с seed=13. Был выбран консервативный по всем последовательностям мотив K-I-[RS]-Q-Q-L-Q-X(1)-[LA]-R-D в выравнивании 13 последовательностей.
С помощью MyHits было плучено 2 находки базе данных SwissProt и они обе пренадежат к семейству DpnI_C.
2. Поиск мотива, специфичного для клады
3. PSI-BLAST
Был выбран белок с идентификатором C4Z088. Это ингибитор клеточного деления Eubacterium eligens ATCC 27750
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 162 | Q2P036.1 | 0.004 | Q5F5V4.1 | 0.005 |
| 2 | 188 | O25693.2 | 2e-09 | - | - |
| 3 | 188 | Q9ZM51.1 | 1e-11 | A7H8E6.1 | 0.012 |
| 4 | 188 | Q9ZM51.1 | 5e-13 | A7H8E6.1 | 0.016 |
Начиная с третьей итерации, число находок с E-value меньше 0.005 прекратило изменяться и отличие E-value лучшей неправильной от худшей правильной значительно, а значит полученная группа хорошо обособлена. Значит данное семейство хорошо обоснованно схотсвом последовательностей его белков.
Проверка числа ТА в бактериальном геноме
Была взята бактерия Croceicoccus marinus и ее референсный геном GCF_014211955.1. После работы скрипта было найдено ожидаемое число сайтов TA в полном геноме, равное 66470 и реальное значение числа сайта TA в полном геноме, равное 32929. Отношение реального к ожидаемому составляет 60%. P-value фактически оказывается нулевым, то есть часто теоритической встречаемости TA не равна реальной.