Практикум 4. Colabfold
Задание Deletion (D11)
Для выполнения практикума были даны две последовательности (seqA, seqB).
seqA MDAPEEEDHVLVLRKSNFAEALAAHKYLLVEFYAPWCGHCKALAPEYAKAAGKLKAEGSEIRLAKVDATEESDLAQQYGVRGYPTIKFFRNGDTASPKEYTAGREADDIVNWLKKRT GPAATTLPDGAAAESLVESSEVAVIGFFKDVESDSAKQFLQAAEAIDDIPFGITSNSDVFSKYQLDKDGVVLFKKFDEGRNNFEGEVTKENLLDFIKHNQLPLVIEFTEQTAPKIFG GEIKTHILLFLPKSVSDYDGKLSNFKTAAESFKGKILFIFIDSDHTDNQRILEFFGLKKEECPAVRLITLEEEMTKYKPESEELTAERITEFCHRFLEGKIKPHLMSQELPEDWDKQ PVKVLVGKNFEDVAFDEKKNVFVEFYAPWCGHCKQLAPIWDKLGETYKDHENIVIAKMDSTANEVEAVKVHSFPTLKFFPASADRTVIDYNGERTLDGFKKFLESGGQDGAGDDDDL EDLEEAEEPDMEEDDDQKAVKDEL
seqB MDAPEEEDHVLVLRKSNFAEALAAHKYLLVEFYAPWGHCKALAPEYAKAAGKLKAEGSEIRLAKVDATEESDLAQQYGVRGYPTIKFFRNGDTASPKEYTAGREADDIVNWLKKRTG PAATTLPDGAAAESLVESSEVAVIGFFKDVESDSAKQFLQAAEAIDDIPFGITSNSDVFSKYQLDKDGVVLFKKFDEGRNNFEGEVTKENLLDFIKHNQLPLVIEFTEQTAPKIFGGE IKTHILLFLPKSVSDYDGKLSNFKTAAESFKGKILFIFIDSDHTDNQRILEFFGLKKEECPAVRLITLEEEMTKYKPESEELTAERITEFCHRFLEGKIKPHLMSQELPEDWDKQPVK VLVGKNFEDVAFDEKKNVFVEFYAPWCGHCKQLAPIWDKLGETYKDHENIVIAKMDSTANEVEAVKVHSFPTLKFFPASADRTVIDYNGERTLDGFKKFLESGGQDGAGDDDDLEDLE EAEEPDMEEDDDQKAVKDEL
Отличие последовательностей seqА и seqB заключается в наличии у второй N-концевой делеции (1 цистеин в положении 37)

Цистеин играет роль в формировании дисульфидных связей, которые стабилизируют трехмерную структуру белков. Если произошла делеция цистеина на N-конце, то можно ожидать:
- Снижение стабильности: Удаление цистеина может привести к потере дисульфидной связи, что делает белок более уязвимым к денатурации при повышенных температурах или изменениях pH.
- Изменение конформации: Потеря цистеина может изменить пространственную конфигурацию белка, что может повлиять на его функциональные свойства
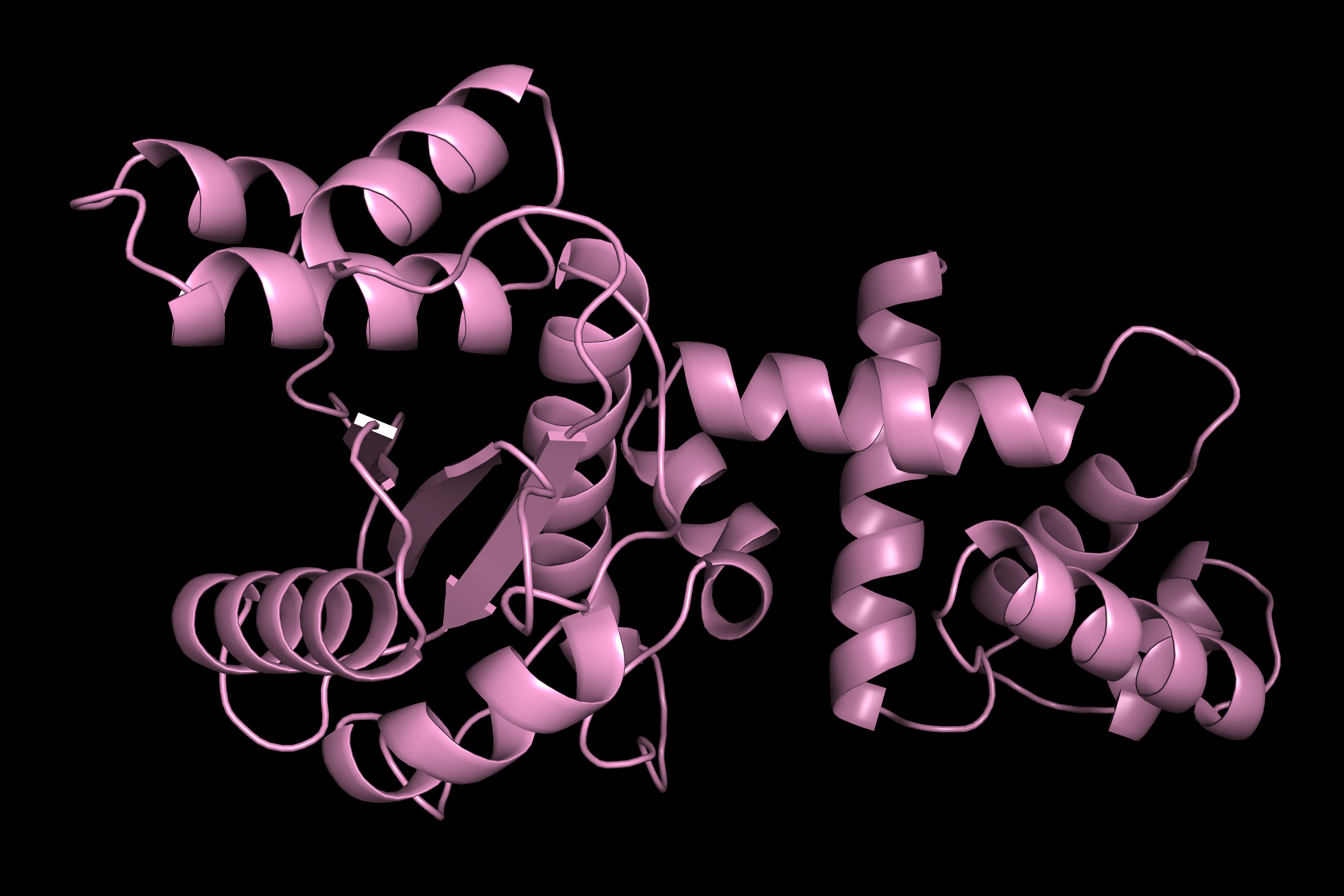
seqA
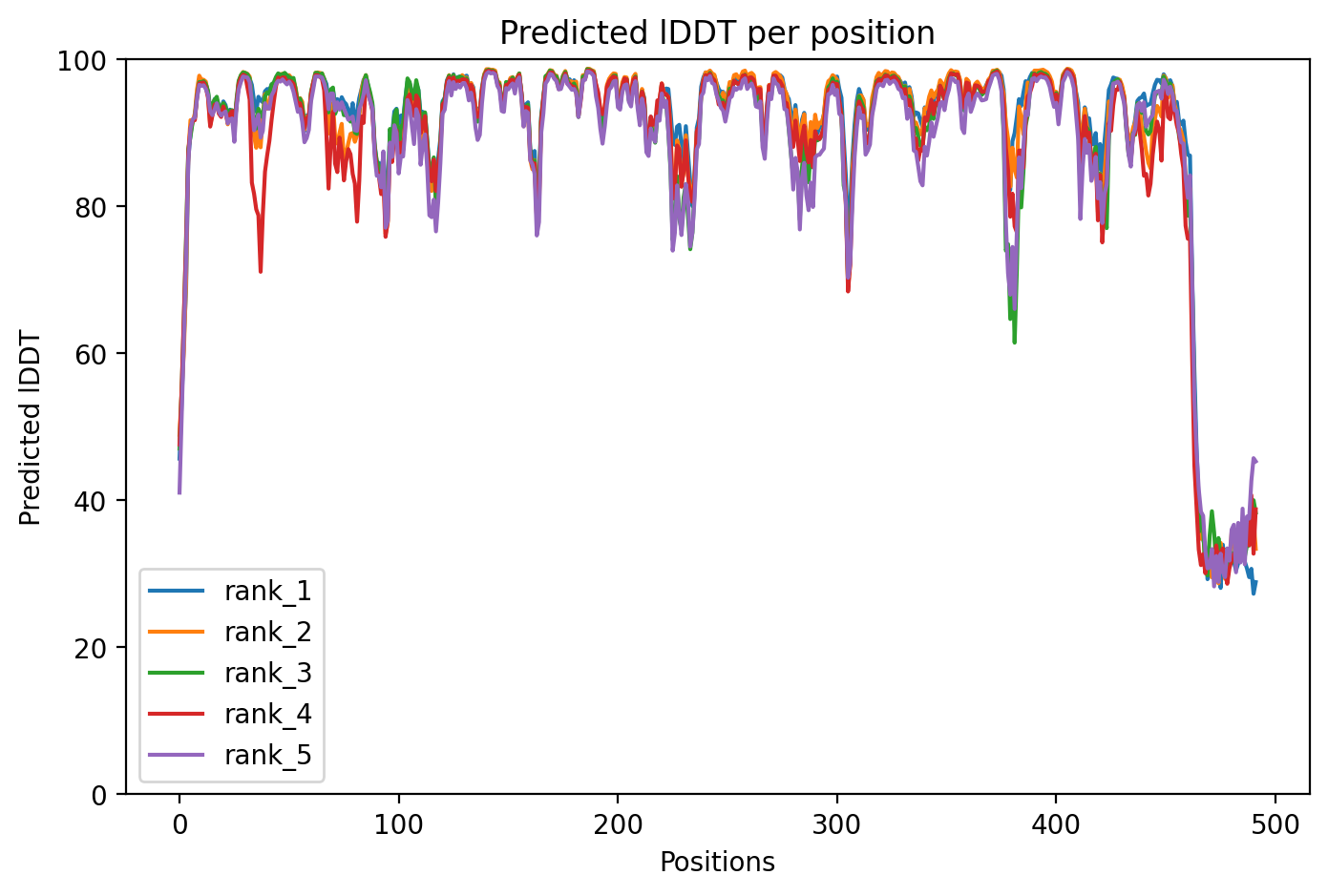
Была предсказана структура для seqA с помощью ColabFold
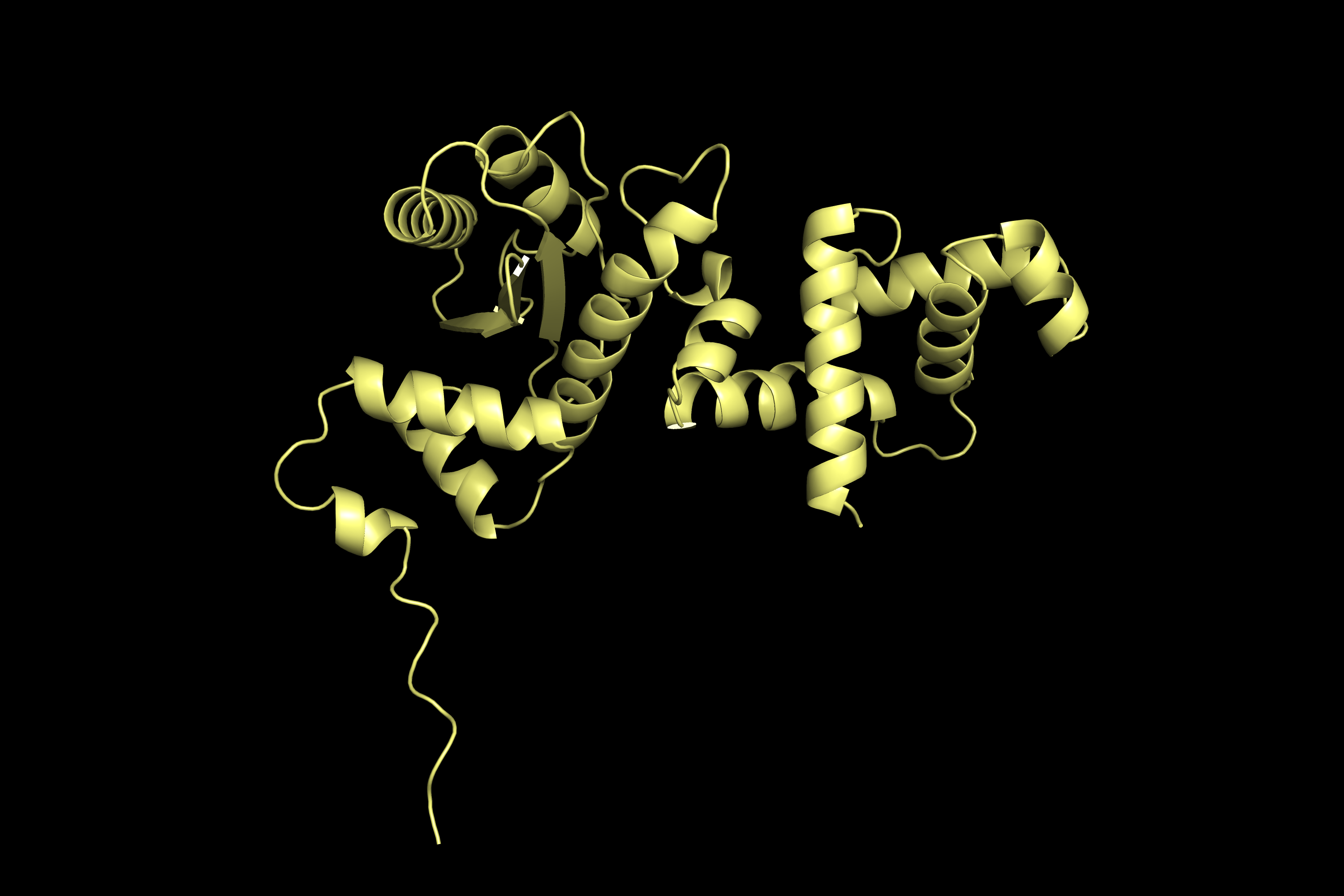
seqB
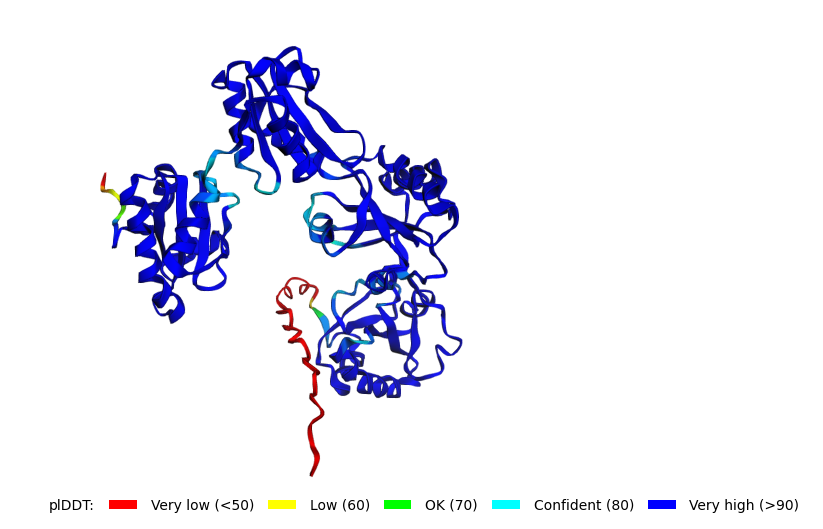
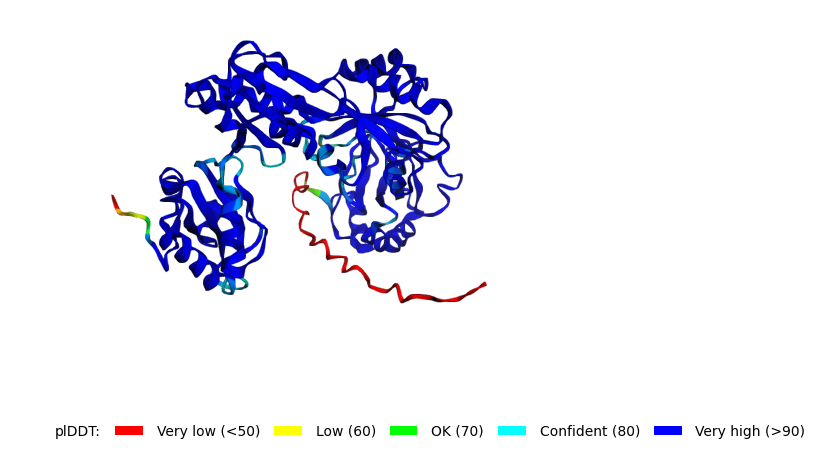
Была предсказана структура для seqB с помощью ColabFold
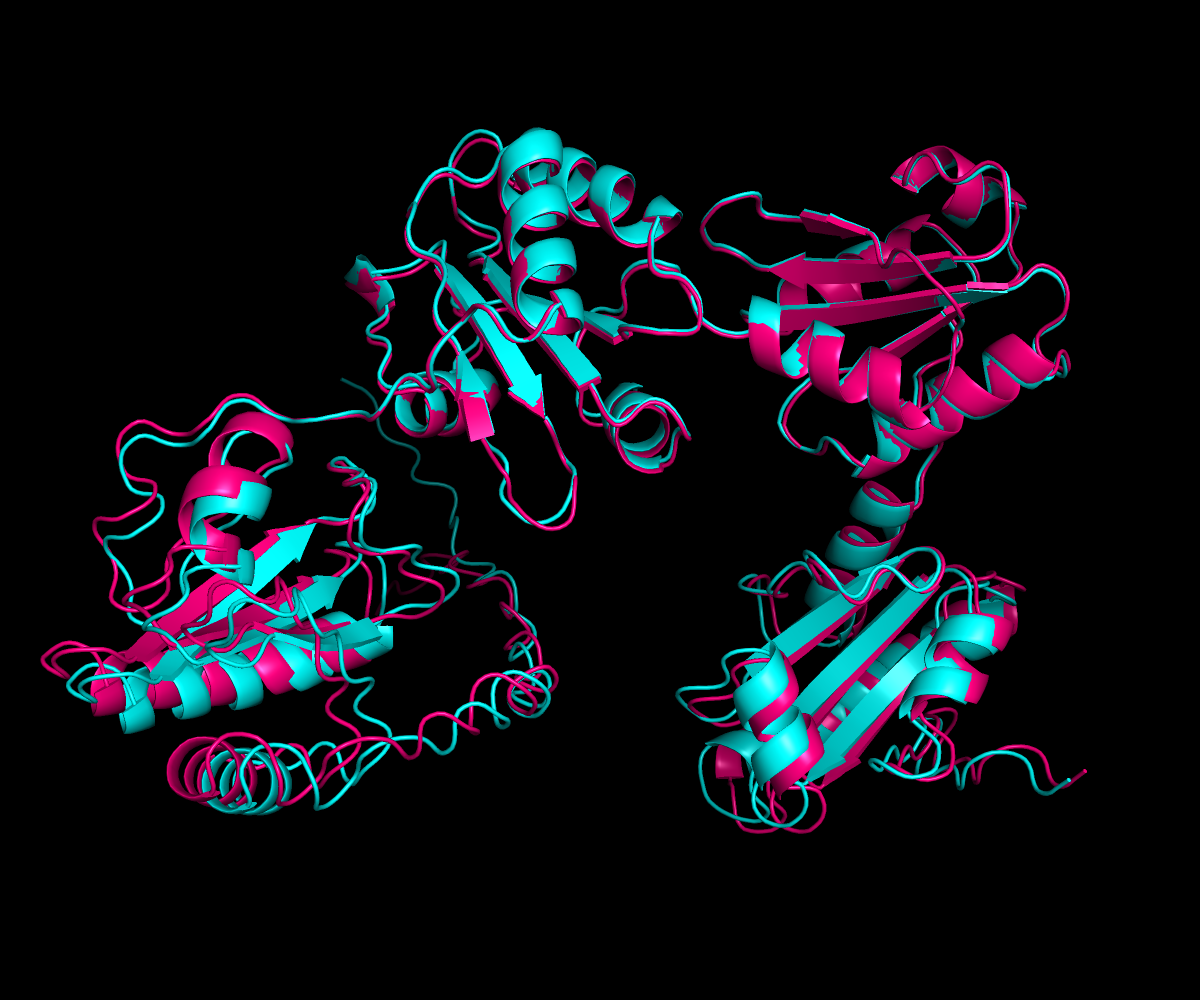
Сравнение предсказанных структур
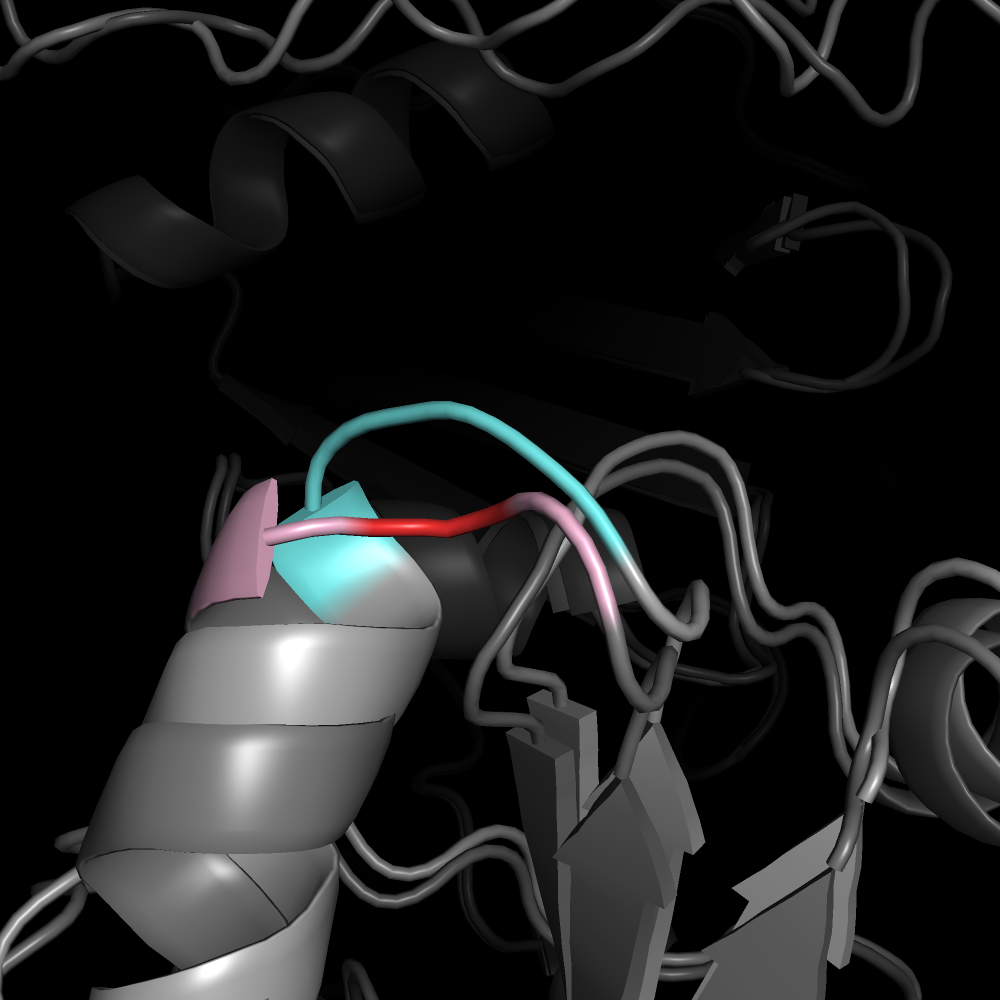
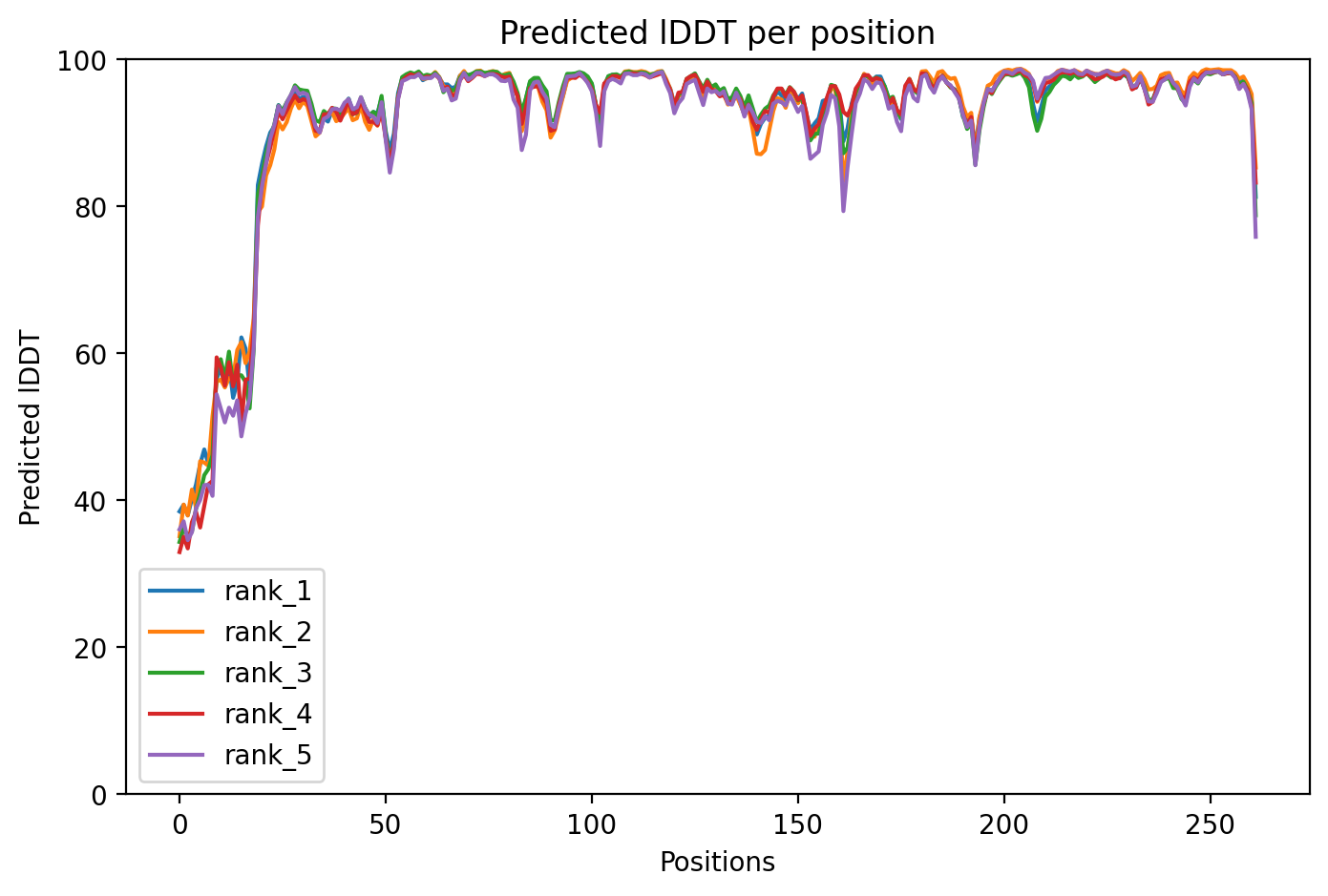
По предсказанным структурам, как будто, при делеции не меняется ни стабильность, ни конфигурация, как будто нет дисульфидной связи в исходной seqA. Поэтому для оценки адекватности предсказания нужно проверить гипотезу гипотезы о потере дисульфидных связей при переходе из seqA в seqB. Если связи и не сущствовало в seqA, то предсказание можно считать правильным.
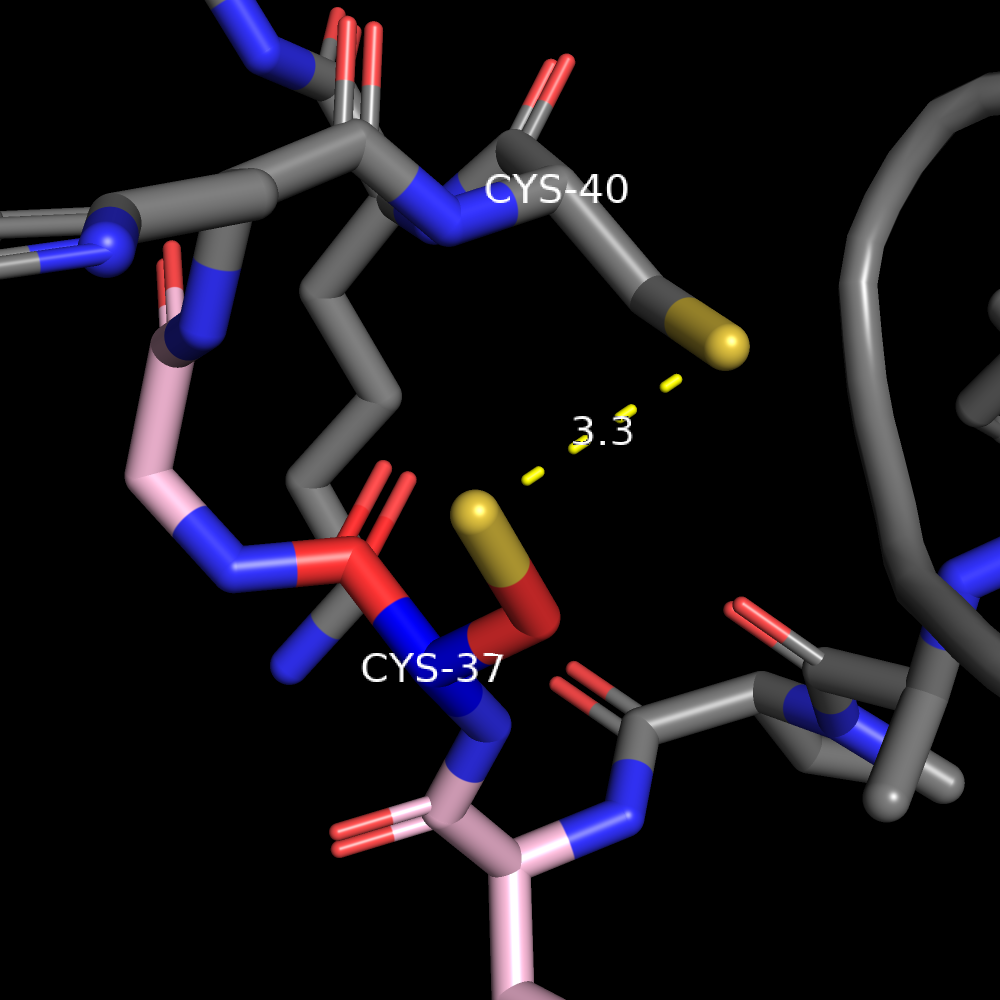
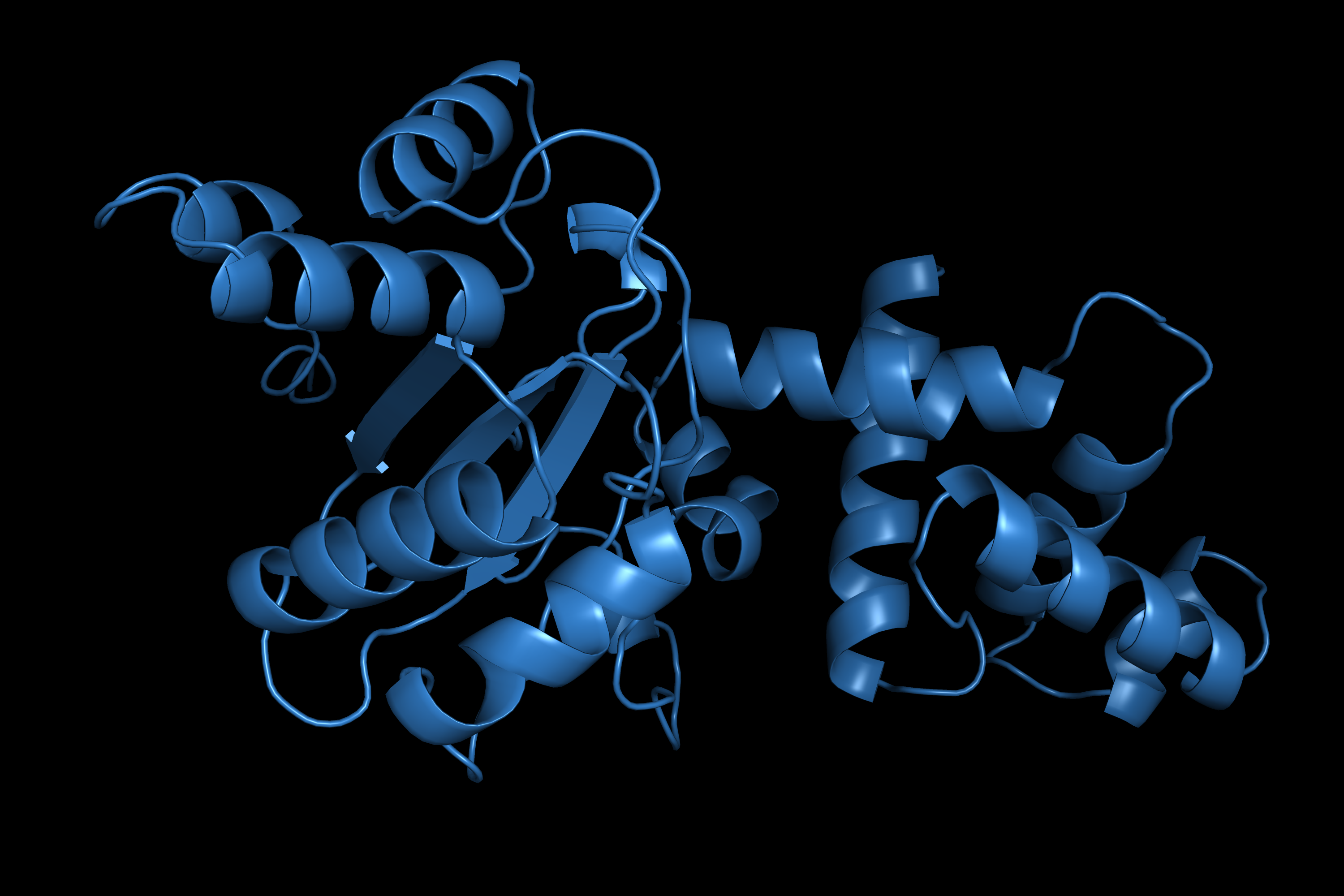
Для проверки наличия потенциальных дисульфидных связей C37 в seqA нужно посмотреть на другие цистеины в структуре и оценить возможно взаимодействия с ними.
Существует С40, который входит в ближайшую альфа-спираль и явно может образовывать дисульфидную связь с C37 (рис 5a), а значит при делеции это взаимодействие исчезает и структура должна претерпевать более сильные изменения, чем предсказывается в seqB (рис 5b).
Задание Helices (B9)
Для выполнения практикума были даны три последовательности (seqA, seqB, seqC).
seqA SNAMNSEQADILDLLSGHTDDTTIERLAFECLLTNMTDDRVVSLMNILGWQGDFNCFAIGGVPSASLASTSLAIRKAVRDLGGEHVVIGTYGTFLLALACQ MGAVTPEVTCTAVMPAFSEDEPLYLSPVRSGVAGASHALRETMFSLQAAPALSTPSRPLRADELLPERALLGDDYAREELYRNVYQVLRGENPDDPTYLTV STFLKYGSSLENTAKELNVHPNTVRYRLKRAAETTGWDATDPRDAYVLTTALAIGRMRDR
seqB SNAMNSEQADILDLLSGHTDDTTIERLAFECLLTNMTDDRVVSLMNILGWQGDFNCFAIGGVPSASLASTSLAIRKAVRDLGGEHVVIGTYGTFLLALACQM GAVTPEVTCTAVMPAFSEDEPLYLSPVRSGVAGASHALPPPMFSLQAAPALSTPSRPLRADELLPERALLGDDYAREELYRNVYQVLRGENPDDPTYLTVS TFLKYGSSLENTAKELNVHPNTVRYRLKRAAETTGWDATDPRDAYVLTTALAIGRMRDR
seqC SNAMNSEQADILDLLSGHTDDTTIERLAFECLLTNMTDDRVVSLMNILGWQGDFNCFAIGGVPSASLASTSLAIRKAVRDLGGEHVVIGTYGTFLLALACQMG AVTPEVTCTAVMPAFSEDEPLYLSPVRSGVAGAPPPPPPPPPPPPAAPALSTPSRPLRADELLPERALLGDDYAREELYRNVYQVLRGENPDDPTYLTVSTFLKY GSSLENTAKELNVHPNTVRYRLKRAAETTGWDATDPRDAYVLTTALAIGRMRDR
Отличие последовательностей seqB и seqC от seqA заключается в замене нескольких аминокислот на пролин (в частности, в seqB – 3 а.о, в seqC – 12). Пролин обладает конформационно жесткой структурой и сильно изгибает пептидную цепь. Также пролин не может быть донором водорода при формировании водородной связи. Участки белков с высоким содержанием пролина могут образовывать вторичную структуру полипролиновой спирали.

seqA
Была предсказана структура для seqA с помощью ColabFold
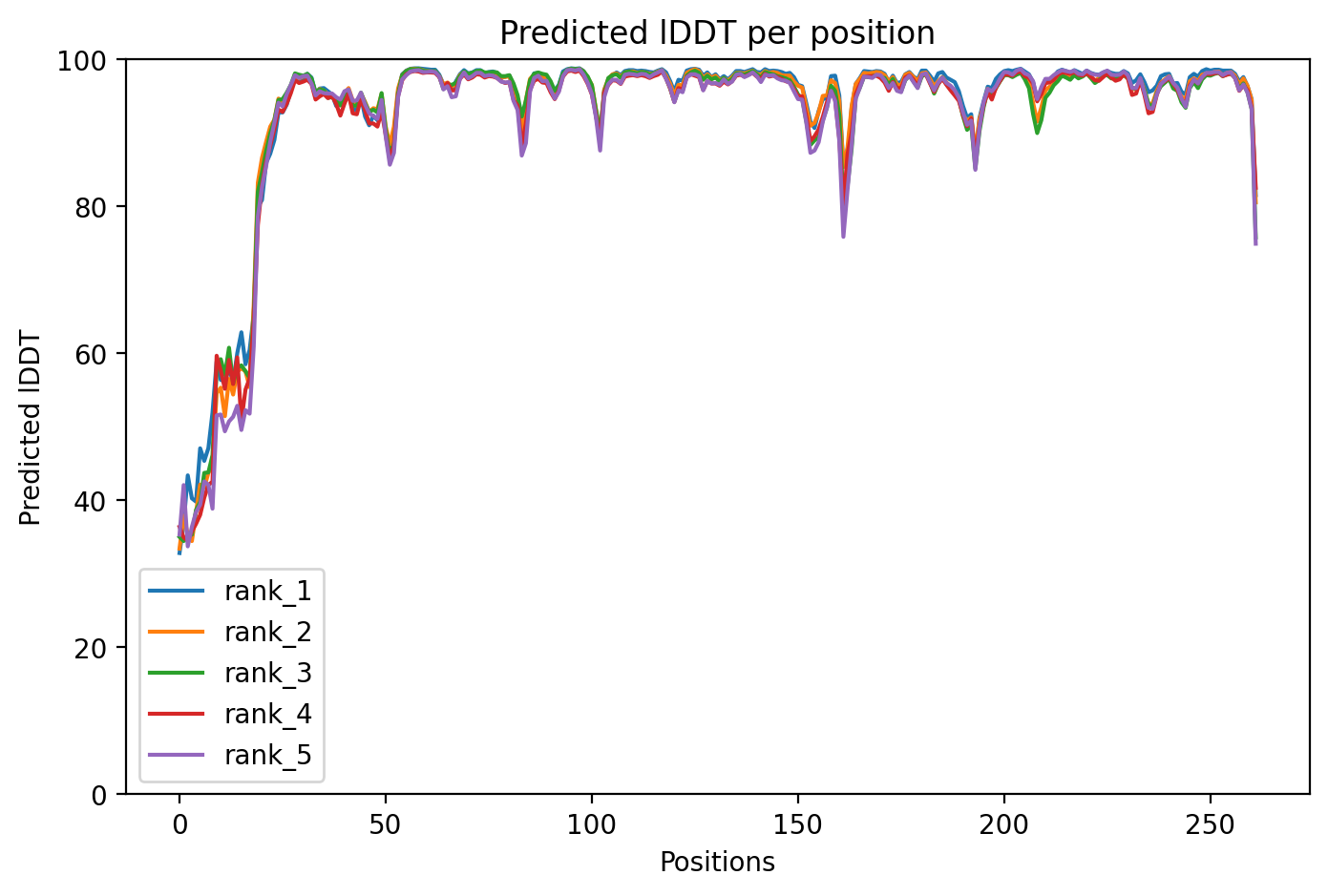
IDDT (степень уверенности программы в предсказании) для всех rank практически не отличался, снижение значений наблюдается в ожидаемом участке с пролинами, а так же в концевой части структуры.
seqB
Была предсказана структура для seqB с помощью ColabFold
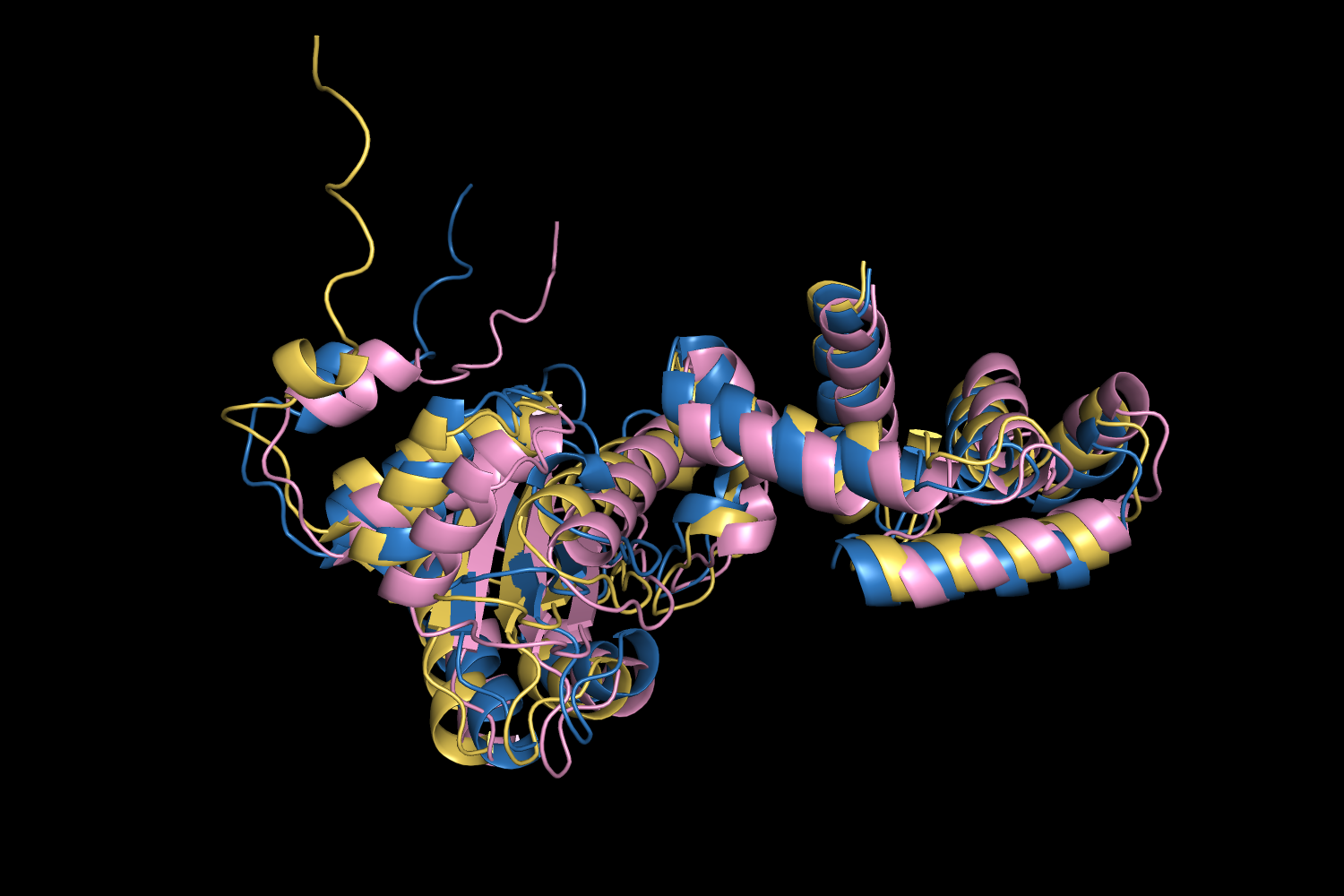
lDDT показывает более высокие показатели, чем для seqA, но почему-то предсказанная структура является зеркальным отображением по отношению к аналогичной seqA, поэтому была отображена в PyMol для удобства сравнения.
seqC
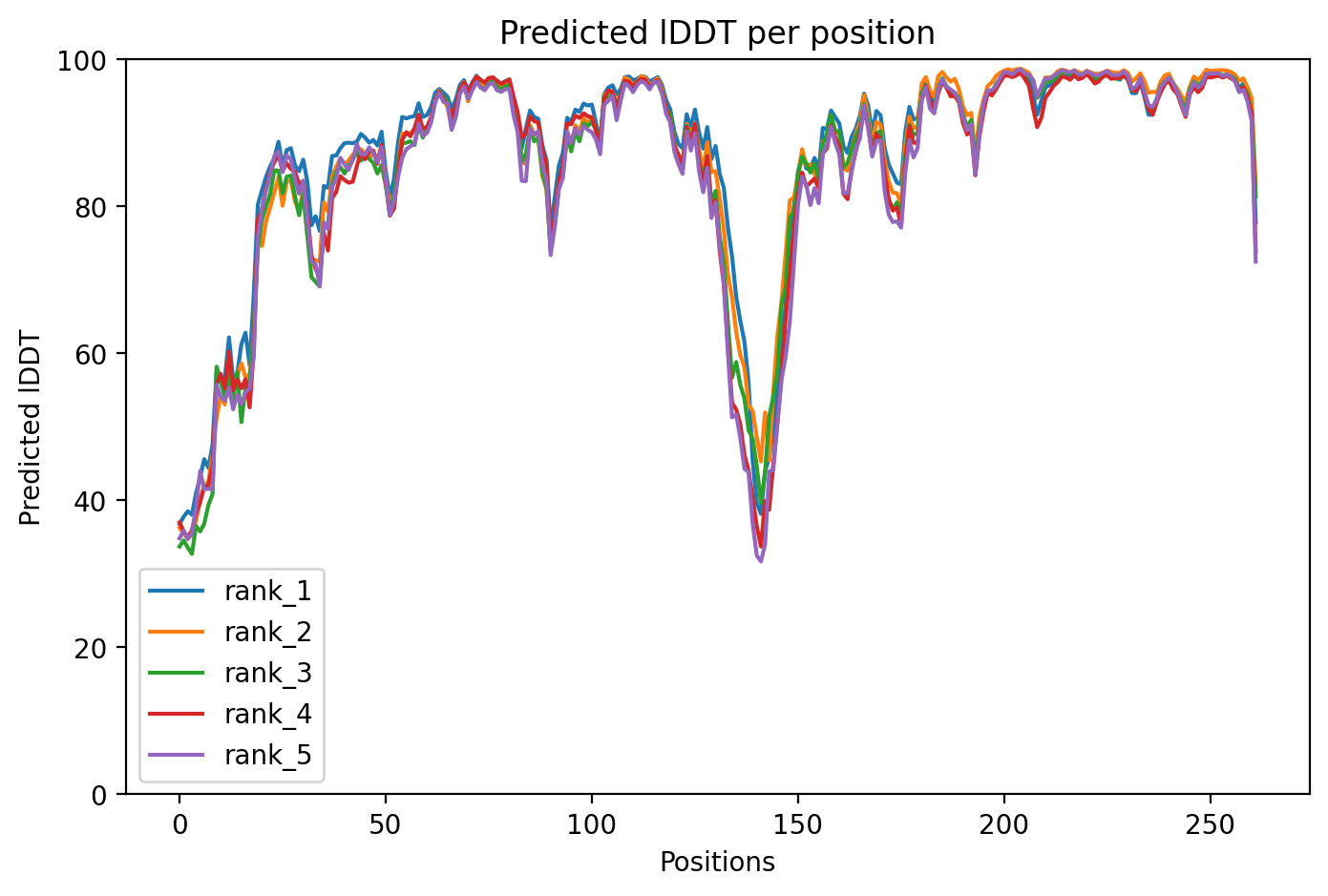
Была предсказана структура для seqC с помощью ColabFold
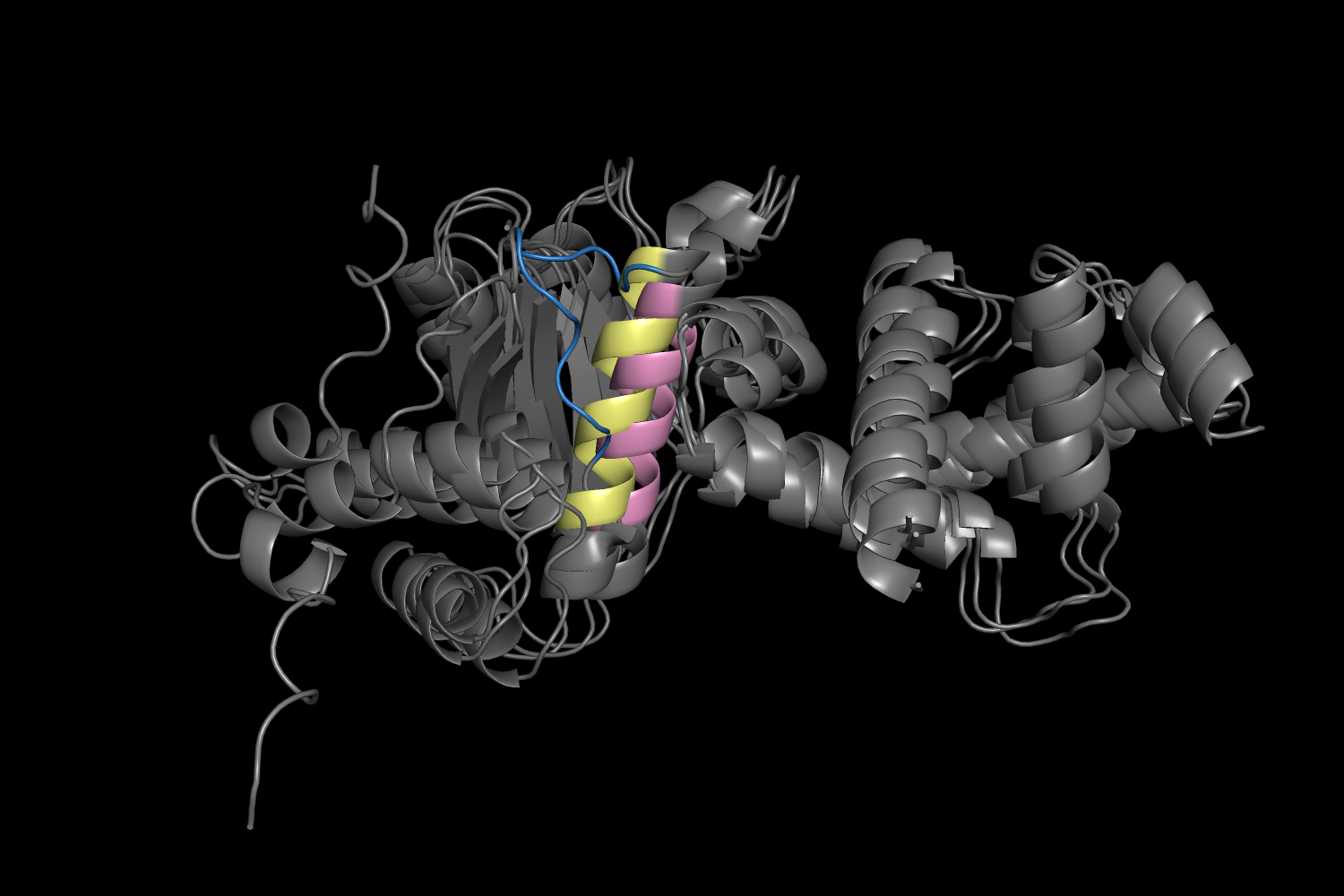
lDDT имеет уже значительное снижение значений в области богатой пролинами и на структуре не образуется альфа-спираль, а получается неструктурированный участок (что соответсвует ожидаемому жесткому участку, который и не должен сворачиваться в спираль).
Сравнение предсказанных структур
Предсказанные структуры накладываются не очень хорошо друг на друга, особенно это выражено на нашем вариабельном участке, предсказанном множественным выравниванием, и на концевом участке, который имеет более низкие значения увренности lDDT во всех предсказанных структурах. Но это правильно отражает изменения структуры с изменнием аминокислотного состава, а значит, AlphaFold правильно учел эти изменения в процессе предсказания.