 Получены итоговые нуклеотидные последовательности двух цепей ДНК, с помощью программы JalView построено их выравнивание с раскраской по нуклеотидам. На основании полученых
данных была составлена итоговая нуклеотидная последовательность.
Получены итоговые нуклеотидные последовательности двух цепей ДНК, с помощью программы JalView построено их выравнивание с раскраской по нуклеотидам. На основании полученых
данных была составлена итоговая нуклеотидная последовательность.
В задании необходимо было проанализировать данные (хроматограмму и автоматически прочтенную последовательность), полученную с капиллярного секвенатора. Все файлы были даны в формате .ab1. Для просмотра и редактирования автоматического прочтения этих хроматограмм использовалась программа Chromas (Lite). Исходные файлы:
В программе Chromas (Lite) требовалось открыть файлы с прямой и обратной последовательностями ДНК, во втором окне с обратной цепью перейти к комплементарной цепи (с помощью команды Edit > Reverse+Complement),
настроить одинаковый масштаб по горизонтали и произвести выравнивание двух хроматограмм с использованием поиска подслов Find.
С помощью опции Continuous edit были удалены не читаемые 5'- и 3'- концы, чьи координаты в разных цепях представлены в таблице ниже. Все координаты определены по прямой последовательности.
Границы не читаемых 5'- и 3'-участков
| Нечитаемый участок (координаты по прямой последовательности) | 5'-конец | 3'-конец |
| Прямая | 1-130 (130 bp) | 684-717 (33 bp) |
| Обратная | 1-32 (32 bp) | 587-717 (130 bp) |
Некоторые участки(как в середине последовательности, так и по краям) нечитаемы в одной последовательности, но при этом их можно определить в другой.
Объединяя результаты для двух цепей, можно получить наиболее точную и полную нуклеотидную последовательность.
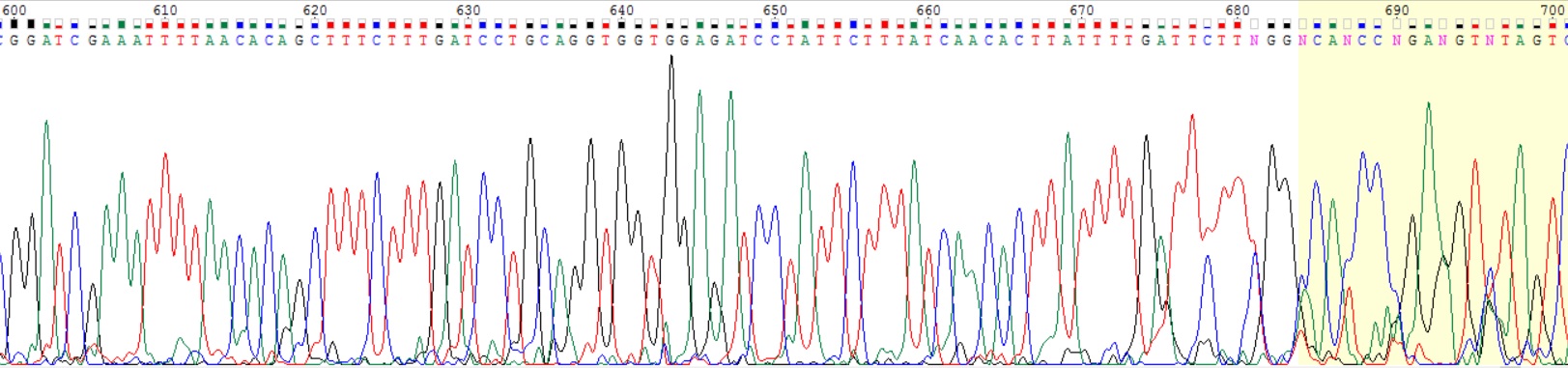
Качество хроматограммы неплохое, мощность сигнала в интервале 131-650 bp достаточная для однозначного определения последовательности ДНК. Качество сигнала от каждого нуклеотида в среднем довольно
высокое (Q~20), можно заметить разброс силы сигнала между разными нуклеотидами: у аденина и гуанина пики в разы выше, чем у пиримидиновых оснований.
Ближе к зоне разделения между читаемым и нечитаемым фрагментом качество хроматограммы и сигнала падает, пики становятся более размытыми, на некоторых наблюдается перекрывание. На концевых участках
уровень шума возрастает и отличить его от сигнала становится невозможно. Размер нечитаемого конечного участка длиннее на 5'-конце.
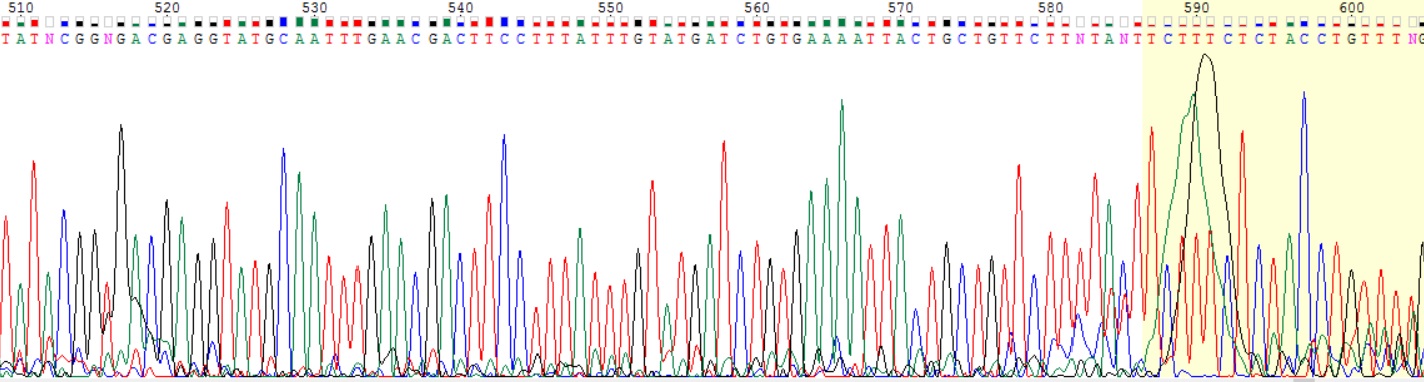
Качество хроматограммы обратной цепи также хорошее, мощность сигнала в интервале 50-580 bp достаточная для однозначного определения последовательности ДНК. Качество сигнала от каждого нуклеотида
в среднем довольно высокое (Q~20), можно заметить разброс силы сигнала между разными нуклеотидами: у цитозина и тимина пики в разы выше, чем у пуриновых оснований.
Ближе к зоне разделения между читаемым и нечитаемым фрагментом качество хроматограммы и сигнала падает, пики становятся более размытыми, на некоторых наблюдается перекрывание. На концевых участках
уровень шума возрастает и отличить его от сигнала становится невозможно. Размер нечитаемого конечного участка длиннее на 3'-конце(после команды Edit > Reverse+Complement).
Далее я проанализировала последовательности и отредактировала их посредством сравнения результатов двух хроматограмм и исправления потенциально возможных ошибок софта секвенатора. Все исправления
внесены строчными буквами.
В процессе редактирования хроматограмм нередко возникали ситуации, когда однозначный выбор нуклеотида в позиции был невозможен. В итоговых fasta-файлах на таких позициях
ставились буквы вырожденного кода(IUPAC Ambiguity Codes[1]).
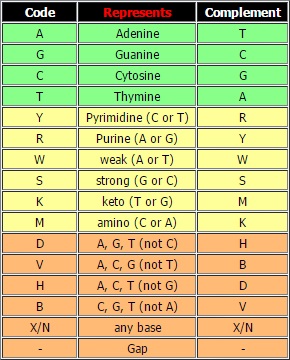
Таблица 1. IUPAC Ambiguity Codes
 Получены итоговые нуклеотидные последовательности двух цепей ДНК, с помощью программы JalView построено их выравнивание с раскраской по нуклеотидам. На основании полученых
данных была составлена итоговая нуклеотидная последовательность.
Получены итоговые нуклеотидные последовательности двух цепей ДНК, с помощью программы JalView построено их выравнивание с раскраской по нуклеотидам. На основании полученых
данных была составлена итоговая нуклеотидная последовательность.

Рис.3 Выравнивание, раскрашенное по схеме Nucleotide. Проблемные нуклеотиды отмечены строчными символлами с использованием IUPAC Ambiguity Codes.
| Описание проблемы | Изображение прямой цепи | Изображение обратной цепи |
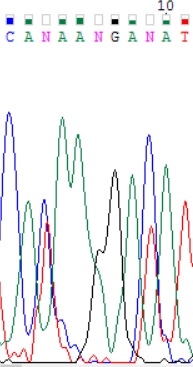
| 1. Обозначенный буквой N нуклеотид был не определен программой в прямой цепи, так как в ней на этой позиции наблюдается довольно сильный шумовой сигнал. В обратной последовательности присутствует лишь один выраженный пик аденина при минимальном сигнале шума. |
 | 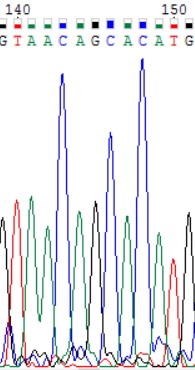 |
| 2. Обозначенный буквой N нуклеотид был не определен программой в обратной цепи, так как в ней на этой позиции наблюдается сигнал, практически не отличающийся по силе от шумового. В прямой последовательности присутствует лишь один выраженный пик цитозина при практически отсутствующем сигнале шума. |
 | 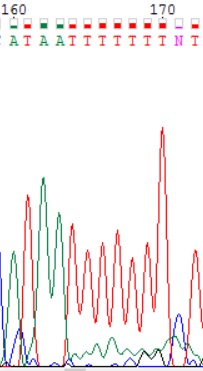 |
| 3. Обозначенный буквой N нуклеотид на 3 позиции был не определен программой в обратной цепи, так как в ней на этой позиции наблюдается два эквивалентных по силе сигнала: тимина и цитозина. В прямой последовательности аналогичный участок не читаем. В соответствии с таблицей IUPAC Ambiguity Codes был указан символ Y вырожденного кода. |
- |  |
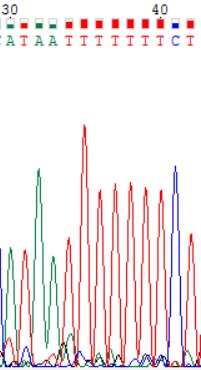
| 4. Обозначенные буквой N нуклеотиды в позциях 480 и 484 были не определены программой в обратной цепи, так как в ней на этой позиции наблюдается слабые сигналы, которые легко могут быть спутаны с шумом. В прямой последовательности на этих позициях присутствуют достаточно выраженные пики аденина и тимина при слабом сигнале шума. |
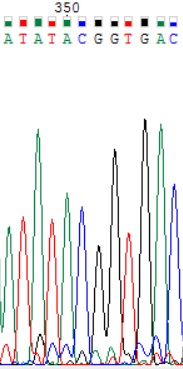 | 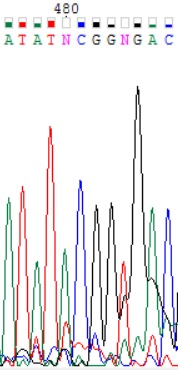 |
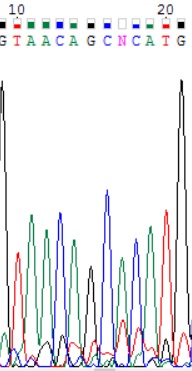
На рис. 4 приведен пример не читаемого фрагмента хроматограммы. Высокий уровень шума и перекрывающиеся пики указывают на загрязнение образца или возможное нахождение в нем
нескольких разных ДНК. Сильно выдающиеся и размытые пики по центру могут быть пятнами краски.
Рис.4 Пример плохой хроматограммы.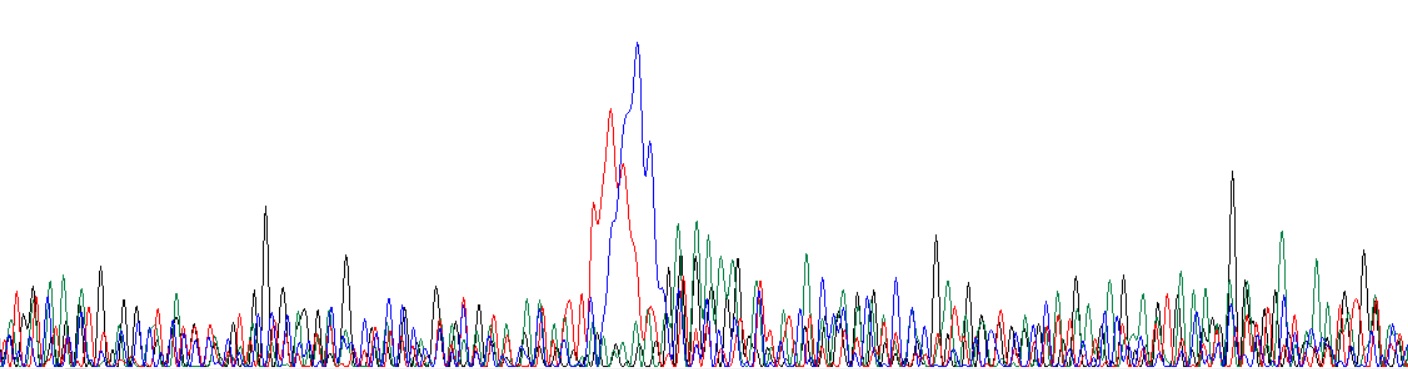
Источники:
© Avdiunina Polina, 2015