В задании необходимо было выбрать эукариотический организм и охарактеризовать качество сборки его генома, используя базу данных NCBI Genome и выполняя поиск по организму (Genome > Browse by organism).
Для оценки качества сборки я выбрала Chinese hamster (Cricetulus griseus).
 |
В дикой природе китайский хомячок обитает в пустынях Северного Китая и Монголии. Взрослые особи
вырастают до 82-127 мм (длина хвоста 20-33 мм) и весят до 30-45 г. Средняя продолжительность жизни
у китайского хомячка составляет 2-3 года.
В прошлом китайские хомячки часто использовались в качестве лабораторных животных, но были вытеснены
домовой мышью (Mus musculus) и серой крысой (Rattus norvegicus), которых легче содержать
и разводить в лабораторных условиях. Тем не менее, для разработки и изготовления многих
биотехнологических препаратов до сих пор используется культура клеток яичника китайского хомячка.
Часто в клетки такой культуры вносят ген интересующего белка для его получения в нужном количестве.[1]
Из культуры клеток яичника китайского хомячка изготавливают Rebif(интерферон бета-1a) — препарат
для патогенетического лечения рассеянного склероза.[2]
Для данного организма пока есть три сборки генома - GCA_000223135.1 (CriGri_1.0),
GCA_000419365.1 (C_griseus_v1.0), GCA_000448345.1 (Cgr1.0).
Зарегистрировано 3 проекта по секвенированию генома: PRJNA69991, PRJNA189319, PRJNA167053.
Сводную информацию по проектам можно найти в таблице ниже. |
Проекты
| PRJNA69991 | PRJNA189319 | PRJNA167053 |
| ID проекта | 69991 | 189319 | 167053 |
| Дата регистрации проекта | 3-Aug-2011 | 8-Aug-2013 | 8-Jul-2013 |
| Цель изучения | Medical | Model Organism | Evolution |
| Идентификатор образца | SAMN02981352 | SAMN02981520 | SAMN02981459 |
| Сборка | GCA_000223135.1 | GCA_000448345.1 | GCA_000419365.1 |
| Размер (Mb) | 2399.79 | 2332.77 | 2360.13 |
| GC% | 41.60 | 42.00 | 41.50 |
| Гены | 27752 | 21779 | 27756 |
| Белки | 34935 | 29144 | 32843 |
Описание сборки GCA_000223135.1
Количество образцов в проекте - 3. Конечная сборка сделана по образцу BioSample: SAMN02981352; GenBank: gb|AFTD00000000.1. Образец представлял собой клетки из клеточной линии CHO-K1.
Клеточная линия CHO-K1 представляет собой эпителиальные клетки, полученные из яичников Cricetulus griseus, которые широко используются в генетических исследованиях и исследованиях
экспрессии генов, в частности, экспрессии рекомбинантных белков. Клетки CHO-K1 характеризуются быстрым ростом в суспензионной культуре, высокой продукцией белка и малым количеством (для млекопитающего)
числа хромосом (2n=22).
Образец взят у самки Cricetulus griseus в Китае. Описание соответствующего проекта см. в таблице выше.
 Два других образца:
Два других образца:
- BioSample: SAMN00691388; Sample name: CHO_K1; SRA: SRS255210
- BioSample: SAMN00691386; Sample name: BGI_CHO-K1; SRA: SRS255208
Для сборки GCA_000223135.1
- Длина полследовательности - 2,399,786,748
- Гэпы между скэффолдами - 0
- Число скэффолдов - 109,152
- Scaffold N50 - 1,147,233
- Scaffold L50 - 547
- Scaffold N50 - 1,147,233
- Число контигов - 265,787
- Contig N50 - 39,361
- Contig L50 - 16,414
- Общее число хромосом и плазмид - 1
- Таблица контигов
- Самый короткий контиг - 50 (AFTD01001448)
- Самый длинный контиг - 412584 (AFTD01050443)
- Последовательность контига AFTD01000004
Feature Key
Последовательности нуклеиновых кислот предоставляют фундаментальную отправную точку для описания и понимания структуры, функций и развития генетически разнообразных организмов. Банки нуклеотидных
последовательностей, такие как GenBank, EMBL, и DDBJ с самого своего основания используют таблицы сайтов и особенностей для описания местонахождения и роли наиболее высокоорганизованных
доменов нуклеотидных последовательностей и элементов генома организма.
В феврале 1986 года, GenBank и EMBL(а затем и DDBJ) разрабатывают единые стандарты таблиц аннотирования.
Документация таблиц особенностей демонстрирует общие правила, которые дают возможность обмена данных между тремя вышеупомянутыми банками последовательностей на регулярной основе.
Участки с различными особенностями, которые будут представлены:
- выполняют определенную биологическую функцию
- влияют или являются результатом эквпрессии определенной биологической функции
- взаимодействуют с другими молекулами
- влияют на репликативную активность
- влияют или являются результатом рекомбинации различных последовательностей
- являются узнаваемым повторяющимся участком
- имеют вторичную или третичную структуру
- демонстрируют вариабельность или были отредактированы
Формат записей основывается на табличном подходе и включает следующие пункты:
- Feature key - слово или аббревиатура, указывающие на функциональную группу
- Location - инструкции, указыващие где найти данную особенность
- Qualifiers- дополнительная информация
Формат и формулировки таблиц ключей используют общепринятую биологическую терминологию. Например:
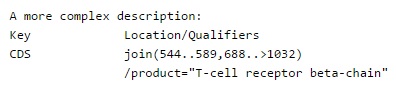 Эта запись может быть прочтена так: последовательность, кодирующая бета-цепь Т-клеточного рецептора, состоит из двух отдельных частей с координатами (544..589,688..>1032).
Эта запись может быть прочтена так: последовательность, кодирующая бета-цепь Т-клеточного рецептора, состоит из двух отдельных частей с координатами (544..589,688..>1032).
Примеры ключей
Feature Key sig_peptide
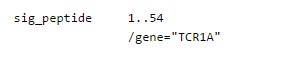 Если в белке присутствует сигнальный пептид - короткая (от 3 до 60 аминокислот) аминокислотная последовательность в составе белка, которая обеспечивает котрансляционный или посттрансляционный транспорт
белка в соответствующую органеллу (ядро, митохондрия, эндоплазматический ретикулум, хлоропласт, апопласт или пероксисома), то в этом ключе даются координаты кодирующей его последовательности и название гена,
в который входит эта последовательность.
Feature Key source
Если в белке присутствует сигнальный пептид - короткая (от 3 до 60 аминокислот) аминокислотная последовательность в составе белка, которая обеспечивает котрансляционный или посттрансляционный транспорт
белка в соответствующую органеллу (ядро, митохондрия, эндоплазматический ретикулум, хлоропласт, апопласт или пероксисома), то в этом ключе даются координаты кодирующей его последовательности и название гена,
в который входит эта последовательность.
Feature Key source
 Ключ - идентификатор биологического источника последовательности, является обязательным. Допускается многоразовое использование этого ключа(при нескольких источниках).
Feature Key mRNA
Ключ - идентификатор биологического источника последовательности, является обязательным. Допускается многоразовое использование этого ключа(при нескольких источниках).
Feature Key mRNA
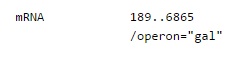 Ключ, содержащий координаты кодирующей мРНК последовательности, включающей в себя 5'UTR, кодирующую последовательность (CDS, экзон) и 3'UTR.
Feature Key regulatory
Ключ, содержащий координаты кодирующей мРНК последовательности, включающей в себя 5'UTR, кодирующую последовательность (CDS, экзон) и 3'UTR.
Feature Key regulatory
 Ключ - идентификатор участков последовательности, вовлеченных в регуляцию транскрипции или трансляции. Заменил собой следующие ключи: enhancer, promoter, CAAT_signal, TATA_signal, -35_signal, -10_signal,
RBS, GC_signal, polyA_signal, attenuator, terminator, misc_signal. Обязательный спецификатор: /regulatory_class="TYPE".
Feature Key CDS
Ключ - идентификатор участков последовательности, вовлеченных в регуляцию транскрипции или трансляции. Заменил собой следующие ключи: enhancer, promoter, CAAT_signal, TATA_signal, -35_signal, -10_signal,
RBS, GC_signal, polyA_signal, attenuator, terminator, misc_signal. Обязательный спецификатор: /regulatory_class="TYPE".
Feature Key CDS
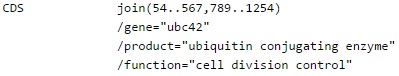 Ключ координат кодирующей последовательности гена, которая соответствует аминокислотной последовательности в белке(включая стоп-кодон). Спецификаторы этого ключа часто указывают на название гена, кодируемого
белка, его функции(-ий) и др.
Feature Key intron
Ключ координат кодирующей последовательности гена, которая соответствует аминокислотной последовательности в белке(включая стоп-кодон). Спецификаторы этого ключа часто указывают на название гена, кодируемого
белка, его функции(-ий) и др.
Feature Key intron
 Координаты транскрибирующегося участка ДНК, который потом удаляется из транскрипта путем сплайсинга в процессе созревания РНК.
Feature Key operon
Координаты транскрибирующегося участка ДНК, который потом удаляется из транскрипта путем сплайсинга в процессе созревания РНК.
Feature Key operon
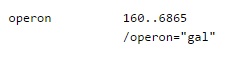 Ключ последовательности, содержащей полицистронный транскрипт, который включает в себя кластер генов, находящихся под контролем одного промотера.
Feature Key rep_origin
Ключ последовательности, содержащей полицистронный транскрипт, который включает в себя кластер генов, находящихся под контролем одного промотера.
Feature Key rep_origin
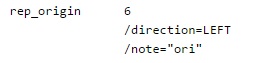 Координаты сайта начала репликации.
Feature Key V_region
Координаты сайта начала репликации.
Feature Key V_region
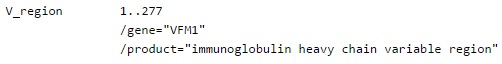 Вариабельные участки легких и тяжелых цепей иммуноглобулинов, α-, β- и γ-цепей Т-клеточных рецепторов.
Feature Key tRNA
Вариабельные участки легких и тяжелых цепей иммуноглобулинов, α-, β- и γ-цепей Т-клеточных рецепторов.
Feature Key tRNA
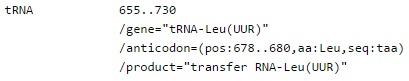 Ключ, содержащий координаты кодирующей тРНК последовательности. Обычно в ключе указывается позиция антикодона в этой последовательности и аминокислота, которую несет конкретная тРНК.
Ключ, содержащий координаты кодирующей тРНК последовательности. Обычно в ключе указывается позиция антикодона в этой последовательности и аминокислота, которую несет конкретная тРНК.
The Cancer Genome Atlas
|
Cancer Genome Atlas (TCGA)- проект, создающий каталоги генетических мутаций, ответственных за рак, используя методы секвенирования
генома и биоинформатику. TCGA использует методы анализа генома с высокой пропускной способностью, с целью улучшить возможности диагностирования,
лечения и профилактики рака благодаря более глубокому пониманию генетических основ этого заболевания. Проект стартовал в 2005 году с пилотной
версии и продолжается до сих пор. Финансирование предоставляется правительством США. Трехлетний пилотный проект был сосредоточен на характеристике
трех типов рака человека: мультиформной глиобластоме, раке легких и раке яичников. В 2009 году проет перешел в II этап, в процессе которого
планировалось завершить геномную характеризацию и анализ последовательностей 20-25 различных типов опухолей к 2014 году. Проект превзошел цели,
было охарактеризовано 33 типа рака, включая 10 редких видов.
TCGA управляется учеными и менеджерами из Национального института рака (NCI) и Национального исследовательского института генома человека (NHGRI).
Посдедняя из опубликованных статей, связанных с проектом: Relation between Established Glioma Risk Variants and DNA Methylation in the Tumor.
Перейти на страницу проекта Вы можете нажав на картинку. |
 |
|
Таблица митохондриальных генов
|
В задании требовалось найти все полные митохондриальные геномы по таксону Euglenozoa, выбрать представителя и создать таблицу всех митохондриальных генов
этого организма. Поиск производился по запросу ((((euglenozoa[ORGN]) AND ("complete genome"[Title] OR "complete sequence"[Title])) AND (mitochondrion[Title] OR
kinetoplast[Title])) NOT partial[Title]). Результаты поиска можно посмотреть по ссылке.
Было найдено 263 результата, 262 из GenBank, 1 из RefSeq.
Можно заметить, что в запросе присутствует фраза mitochondrion[Title] OR kinetoplast[Title]. Это изменение было введено в запрос, т.к. у некоторых организмов,
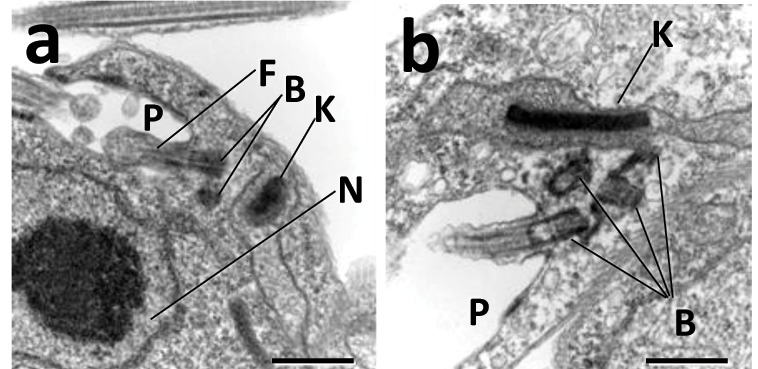
принадлежащих к выданному мне таксону Euglenozoa, имеется кинетопласт(рис.1, K) — клеточная органелла протистов, находящаяся внутри гигантской
митохондрии и содержащая множество копий митохондриального генома.[3]
На рис.1 можно увидеть электронную микрофотографию кинетопласта (K) из Trypanosoma brucei.
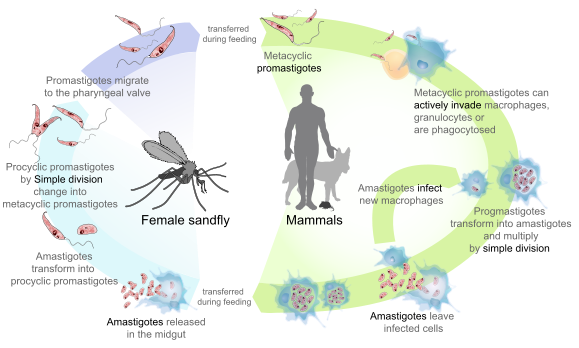
Был взят организм Leishmania tarentolae - облигатный паразитический протист, обитающий в крови человека и вызывающий лейшманиоз. Жизненный цикл
Leishmania donovani можно увидеть на рис.2. |
 |
Для получения списка митохондриальных генов нужно было перейти по ссылке gene
в разделе Related information. Cписок был отсортирован в соответствии с порядком
генов в геноме. Всего был найдено 24 гена. Итоговый файл Exel.
Таблица размеров геномов
| Вироиды | Вирусы | Археи | Бактерии | Эукариоты |
| Минимальный | 220
(RYMV, rice yellow mottle sobemovirus) | 1760
(Porcine Circovirus) | 491000
(Nanoarchaeum equitans) | 580073
(Mycoplasma genitalium) | 551000
(Guillardia theta) |
| Средний | 300-350 | 104 | 106 | 107 | ~109 |
| Максимальный | 467 | 2800000
(Pandoravirus salinus) | 5751000
(Metanosoma acetivorans) | 9200000
(Bradhyrhizobium japonicum) | 670000000000
(Amoeba dubia) |
Источники:
[1] Chinese hamster
[2] подсемейство Хомяки
[3] Кинетопласт Wiki
©
Avdiunina Polina, 2015

 Два других образца:
Два других образца:
 Эта запись может быть прочтена так: последовательность, кодирующая бета-цепь Т-клеточного рецептора, состоит из двух отдельных частей с координатами (544..589,688..>1032).
Эта запись может быть прочтена так: последовательность, кодирующая бета-цепь Т-клеточного рецептора, состоит из двух отдельных частей с координатами (544..589,688..>1032).
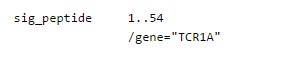 Если в белке присутствует сигнальный пептид - короткая (от 3 до 60 аминокислот) аминокислотная последовательность в составе белка, которая обеспечивает котрансляционный или посттрансляционный транспорт
белка в соответствующую органеллу (ядро, митохондрия, эндоплазматический ретикулум, хлоропласт, апопласт или пероксисома), то в этом ключе даются координаты кодирующей его последовательности и название гена,
в который входит эта последовательность.
Feature Key source
Если в белке присутствует сигнальный пептид - короткая (от 3 до 60 аминокислот) аминокислотная последовательность в составе белка, которая обеспечивает котрансляционный или посттрансляционный транспорт
белка в соответствующую органеллу (ядро, митохондрия, эндоплазматический ретикулум, хлоропласт, апопласт или пероксисома), то в этом ключе даются координаты кодирующей его последовательности и название гена,
в который входит эта последовательность.
Feature Key source
 Ключ - идентификатор биологического источника последовательности, является обязательным. Допускается многоразовое использование этого ключа(при нескольких источниках).
Feature Key mRNA
Ключ - идентификатор биологического источника последовательности, является обязательным. Допускается многоразовое использование этого ключа(при нескольких источниках).
Feature Key mRNA
 Ключ, содержащий координаты кодирующей мРНК последовательности, включающей в себя 5'UTR, кодирующую последовательность (CDS, экзон) и 3'UTR.
Feature Key regulatory
Ключ, содержащий координаты кодирующей мРНК последовательности, включающей в себя 5'UTR, кодирующую последовательность (CDS, экзон) и 3'UTR.
Feature Key regulatory
 Ключ - идентификатор участков последовательности, вовлеченных в регуляцию транскрипции или трансляции. Заменил собой следующие ключи: enhancer, promoter, CAAT_signal, TATA_signal, -35_signal, -10_signal,
RBS, GC_signal, polyA_signal, attenuator, terminator, misc_signal. Обязательный спецификатор: /regulatory_class="TYPE".
Feature Key CDS
Ключ - идентификатор участков последовательности, вовлеченных в регуляцию транскрипции или трансляции. Заменил собой следующие ключи: enhancer, promoter, CAAT_signal, TATA_signal, -35_signal, -10_signal,
RBS, GC_signal, polyA_signal, attenuator, terminator, misc_signal. Обязательный спецификатор: /regulatory_class="TYPE".
Feature Key CDS
 Ключ координат кодирующей последовательности гена, которая соответствует аминокислотной последовательности в белке(включая стоп-кодон). Спецификаторы этого ключа часто указывают на название гена, кодируемого
белка, его функции(-ий) и др.
Feature Key intron
Ключ координат кодирующей последовательности гена, которая соответствует аминокислотной последовательности в белке(включая стоп-кодон). Спецификаторы этого ключа часто указывают на название гена, кодируемого
белка, его функции(-ий) и др.
Feature Key intron
 Координаты транскрибирующегося участка ДНК, который потом удаляется из транскрипта путем сплайсинга в процессе созревания РНК.
Feature Key operon
Координаты транскрибирующегося участка ДНК, который потом удаляется из транскрипта путем сплайсинга в процессе созревания РНК.
Feature Key operon
 Ключ последовательности, содержащей полицистронный транскрипт, который включает в себя кластер генов, находящихся под контролем одного промотера.
Feature Key rep_origin
Ключ последовательности, содержащей полицистронный транскрипт, который включает в себя кластер генов, находящихся под контролем одного промотера.
Feature Key rep_origin
 Координаты сайта начала репликации.
Feature Key V_region
Координаты сайта начала репликации.
Feature Key V_region
 Вариабельные участки легких и тяжелых цепей иммуноглобулинов, α-, β- и γ-цепей Т-клеточных рецепторов.
Feature Key tRNA
Вариабельные участки легких и тяжелых цепей иммуноглобулинов, α-, β- и γ-цепей Т-клеточных рецепторов.
Feature Key tRNA
 Ключ, содержащий координаты кодирующей тРНК последовательности. Обычно в ключе указывается позиция антикодона в этой последовательности и аминокислота, которую несет конкретная тРНК.
Ключ, содержащий координаты кодирующей тРНК последовательности. Обычно в ключе указывается позиция антикодона в этой последовательности и аминокислота, которую несет конкретная тРНК.

