В задании необходимо было сравнить предсказания генов, хранящихся в базе данных GenBank и сгенерированных программой Prodigal. Для выполнения была выдана плазмида LN606601.
Она имеет размер 80028 bp, кодирует 3,674 гена, из которых 3,467 белок-кодирующих, 135 псевдогенов, 15 генов rRNA(5S, 16S, 23S), 56 генов tRNA и 1 ген ncRNA(non-coding RNA).
 |
| Acetobacter senegalensis |
Плазмида принадлежит организму Acetobacter senegalensis - представителю рода Acetobacter - группы грам-отрицательных бактерий,
обладающих ферментативной способностью окислять сахара или этанол для получения уксусной кислоты. Некоторые из ни используются в
промышленных масштабах для производства пищевых продуктов и химических веществ. Морфотип бактерий данного рода - палочковиный,
все представители являются аэробами. Повсеместно распространены в природных средах, где в результате ферментации сахаров этанол
остается в качестве побочного продукта: их можно выделить из цветочного нектара или поврежденных созревших плодов. Другими
хорошими источниками являются свежий яблочный сидр, вино и непастеризованное пиво, где эти бактерии активно растут на
поверхностной пленке. Типичные переносчики: плодовые мушки. Один из видов рода Acetobacter, Acetobacter xylinum обладает
способностью синтезировать целлюлозу, что характерно, в целом, только для растений.[1]
Необходимые файлы с генами были получены с помощью пакета EMBOSS.
Командой seqret embl:LN606601 plasmid.fasta был извлечен из базы данных файл plasmid.fasta для последующей работы в Prodigal.
Командой seqret embl:LN606601 -feature plasmid_1.gff был извлечен из базы данных файл plasmid_1.gff c сохраненными
особенностями для последующего извлечения координат белок-кодирующих последовательностей.
Работа с Prodigal
Для предсказания генов в выданной плазмиде использовалась программа Prodigal. Запуск программы из командной строки был осуществлен с помощью команды prodigal.windows.exe -i plasmid.fasta -o plasmid_an.pro -f sco.
Опции -i и -oзадают входной и выходной файлы соответсвенно, а опция -f выбирает нужный формат выходного файла. Был использован минималистичный формат sco для удобства дальнейшей работы.
Итоговый файл: plasmid_an.pro.
Обработка итоговых файлов
Для сравнения результатов предсказания генов выданной плазмиды необходимо было сопоставить информацию, содержащуюся в ранее полученных файлах файлах plasmid_an.pro и plasmid_1.gff.
Для возможности удобной обработки, файлы были преведены к единому, простому для анализа с помощью Python, формату.
Скрипт test_plasmid_pro.py перевел файл plasmid_an.pro в plasmid_an_final.txt, содержащий только координаты и ориентацию предсказанных
программой Prodiga генов.
Скрипт make_single_format_gff.py перевел файл plasmid_1.gff в plasmid_gff.txt, содержащий ту же информацию, аннотированную в Genbank.
После этого был написан скрипт analys_test_2.py, сравнивающий файлы plasmid_1.gff и plasmid_an.pro.
Общий алгоритм работы скрипта:
Скрипт открывает на чтение два файла, записывает в два массива все строки, прочитанные из полученных файлов, затем разбивает каждую строку из каждого массива в список. Далее, обращаясь к определенным элементам каждого
списка, мы можем сравнивать интересующие нас координаты, и, выставляя счетчики в виде переменных, фиксировать количество генов с определенным совпадением координат. После этого, мы выводим исходное количество генов,
значение каждого счетчика, а также процентное значение такого счетчика к общему количеству генов.
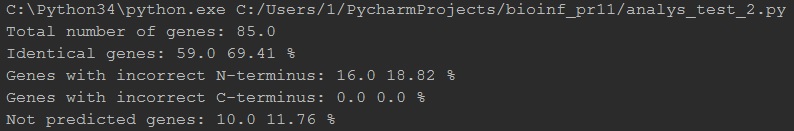 Рис.1 Результат работы скрипта
Рис.1 Результат работы скрипта
|
Количество генов |
Процентное соотношение |
| Полностью идентичные |
59 |
69,41% |
| Не совпадает N-конец |
16 |
18,82% |
| Не совпадает C-конец |
0 |
0% |
| Не совпадают оба конца |
10 |
11,76% |
| Итог: |
85 |
|
Таблица 1. Результаты работы Prodigal
Результаты
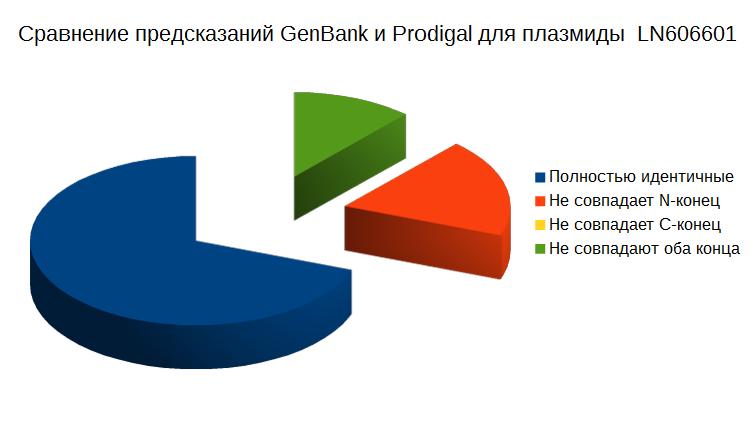 |
В целом можно заметить, что предсказание, выполненной Prodigal,
в данном случае не очень точное - полностью идентичными предсказано
только 70% генов.
Несовпадающих по C-концу генов не найдено, а вот по N-концу программа
определила целых 16 генов. Разберем некоторые такие примеры.
|
Был написан дополнительный скрипт analys_test_1.py, который отдельно находил и выдавал в output все пары генов из обоих файлов, которые имели одинаковые координаты по С-концам, но неодинаковые по N-концам.
Исходя из результата работы этого скрипта, я сразу анализировала пары генов с выданными им координатами.
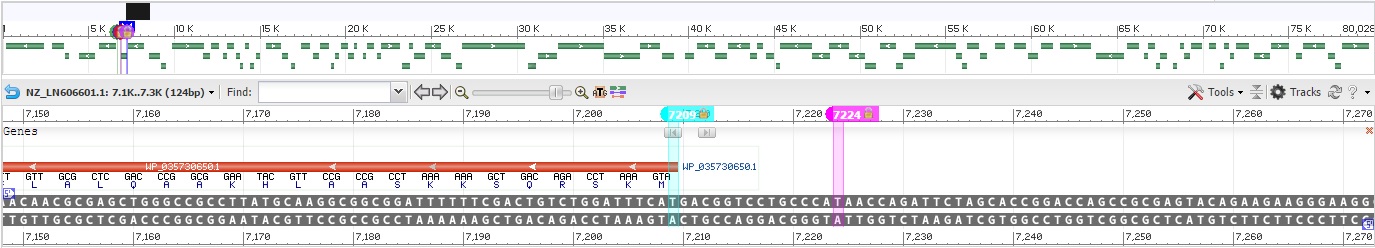
Рассмотрим ген с координатами 6904-7209 (-). Аннотацию с такими координатами мы получили из GenBank, но программа Prodigal иначе предсказала N-конец с координатой 7224.
Программа включила 15 лишних нуклеотидов. Обе границы N-конца, визуализированные с помощью геномного браузера, можно увидеть на рисунке ниже.
 | 
|
|
Продуктом данного гена является регуляторный белок, участвующий в метаболизме молибдена. Вероятнее всего, программа Prodigal
во время предсказания добавила лишние нуклеотиды к гену из-за дополнительного старт-кодона ATG, который стоит раньше истинного
старт-кодона этого гена. Стоит заметить, что в аннотации GenBank к этому гену указано, что при трансляции гена используется
таблица генетического кода 11, со старт-кодонами из которой было полезно свериться при изучении добавленного участка. Как
видно из таблицы, кодон ATG, из-за которого программа выдала неверно предсказанные координаты гена, входит в группу старт-кодонов
у изучаемых организмов.
На рисунке ниже желтым цветом отмечены реальные границы гена из GenBank, синим-добавленные программой лишние нуклеотиды (показана
прямая цепь, в то время, как ген читается по обратной). |  |
Следует упомянуть, что некоторые гены, правильно предсказанные программой Prodigal отбражались в геномном браузере, но их координат
не было обнаружено в файле, полученном из GenBank.
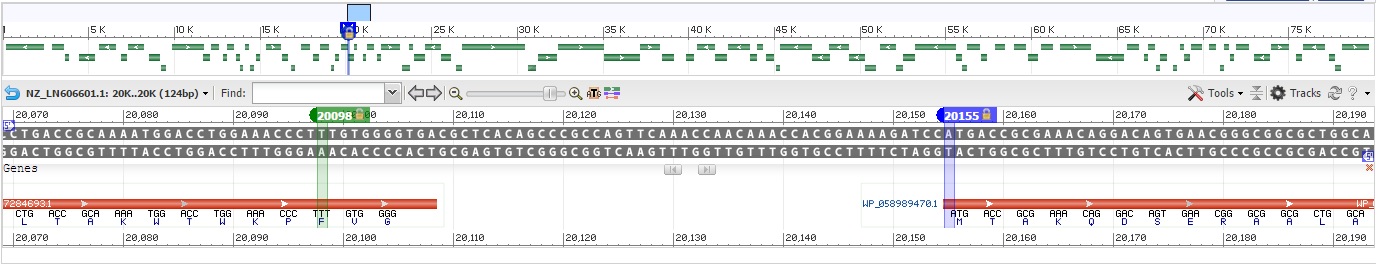
Рассмотрим ген из выданных нам скриптом analys_test_1.py с кординатами 20454-20098 (+). Аннотацию с такими координатами мы получили из GenBank, но программа Prodigal иначе предсказала N-конец с координатой 20155.
Программа исключила 57 лишних нуклеотидов, тем самым предсказав ген правильно. Обе границы N-конца, визуализированные с помощью геномного браузера, можно увидеть на рисунке ниже.
 | 
|
|
Продуктом данного гена является гипотетический белок без четко определенной функции. Стоит заметить, что при поиске записи, относящейся
к этому участку CDS, координаты изучаемого гена указаны в точности такие же, как и предсказанные программой Prodigal, а не такие, которые
указаны в файле, скачанном из GenBank с помощью EMBOSS. Вполне возможно, что здесь в базе данных ошибка.
Но, т.к. первым триплетом в гене с координатами, лежавшими в файле из GenBank(20454-20098 (+)) был старт-кодон TTG(сверяясь
с таблицей генетического кода 11 проверяем, что есть такой старт-кодон), то вполне можно предположить, что так был ошибочно предсказан ген и
эта запись осталась в базе.
На рисунке ниже синим цветом отмечены реальные границы гена из предсказания Prodigal, красным-добавленные в файле из GenBank(вероятно,
ошибочно) лишние нуклеотиды. |  |
Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для геномной записи
Источники:
[1] Acetic acid bacteria
[2] подсемейство Хомяки
[3] Кинетопласт Wiki
©
Avdiunina Polina, 2015


 Рис.1 Результат работы скрипта
Рис.1 Результат работы скрипта






