Для выполнения задания был выдан скэффолд NW_004457511 пренадлежащий организму Acanthamoeba castellanii - одному из представителей рода Acanthamoeba.
Представители этого таксона широко распространены: их можно обнаружить в почве, они часто встречаются в пресной воде
и других средах обитания. Как правило, амебы этого рода являются свободноживущими, но некоторые из них, например
Acanthamoeba keratitis и Acanthamoeba castellanii могут вызывать инфекции (Acanthamebiasis) у человека и животных.
Поскольку клетки представителей Acanthamoeba не сильно отличаются на ультраструктурном уровне от клетки млекопитающего,
они является хорошей моделью для клеточной биологии. В качестве модельного организма их используют в экологии,
физиологии, моделированиии клеточных взаимодействий, молекулярной биологии, биохимии и эволюционных исследованиях.
Благодаря простоте и экономичности культивирования, штамм Neff А. castellanii был эффективно использован в качестве
классического модельного организма.[1]
Электронную микрофотографию и таксономию Acanthamoeba castellanii можно увидеть на рис. 1
Была получена последовательность скэффолда в формате fasta: seq_scaff.fasta.
Выданный скэффолд имеет размер 123495 bp.
Информации о количестве генов и белок-кодирующих последовательностях в аннотации представлено не было. Для подсчета
количества генов и белок-кодирующих последовательностей(CDS) из базы данных Genbank был получен файл seq_scaff_CDS.gb,
который был предварительно обработан из командной строки с помощью EMBOSS:командой extractfeat seq_scaff_CDS.gb -type
CDS -join seq_test_CDS.fasta был создан новый файл seq_test_CDS.fasta, где лежат только объединенные по указанным в
аннотации координатам белок-кодирующие участки(CDS), число которых было подсчитано с помощью скрипта count_CDS.py.
Результатом программы служит число аннотированных белок-кодирующих участков в записи: данный скэффолд содержит 40
таких участков.
Командой extractfeat seq_scaff_CDS.gb -type gene seq_test_gene.fasta был создан новый файл seq_test_gene.fasta,
где лежат только последовательности аннотированных в записи генов, число которых было подсчитано с помощью скрипта
count_gene.py.
Результатом программы служит число аннотированных генов: данный скэффолд содержит 41 ген.
Ниже в таблице 1 приведена краткая информация о скэффолде NW_004457511.
По данным RefSeq не удалось обнаружить ни одного гена во всей последовательности, для которого предсказывается альтернативный сплайсинг.
Стрелки указывают положение гена цепи, в нашем случае ген ACA1_151240 находится на прямой цепи. Фиолетовым цветом показаны
последовательности мРНК, образующиеся после транскрипции. Толстые красные линии указывают на положение белок-кодирующих
последовательностей (CDS).
Продуктом данного гена является белок XP_004340867.1. Этот белок принадлежит к семейству цистеиновых протеиназ, участвующих в процессе
мечения и деградации белков эукариот с помощью убиквитина. Белки данного семейства удаляют убиквитиновые метки с белков, которые ранее
предполагалось расщепить. 3D - структуру белка OTUB2, одного из представителей данного семейства, можно увидеть на рисунке ниже. |
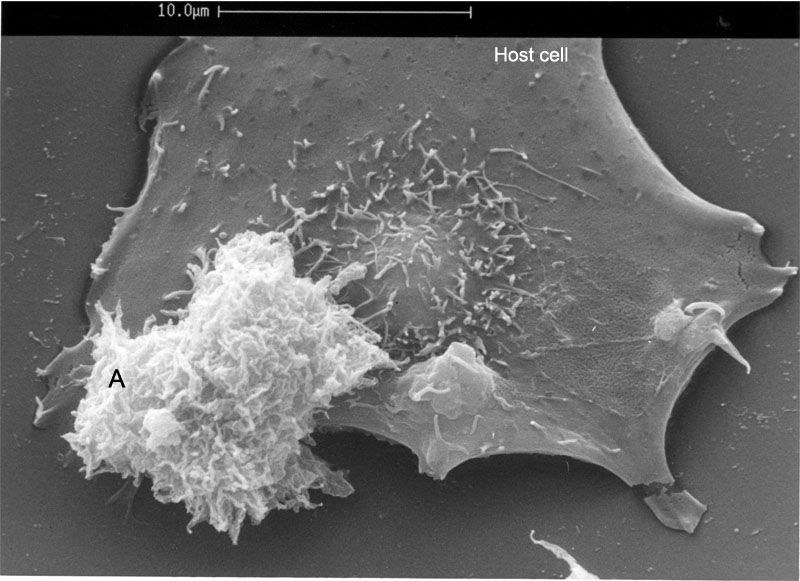 |
| Acanthamoeba castellanii (A) |
Superkingdom: Eukaryota
(unranked): Amoebozoa
(unranked): Discosea
Order: Longamoebia
Genus: Acanthamoeba
Species: A. castellanii
|
|
|
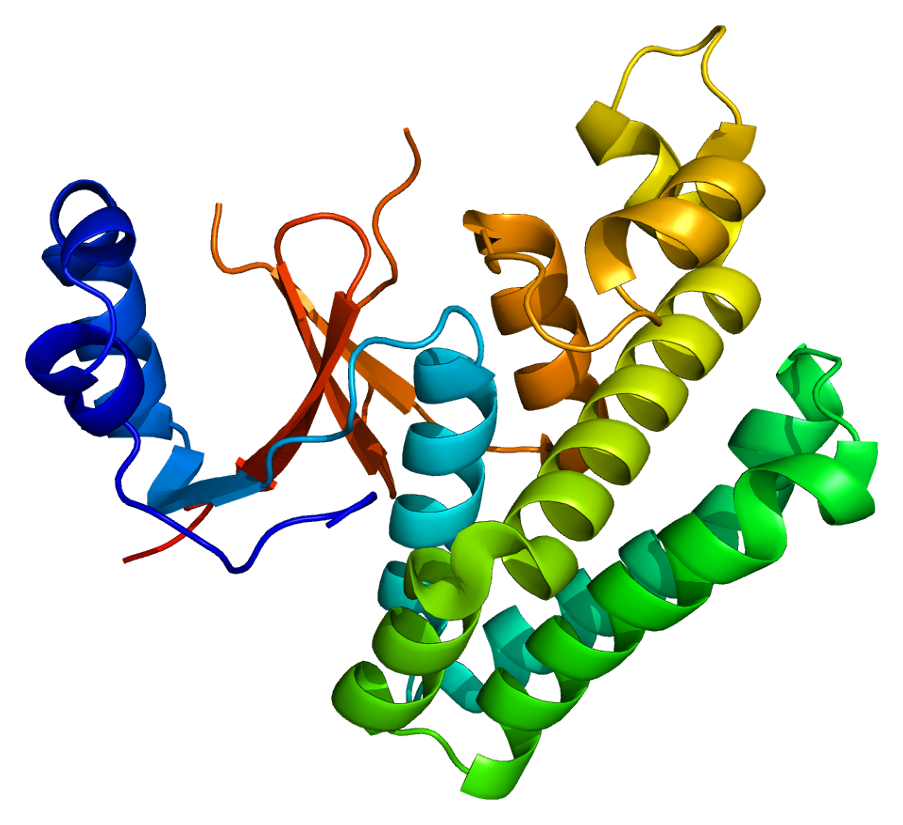 Таблица 1 OTUB2
Таблица 1 OTUB2
При поиске вручную генов, для которых в аннотации предсказан альтернативный сплайсинг, ничего не было найдено. Для анализа был взят средний
по длине ген ACA1_151240 с довольно длинным интроном и 8 экзонами. У белка этого гена нет изоформ(как и у всех белков из данного скэффолда),
как можно увидеть из рисунка, взятого из геномного браузера.
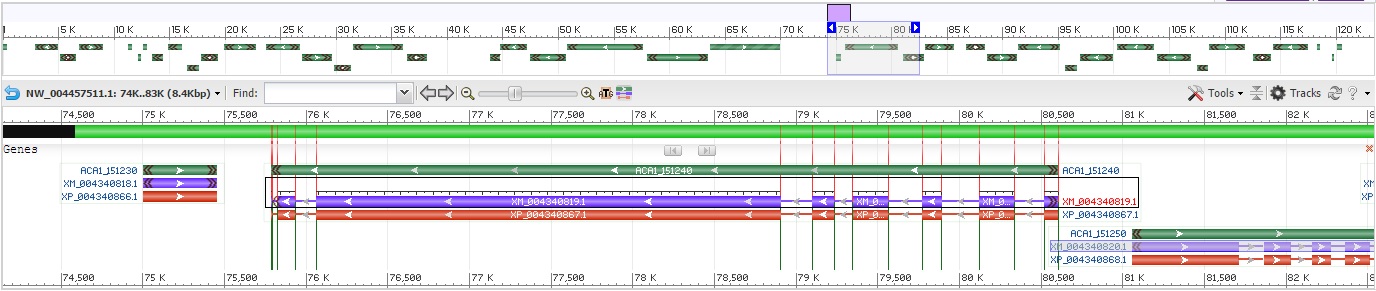 Риc. 1 Геномное окружение гена ACA1_151240
Риc. 1 Геномное окружение гена ACA1_151240
Предсказание AUGUSTUS
Для выполнения этой части задания использовался web-сервера AUGUSTUS[2]. В качестве организма, из генома которого будут браться параметры модели, был выбран представитель рода Toxoplasma - Toxoplasma gondii.
Далее для выданного скэффолда были предсказаны гены и белок-кодирующие области с помощью сервера AUGUSTUS в режиме prediction.
Параметры запуска можно увидеть на изображении ниже.
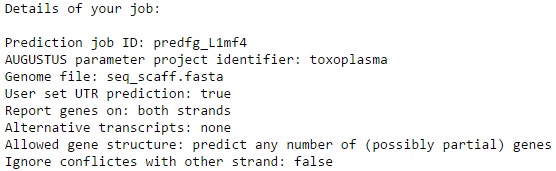 Риc. 2 Параметры запуска
Предсказание UTR аннонсирует нетранслируемые участки, а также делает предсказание кодирующих участков чуть более точным. Эта опция доступна не для всех организмов, однако для Toxoplasma gondii такая опция
предусмотрена, поэтому поле predict UTRs было отмечено, несмотря на увеличение времени работы сервера.
По данным, полученным в первой части задания, ни для одного гена в выданном скэффолде не был предсказан альтернативный сплайсинг, из-за чего в поле alternative transcripts было оставленно отмеченным поле
none.
Остальные параметры были взяты по умолчанию (предсказывать гены на обеих цепях, предсказывать любое число полных/частичных генов, не игнорировать конфликты с комплементарной цепью).
В результате работы сервера был получен архив predictions.tar.gz, распакованный с помощью команды tar -xzvf predictions.tar.gz. Архив содержал 6 файлов, комментарии к которым можно найти в таблице ниже.
Таблица 2
Риc. 2 Параметры запуска
Предсказание UTR аннонсирует нетранслируемые участки, а также делает предсказание кодирующих участков чуть более точным. Эта опция доступна не для всех организмов, однако для Toxoplasma gondii такая опция
предусмотрена, поэтому поле predict UTRs было отмечено, несмотря на увеличение времени работы сервера.
По данным, полученным в первой части задания, ни для одного гена в выданном скэффолде не был предсказан альтернативный сплайсинг, из-за чего в поле alternative transcripts было оставленно отмеченным поле
none.
Остальные параметры были взяты по умолчанию (предсказывать гены на обеих цепях, предсказывать любое число полных/частичных генов, не игнорировать конфликты с комплементарной цепью).
В результате работы сервера был получен архив predictions.tar.gz, распакованный с помощью команды tar -xzvf predictions.tar.gz. Архив содержал 6 файлов, комментарии к которым можно найти в таблице ниже.
Таблица 2
| Расширение файла |
Информация |
| *.gff |
предсказания генов в gff-формате |
| *.gtf |
предсказания генов в gtf-формате |
| *.aa |
белковые последовательности предсказанных генов в fasta-формате |
| *.codingseq |
предсказание в виде fasta-последовательности кодирующих участков ДНК (CDS) |
| *.cdsexons |
предсказание в виде fasta-последовательности экзонов |
| *.mrna |
предсказанные последовательности мРНК (с UTRs) в fasta-формате |
| *.gbrowse |
трек-файл для GBrowse |
Ссылка на файл с предсказаниями генов: augustus.gff.
Оценка предсказаний
AUGUSTUS предсказал всего 23 гена, в то время как в файле GenBank аннотирован 41 ген.
При помощи программ Python, аналогичным тем, что использовались в предыдущем практикуме, было проведено сравнение предсказаний AUGUSTUS с данными, лежащими в GenBank. Программа augustus_test.py извлекла из файла augustus.gff
координаты предсказанных генов и поместила в файл augustus_final.txt.
Со страницы скэффолда был скачан файл sequence.gb, а затем с помощью команды featcopy sequence.gb coor_genbank.gff пакета EMBOSS был получен фай coor_genbank.gff, содержащий таблицу аннотаций особенностей. Далее, с помощью
программы genbank_single_format.py координаты генов из этого файла были записаны в файл genbank_final.txt. После этого было проведено сравнение двух файлов с координатами с помощью программы analys_test_2.py.
Результат работы программы можно увидеть ниже.
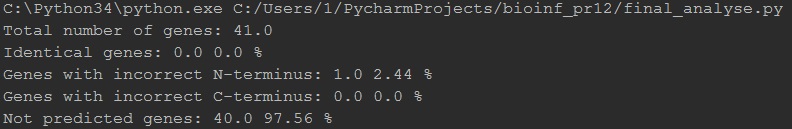 Риc. 3 Результат работы программы
Как можно видеть, предсказание AUGUSTUS получилось совсем некачественным. Ни одного гена не было предсказано полностью правильно, 1 ген был верно предсказан по N-концу и ни одного верного по C-концу. Также было предсказано
всего 23 гена из 41. Очевидно, что данный результат совсем не удовлетворительный.
Вероятно причиной плохого предсказания могло послужить то, что Acanthamoeba castellanii и Toxoplasma gondii недостаточно близки и проводить предсказание для нее на основании другого организма не совсем корректно.
Так как ни одного гена не было предсказано правильно, то очевидно, что и для исследуемого в задании 1 гена ACA1_151240 предсказание оказалось неверным, из-за этого сравнение проводить не с чем. Среди предсказанных
наиболее "близким" по координатам с нашим оказался ген с координатами complement(75571-80790), однако это вообще нельзя считать предсказанием, так как ошибка слишком велика. "Близкий" ген длиннее на 401 bp, и гены
окружения тоже предсказаны неверно.
Риc. 3 Результат работы программы
Как можно видеть, предсказание AUGUSTUS получилось совсем некачественным. Ни одного гена не было предсказано полностью правильно, 1 ген был верно предсказан по N-концу и ни одного верного по C-концу. Также было предсказано
всего 23 гена из 41. Очевидно, что данный результат совсем не удовлетворительный.
Вероятно причиной плохого предсказания могло послужить то, что Acanthamoeba castellanii и Toxoplasma gondii недостаточно близки и проводить предсказание для нее на основании другого организма не совсем корректно.
Так как ни одного гена не было предсказано правильно, то очевидно, что и для исследуемого в задании 1 гена ACA1_151240 предсказание оказалось неверным, из-за этого сравнение проводить не с чем. Среди предсказанных
наиболее "близким" по координатам с нашим оказался ген с координатами complement(75571-80790), однако это вообще нельзя считать предсказанием, так как ошибка слишком велика. "Близкий" ген длиннее на 401 bp, и гены
окружения тоже предсказаны неверно.
Источники:
[1] Acanthamoeba castellanii
[2] AUGUSTUS
[3] PDB
©
Avdiunina Polina, 2015

 Таблица 1 OTUB2
Таблица 1 OTUB2
 Риc. 1 Геномное окружение гена ACA1_151240
Риc. 1 Геномное окружение гена ACA1_151240
 Риc. 2 Параметры запуска
Предсказание UTR аннонсирует нетранслируемые участки, а также делает предсказание кодирующих участков чуть более точным. Эта опция доступна не для всех организмов, однако для Toxoplasma gondii такая опция
предусмотрена, поэтому поле predict UTRs было отмечено, несмотря на увеличение времени работы сервера.
По данным, полученным в первой части задания, ни для одного гена в выданном скэффолде не был предсказан альтернативный сплайсинг, из-за чего в поле alternative transcripts было оставленно отмеченным поле
none.
Остальные параметры были взяты по умолчанию (предсказывать гены на обеих цепях, предсказывать любое число полных/частичных генов, не игнорировать конфликты с комплементарной цепью).
В результате работы сервера был получен архив predictions.tar.gz, распакованный с помощью команды tar -xzvf predictions.tar.gz. Архив содержал 6 файлов, комментарии к которым можно найти в таблице ниже.
Таблица 2
Риc. 2 Параметры запуска
Предсказание UTR аннонсирует нетранслируемые участки, а также делает предсказание кодирующих участков чуть более точным. Эта опция доступна не для всех организмов, однако для Toxoplasma gondii такая опция
предусмотрена, поэтому поле predict UTRs было отмечено, несмотря на увеличение времени работы сервера.
По данным, полученным в первой части задания, ни для одного гена в выданном скэффолде не был предсказан альтернативный сплайсинг, из-за чего в поле alternative transcripts было оставленно отмеченным поле
none.
Остальные параметры были взяты по умолчанию (предсказывать гены на обеих цепях, предсказывать любое число полных/частичных генов, не игнорировать конфликты с комплементарной цепью).
В результате работы сервера был получен архив predictions.tar.gz, распакованный с помощью команды tar -xzvf predictions.tar.gz. Архив содержал 6 файлов, комментарии к которым можно найти в таблице ниже.
Таблица 2
 Риc. 3 Результат работы программы
Как можно видеть, предсказание AUGUSTUS получилось совсем некачественным. Ни одного гена не было предсказано полностью правильно, 1 ген был верно предсказан по N-концу и ни одного верного по C-концу. Также было предсказано
всего 23 гена из 41. Очевидно, что данный результат совсем не удовлетворительный.
Вероятно причиной плохого предсказания могло послужить то, что Acanthamoeba castellanii и Toxoplasma gondii недостаточно близки и проводить предсказание для нее на основании другого организма не совсем корректно.
Так как ни одного гена не было предсказано правильно, то очевидно, что и для исследуемого в задании 1 гена ACA1_151240 предсказание оказалось неверным, из-за этого сравнение проводить не с чем. Среди предсказанных
наиболее "близким" по координатам с нашим оказался ген с координатами complement(75571-80790), однако это вообще нельзя считать предсказанием, так как ошибка слишком велика. "Близкий" ген длиннее на 401 bp, и гены
окружения тоже предсказаны неверно.
Риc. 3 Результат работы программы
Как можно видеть, предсказание AUGUSTUS получилось совсем некачественным. Ни одного гена не было предсказано полностью правильно, 1 ген был верно предсказан по N-концу и ни одного верного по C-концу. Также было предсказано
всего 23 гена из 41. Очевидно, что данный результат совсем не удовлетворительный.
Вероятно причиной плохого предсказания могло послужить то, что Acanthamoeba castellanii и Toxoplasma gondii недостаточно близки и проводить предсказание для нее на основании другого организма не совсем корректно.
Так как ни одного гена не было предсказано правильно, то очевидно, что и для исследуемого в задании 1 гена ACA1_151240 предсказание оказалось неверным, из-за этого сравнение проводить не с чем. Среди предсказанных
наиболее "близким" по координатам с нашим оказался ген с координатами complement(75571-80790), однако это вообще нельзя считать предсказанием, так как ошибка слишком велика. "Близкий" ген длиннее на 401 bp, и гены
окружения тоже предсказаны неверно.