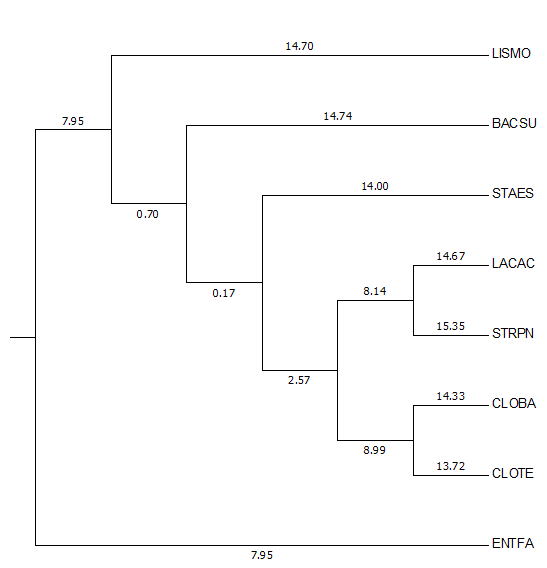
Дерево, построенное по выравниванию белков факторов элонгации трансляции Ts (мнемоника EFTS) алгоритмом
Neighbour-Joining % Ident (рисунок 1), было переукоренено в среднюю точку с
помощью пакета PHYLIP.
Полученное дерево представлено на рисунке 2.

Рис.1 Исходное дерево, построенное алгоритмом Neighbour-Joining % Ident, реализованным в JalView.
Изображение получено с помощью программы MEGA (дерево в Newick-формате).
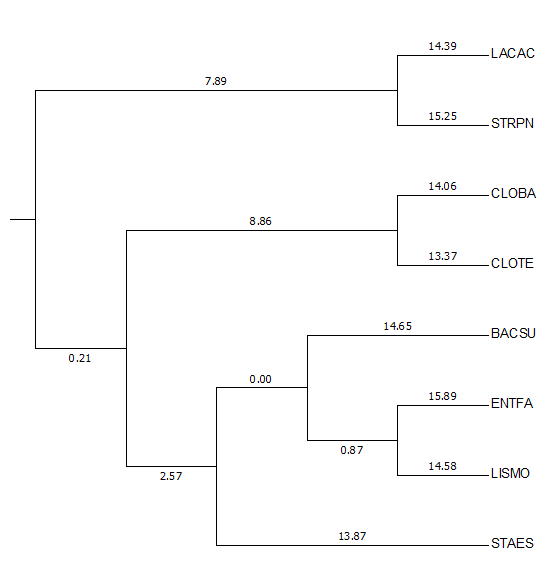
Рис.2 Укорененное дерево, построенное методом укоренения в среднюю точку.
Изображение получено с помощью программы MEGA (дерево в Newick-формате).
Как видно, в данном случае укоренение получилось не совсем правильное: дерево видов укоренено в ветвь Clostridium(CLOB1, CLOTE),
а на рис.2 произошло укоренение
в ветвь Lactobacillales(только STRPN и LACAC). Кроме того, ветвь Clostridium - единственная общая ветвь
для дерева видов и дерева, укорененного в среднюю точку.
В полученном дереве последовательность ENTFA находится внутри клады порядка
Bacillales(BACSU, LISMO, STAES), на дереве видов же это представитель порядка
Lactobacillales(ENTFA, STRPN, LACAC).
Из деревье практикума 2(доп. задание) построенное дерево по топологии больше всего похоже на результат работы алгоритма
Neighbour-Joining Using Pam250(рисунок 3).
Изменяется только распаложение бактерий порядка Bacillales(BACSU, LISMO, STAES) и последовательности
ENTFA: на построенном дереве отдельную кладу образуют ENTFA и
LISMO, в случае Pam250 отдельную кладу составили BACSU и LISMO.
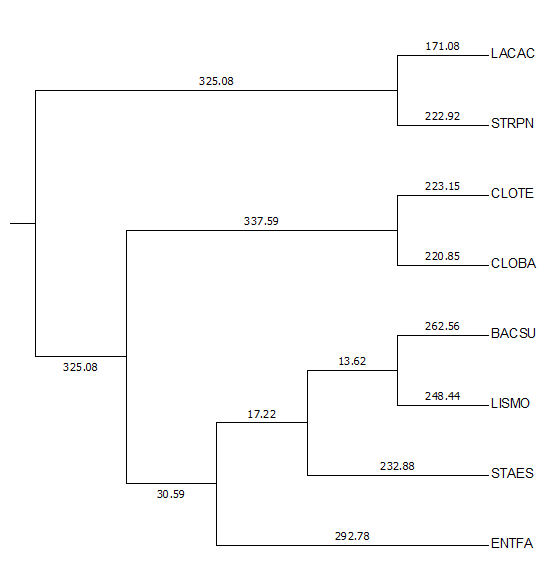
Рис.3 Филогенетическое дерево, построенное с использованием алгоритма Neighbour-Joining Using Pam250.
Изображение получено с помощью программы MEGA.
© Avdiunina Polina, 2017