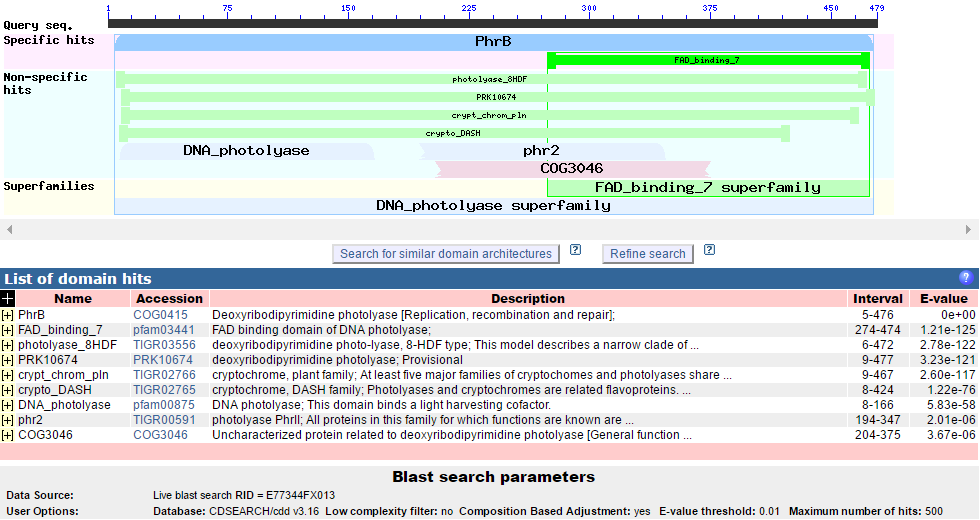
Рис.1 Список хитов для белка NP_354235.1.
При выполнении практикума использовался белок NP_354235.1 из бактерии Agrobacterium fabrum str. C58. Белок состоит из 479 аминокислотный остатков
и является ДНК-фотолиазой-одним из ферментов
репарации ДНК. ДНК-фотолиаза удаляет фотопродукты и пиримидин-пиримидиновые димеры, образующиеся
в молекуле ДНК под действием коротковолнового УФ-излучения. Процесс, в котором участвует фермент,
называется фотореактивацией. Такие фотореактивирующие
ферменты имеются у бактерий и низших эукариотических организмов, но в клетках млекопитающих они не обнаружены.
С помощью сервиса CDD (Conserved Domain Database) для данного белка был получен список хитов (рис. 1), из которого были выбраны те, которые относят белок к тому или иному КОГу.

Рис.1 Список хитов для белка NP_354235.1.
Таких хитов, соответственно и КОГов, нашлось всего 2. Информация по каждому из КОГов в соответствии с последним релизом базы данных представлена в Таблице 1.
| ID | Координаты | E-value | Название(annotation) | Функциональная категория(cat) |
|---|---|---|---|---|
| COG0415 | 5-476 | 0e+00 | Deoxyribodipyrimidine photolyase (Дезоксирибопиридин фотолиаза) |
L - Replication, recombination and repair (репликация, рекомбинация, репарация) |
| COG3046 | 204-375 | 3.67e-06 | Uncharacterized protein related to deoxyribodipyrimidine photolyase (Белок без аннотации, ассоциирован с дезоксирибопиридин фотолиазой) |
R - General function prediction only (только общее предсказание функции) |
Таблица 1. КОГи для белка NP_354235.1
Необходимо получить изображение геномного окружения КОГа c помощью сервисов STRING и/или COGNAT и прокомментировать полученные результаты.
Из двух обнаруженных КОГов я выбрала один с наилучшим E-value, а именно COG0415.
Было получено изображение с помощью STRING (рис. 2). Ограничения по организму выставлено не было, т.к. в базе информации по моему организму найдено не было.
Параметры программы были взяты по умолчанию: максимальное количество взаимодействующих по первой линии белков (first shell interactors) - 10, минимальный требуемый
score взаимодействия - 0.400.
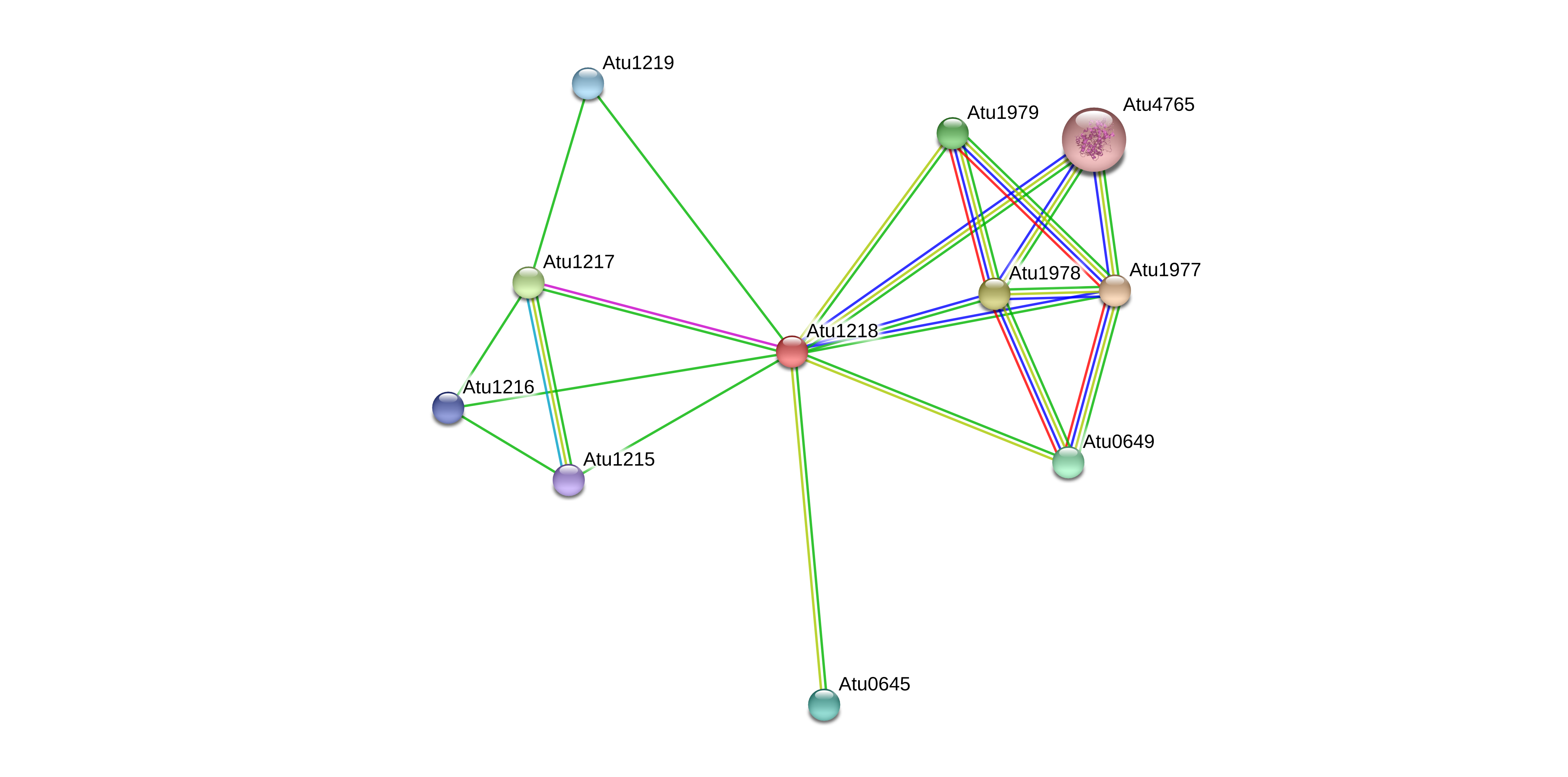
Рис.4 Взаимосвязи белка NP_354235.1(Atu1218)
Пояснения по рисунку можно найти в легенде(рис. 5), а также в разделе Help сервиса STRING, где изложена подробая документация.
На данном рисунке каждый узел графа символизирует белок(совокупность всех его изоформ и посттрансляционных модификаций, транскрибирующихся с одного и того же локуса гена).
Ребра графа
символизируют белок-белковые ассоциации (это не обязательно означает, что белки механически взаимодействуют друг с другом; они могут быть связаны и функционально).
Белки, для которых
известна 3D-структура отображены большимим узлами с соответсвующим изображением внутри, остальные белки отображены узлами меньшего размера.
Красным цветом выделен исходный анализируемый
белок.
Цвет ребер также несет смысловую значимость. Ребра, окрашенные малиновым, отражают экспериментально доказанные взаимосвязи, голубым - взаимосвязи, информация о которых
получена из курируемых
баз данных. Зеленые ребра отражают соседство в геноме, синие - совместную встречаемость, красные - слияние генов. Салатовые ребра означают совместное
упоминание данных белков в Pub-Med
Abstract, черные - коэкспрессию, а светло-фиолетовые - гомологию. Полный список интерпретаций цветовых обозначений можно посмотреть здесь.
На нашем графе наибольшее число ребер окрашены в зеленый, светло-голубой, салатовый или синий (взаимосвязи, которым нельзя верить на 100%). Однако есть и достаточное количество (5)
достоверных взаимосвязей, установленных экспериментально.
Ниже на рисунке 5 можно увидеть таблицу, отражающую все взаимосвязи нашего белка с остальными белками на графе,
а также score данных взаимосвязей.

Рис.5 Легенда
В выданной группе белков наблюдается консервативное геномное окружение. Наблюдаются очевидные повторяющиеся паттерны расположения генов в разных группах организмов (рис. 6).
Наиболее
распространенным является паттерн Atu0645 и исследуемого нами белка Atu1218(на картинке белки обозначены красным и темно-зеленым, блоки их паттернов выделены салатовым
цветом). Также
довольно часто встречается длинный паттерн из 5-ти белков: Atu0645 — Atu1218 — Atu0649 — Atu1978 — Atu1977(на рис.6 блоки выделены синим цветом).
В некоторых случаях наблюдается вариабельность, выражающаяся в виде перестановки белков внутри паттернов или изменения направления. Дополнительные вставки в рамках одного
паттерна не
характерны. Важно заметить, что здесь и в таблице используются идентификаторы генов белков.
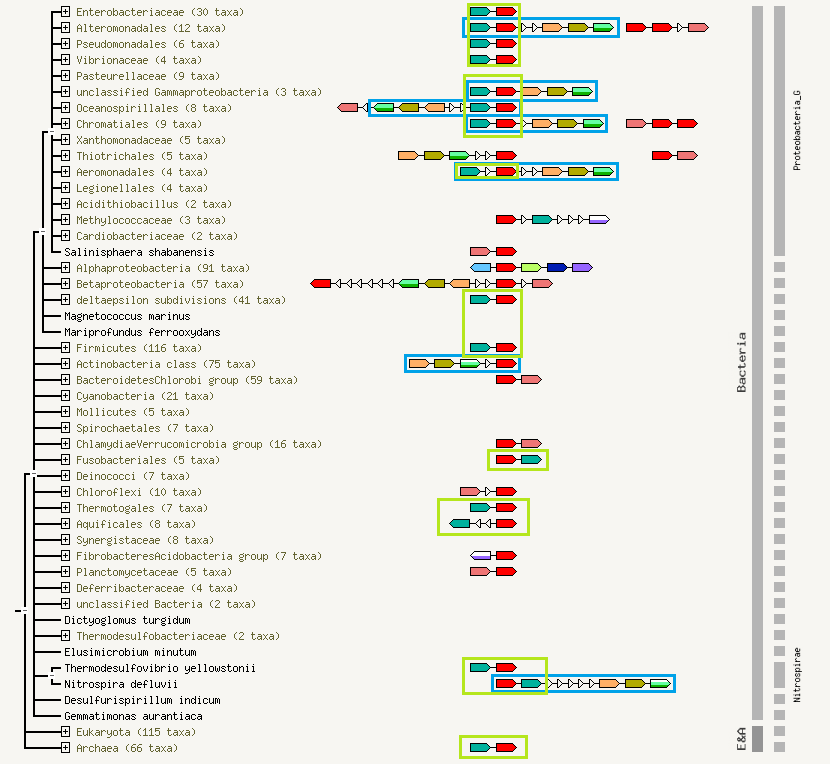
Рис. 6 Геномное окружение COG0415, программа STRING
Описания белков паттернов представлены в таблице 2. Как видно из описаний, статус Reviewed имеет только один белок, Annotation score у всех белков, кроме этого, также был очень
низкий.
Делать выводы о функциональном сходстве в данном случае не очень корректно на мой взгляд.
| Название белка | Функция |
|---|---|
| Atu1977 | Аминооксидаза Status: Unreviewed |
| Atu1978 | Функция неизвестна Status: Unreviewed |
| Atu1218 | Дезоксирибодипиримидин фотолиаза, участвует в репарации Status: Reviewed |
| Atu0645 | Функция неизвестна Status: Unreviewed |
| Atu0649 | Циклопропанацил фосфолипидсинтаза Status: Unreviewed |
Таблица 2. Описания белков, входящих в консервативный паттерн
С помощью инструмента AmiGO поиком BLAST в базе данных GO был обнаружен белок, наиболее похожий на NP_354235.1. (UniProtKB - A9CJC9 (PHRA_AGRFC)). Им оказался белок
Q83CE4 (Q83CE4_COXBU) -
предполагаемая дезоксирибодипиримидин фотолиаза из организма Coxiella burnetii (strain RSA 493). Изначально выданный мне белок выполняет ту же функцию,
но принадлежит другому
организму - Agrobacterium fabrum str. C58.

Рис.7 Выравнивание белков PHRA_AGRFC и Q83CE4_COXBU
На рисунке 7 можно увидеть выравнивание нашего белка и лучшего белка-находки. Длина выравнивания - 472 (длина нашего белка - 479, белка-находки - 472). Probability составляет 5.9e-112.
Очевидно, что найденный в БД GO белок не является тем же самым, что и исходный. Тем не менее степень сходства у этих белков хоть и не высокая, но достаточная, о чем можно судить
по неплохому
Probability и удовлетворительному проценту совпадений.
На странице белка Q83CE4 (Q83CE4_COXBU) по ссылке N term
associations была получена информация о терминах GO, отнесенных к данному белку. Их оказалось всего 2. Найденные термины представлены
в таблице 3. Объяснения встречающихся в таблице
3 кодов достоверности представленs в таблице 4.
| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
|---|---|---|---|---|
| Биологический процесс (Biological process) |
GO:0006281 | DNA repair | Репарация ДНК | ISS |
| Молекулярная функция (Molecular function) |
GO:0003904 | Ddeoxyribodipyrimidine photo-lyase activity |
Дезоксипиримидинфотолиазная активность |
ISS |
Таблица 3. Термины GO, отнесенные к белку с идентификатором Uniprot Q83CE4 (Q83CE4_COXBU)
| Код типа достоверности |
Расшифровка кода |
Пояснение |
|---|---|---|
| ISS | Inferred from Sequence or Structural Similarity (основан на сходстве последовательностей или структурном сходстве) |
Используется, когда аннотация проводилась на основании анализа последовательностей, причем данный анализ был проверен вручную. В случае, когда имел место только автоматический анализ, корректным является использование кода IEA. Общий код ISS используется при применение комбинации анализирующих последовательности инструментов и методов. Если был применен только один метод/ инструмент используют одну из подкатегорий ISS: ISA (Inferred from Sequence Alignment) - заключение на основании парного или множественного выравнивания, ISO (Inferred from Sequence Orthology) - заключение на основании оценки ортологичности продуктов генов из разных организмов, ISM (Inferred from Sequence Model) - заключение на основании какого-либо метода моделирования (например Hidden Markov Models).ISS также можно использовать при наличии структурного сходства с экспериментально описанными продуктами генов, установленного с помощью кристаллографии, ЯМР или вычислительных предсказаний. Однако на практике код ISS крайне редко применяют для аннотации, основанной только на информации о структуре. |
Таблица 4. Описание кодов достоверности
Источники:© Avdiunina Polina, 2017