
Рис.1 Выравнивание выбранного подсемейства белков
Сначала необходимо было выбрать подсемейство из выравнивания последовательностей домена, исследованного в прошлом практикуме.
Подсемейство будем считать хорошим, если оно является кладой на дереве и если есть некоторые признаки (доменная архитектура,
таксономическая принадлежность или диагностические позиции), по которым можно отнести домен к данному подсемейству. Я решила
выбрать
подсемейство, выделенное в выравнивании на рис. 1. Они образуют кладу и имеют диагностические позиции (например, №23 и 48).
Таким образом,
в подсемействе 16 последовательностей. Выбранные последовательности
были сохранены в отдельный файл в формате .fasta.

Рис.1 Выравнивание выбранного подсемейства белков
Далее для полученного подсемейства необходимо было построить профиль. Для этого использовался пакет HMMER, использующий скрытые модели Маркова
для анализа выравнивания последовательностей и
поиска гомологов.
Сначала был построен профиль:
Команда: hmm2build profile.out sec_pr_selected.fasta
Полученный файл c профилем был откалиброван:
Команда: hmm2calibrate profile.out
Затем я скачала с сайта UniProt полноразмерные последовательности белков, содержащих мой домен.
По полученному файлу для итогового профиля был осуществлен поиск гомологов:
Команда: hmm2search profile.out PF14535.fasta > findings.out
В итоговом файле findings.out содержатся все находки.
Для анализа результатов нам предложили использовать ROC-кривую и гистограмму весов находок.
ROC-кривая (Receiver Operator Characteristic) — кривая, которая наиболее часто используется для анализа качества моделей.
ROC-кривая показывает зависимость количества верно
классифицированных положительных примеров от количества неверно классифицированных
отрицательных примеров. Рассмотрим возможные результаты:
TP (True Positives) — верно классифицированные положительные примеры (истинно положительные случаи).
В нашем случае — это количество последовательностей,
расположенных выше некоторого порога и достоверно содержащих искомый домен;
TN (True Negatives) — верно классифицированные отрицательные примеры (истинно отрицательные случаи).
В нашем случае — это количество последовательностей, расположенных ниже
некоторого порога и достоверно не содержащих искомый домен;
FP (False Positives) — отрицательные примеры, классифицированные как положительные (ошибка II рода);
Это ложное обнаружение, т.к. при отсутствии события ошибочно выносится
решение о его присутствии (ложно положительные случаи).
В нашем случае — это количество последовательностей, расположенных выше некоторого порога, но не содержащих домен;
FN (False Negatives) — положительные примеры, классифицированные как отрицательные (ошибка I рода).
Это так называемый "ложный пропуск" – когда интересующее нас событие
ошибочно не обнаруживается (ложно отрицательные примеры).
В нашем случае — это количество последовательностей, расположенных ниже некоторого порога и содержащих домен.
Специфичность (SP) — доля истинно отрицательных случаев, которые
были правильно идентифицированы моделью (т.е. доля достоверно предсказанных белков, не содержащих домен,
2 от общего количества последовательностей, известно не содержащих этот домен).
Чувствительность (SE) — доля истинно положительных случаев (т.е. доля достоверно предсказанных белков, содержащих домен, от общего количества последовательностей,
известно содержащих домен.
Из определений следуют формулы, по которым считаются данные показатели:
SP = TN/(TN+FP)
SE = TP/(TP+FN)
Были проанализированы результаты поиска гомологов по профилю.
Файл с находками был открыт в Excel (лист 'profile'), на другом листе ('roc_line') для
каждого значения порога были подсчитаны TP, TN, FP и FN, а также SP и SN,
а также значения 1-SP, построена ROC-кривая: по оси ОY
откладывается SЕ, по оси ОX — 1–SP (сто процентов минус специфичность).
Результат приведен на рис. 2.
Итоговая таблица: table_1_1.ods.
E-value: 1.1e-46, Score: 163.7
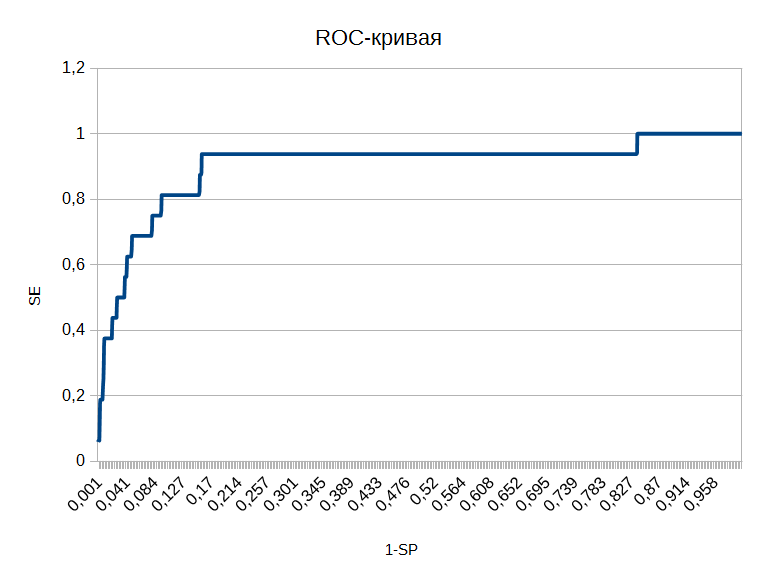
Рис.2 Полученная ROC-кривая
| Находок | принадлежит подсемейству | не принадлежит подсемейству | Сумма |
|---|---|---|---|
| Выше порога по профилю | 15 | 323 | 338 |
| Ниже порога | 0 | 1733 | 1733 |
| Сумма | 15 | 2056 | 2071 |
Источники:
© Avdiunina Polina, 2017